20 Valores faltantes
En esta página se explica cómo:
- Evaluar la falta de información
- Filtrar las filas por valores faltantes
- Representar la falta de datos a lo largo del tiempo
- Manejar cómo se muestra
NAen los gráficos - Realizar la imputación de valores faltantes: MCAR, MAR, MNAR
20.1 Preparación
Cargar paquetes
Este trozo de código muestra la carga de los paquetes necesarios para los análisis. En este manual destacamos p_load() de pacman, que instala el paquete si es necesario y lo carga para su uso. También puedes cargar los paquetes instalados con library() de de R base Consulta la página sobre fundamentos de R para obtener más información sobre los paquetes de R.
pacman::p_load(
rio, # importación/exportación
tidyverse, # gestión y visualización de datos
naniar, # evaluación y visualización de datos faltantes
mice # imputación de datos faltantes
)##hastaqui
Importar datos
Importamos los datos de casos de una epidemia de ébola simulada. Si quieres seguir el proceso, clica para descargar linelist “limpioa” (como archivo .rds). Importa tus datos con la función import() del paquete rio (acepta muchos tipos de archivos como .xlsx, .rds, .csv - Mira la página de importación y exportación para más detalles).
# importar linelist
linelist <- import("linelist_cleaned.rds")A continuación se muestran las primeras 50 filas de linelist.
Convertir valores faltantes en la importación
Al importar los datos, ten en cuenta los valores que deben clasificarse como faltantes. Por ejemplo, 99, 999, “Missing”, celdas en blanco (““) o celdas con un espacio vacío (” “). Puedes convertirlos en NA (la versión de R de los valores faltantes) con el comando de importación de datos. Consulta la sección de datos faltantes de la página de Importación para obtener más detalles, ya que la sintaxis exacta varía según el tipo de archivo.
20.2 Valores faltantes en R
A continuación, exploramos las formas en que se presenta y evalúa los datos faltantes en R, junto con algunos valores y funciones adyacentes.
NA
En R, los valores faltantes se representan con un valor reservado (especial): NA. Ten en cuenta que se escribe sin comillas. “NA” es diferente y es sólo un valor de carácter normal (también una letra de los Beatles de la canción Hey Jude).
Tus datos pueden tener otras formas de representar la falta de información, como “99”, o “Missing”, o “Desconocido” - incluso puedes tener el valor de carácter vacío “” que parece “en blanco”, o un solo espacio ” “. Se consciente de ello y considera la posibilidad de convertirlos en NA durante la importación o durante la limpieza de datos con na_if().
En tu limpieza de datos, también puedes convertir en el otro sentido - cambiando todos los NA a “Missing” o similar con replace_na() o con fct_explicit_na() para los factores.
Versiones de NA
La mayoría de las veces, NA representa un valor que falta y todo funciona bien. Sin embargo, en algunas circunstancias puedes encontrar la necesidad de variaciones de NA específicas para un tipo de objeto (carácter, numérico, etc.). Esto será poco frecuente, pero debes tenerlo en cuenta.
El escenario típico para esto es cuando se crea una nueva columna con la función dplyr case_when(). Como se describe en la página de Limpieza de datos y funciones básicas, esta función evalúa cada fila del dataframe, valora si las filas cumplen con los criterios lógicos especificados (lado derecho del código), y asigna el nuevo valor correcto (lado izquierdo del código). Importante: todos los valores del lado derecho deben ser del mismo tipo.
linelist <- linelist %>%
# Crear nueva columna "age_year " a partir de la columna "age"
mutate(age_years = case_when(
age_unit == "years" ~ age, # si la edad se da en años, asigna el valor original
age_unit == "months" ~ age/12, # si la edad se da en meses, se divide por 12
is.na(age_unit) ~ age, # si falta age_unit, asume años
TRUE ~ NA_real_)) # cualquier otra circunstancia, asigna valor faltanteSi deseas NA en el lado derecho, es posible que tengas que especificar una de las opciones especiales de NA que se indican a continuación. Si los otros valores del lado derecho son caracteres, considera usar “Missing” en su lugar o, de lo contrario, usa NA_character_. Si todos son numéricos, utiliza NA_real_. Si todos son fechas o lógicos, puedes utilizar NA.
-
NA- utilizar para fechas o TRUE/FALSE lógico -
NA_character_- utilizar para caracteres -
NA_real_- uso para numérico
De nuevo, no es probable que te encuentres con estas variaciones a menos que estés utilizando case_when() para crear una nueva columna. Consulta la documentación de R sobre NA para obtener más información.
NULL
NULL es otro valor reservado en R. Es la representación lógica de una declaración que no es ni verdadera ni falsa. Es devuelto por expresiones o funciones cuyos valores son indefinidos. Generalmente no asignes NULL como valor, a menos que escribas funciones o si escribes una aplicación Shiny para devolver NULL en escenarios específicos.
La nulidad puedes evaluarse con is.null() y la conversión puedes hacerse con as.null().
Véase esta entrada del blog sobre la diferencia entre NULL y NA.
NaN
Los valores imposibles se representan con el valor especial NaN. Un ejemplo de esto es cuando se fuerza a R a dividir 0 entre 0. Puedes evaluar esto con is.nan(). También puedes encontrar funciones complementarias incluyendo is.infinite() y is.finite().
Inf
Inf representa un valor infinito, como cuando se divide un número por 0.
Como ejemplo de cómo podría afectar esto a tu trabajo: digamos que tienes un vector/columna z que contiene estos valores: z <- c(1, 22, NA, Inf, NaN, 5)
Si deseas utilizar max() en la columna para encontrar el valor más alto, puedes utilizar el na.rm = TRUE para eliminar el NA del cálculo, pero el Inf y elNaN permanecen y se devolverá Inf. Para resolver esto, puedes utilizar los corchetes [ ] y is.finite() para subconjuntar de manera que sólo se utilicen valores finitos para el cálculo: max(z[is.finite(z)]).
z <- c(1, 22, NA, Inf, NaN, 5)
max(z) # devuelve NA
max(z, na.rm=T) # devuelve Inf
max(z[is.finite(z)]) # devuelve 22Ejemplos
| Comando R | Resultado |
|---|---|
5 / 0 |
Inf |
0 / 0 |
NaN |
5 / NA |
NA |
5 / Inf |0|NA|Inf| "logical"class(NaN)| "numeric"class(Inf)| "numeric"class(NULL)` |
“NULL” |
“NAs introduced by coercion” es un mensaje de aviso común. Esto puede ocurrir si se intenta hacer una conversión ilegal como insertar un valor de carácter en un vector que de otra manera es numérico.
as.numeric(c("10", "20", "thirty", "40"))Warning: NAs introduced by coercion[1] 10 20 NA 40NULL se ignora en un vector.
my_vector <- c(25, NA, 10, NULL) # define el vector
my_vector # lo imprime[1] 25 NA 10La varianza de un número da como resultado NA.
var(22)[1] NA20.3 Funciones útiles
Las siguientes funciones de R base son muy útiles a la hora de evaluar o manejar los valores faltantes:
is.na() y !is.na()
Utiliza is.na() para identificar los valores que faltan, o utiliza su opuesto (con ! delante) para identificar los valores que no faltan. Ambos devuelven un valor lógico (TRUE o FALSE). Recuerda que puedes sum() el vector resultante para contar el número de TRUE, por ejemplo, sum(is.na(linelist$date_outcome)).
my_vector <- c(1, 4, 56, NA, 5, NA, 22)
is.na(my_vector)[1] FALSE FALSE FALSE TRUE FALSE TRUE FALSE!is.na(my_vector)[1] TRUE TRUE TRUE FALSE TRUE FALSE TRUEsum(is.na(my_vector))[1] 2na.omit()
Esta función, si se aplica a un dataframe, eliminará las filas con valores faltantes. También es de R base. Si se aplica a un vector, eliminará los valores NA del vector al que se aplica. Por ejemplo:
na.omit(my_vector)[1] 1 4 56 5 22
attr(,"na.action")
[1] 4 6
attr(,"class")
[1] "omit"drop_na()
Esta es una función tidyr que es útil en un proceso de limpieza de datos. Si se ejecuta con los paréntesis vacíos, elimina las filas con valores faltantes. Si se especifican los nombres de las columnas en los paréntesis, se eliminarán las filas con valores faltantes en esas columnas. También puedes utilizar la sintaxis “tidyselect” para especificar las columnas.
linelist %>%
drop_na(case_id, date_onset, age) # drops rows missing values for any of these columnsna.rm = TRUE
Cuando se ejecuta una función matemática como max(), min(), sum() o mean(), si hay algún valor NA presente el valor devuelto será NA. Este comportamiento por defecto es intencionado, para que avise si falta algún dato.
Puedes evitarlo eliminando los valores faltantes del cálculo. Para ello, incluye el argumento na.rm = TRUE (“na.rm” significa “eliminar NA”).
my_vector <- c(1, 4, 56, NA, 5, NA, 22)
mean(my_vector) [1] NAmean(my_vector, na.rm = TRUE)[1] 17.620.4 Evaluar la ausencia de datos en un dataframe
Puedes utilizar el paquete naniar para evaluar y visualizar la falta de datos del dataframe linelist.
# instala y/o carga el paquete
pacman::p_load(naniar)Cuantificación de la ausencia de datos
Para encontrar el porcentaje de todos los valores que faltan utiliza pct_miss(). Utiliza n_miss() para obtener el número de valores faltantes.
# Porcentaje de TODOS los valores del dataframe que faltan
pct_miss(linelist)[1] 6.688745Las dos funciones siguientes devuelven el porcentaje de filas con algún valor ausente, o que están totalmente completas, respectivamente. Recuerda que NA significa que falta, y que "" o " " no se contarán como faltantes.
# Porcentaje de filas en las que falta algún valor
pct_miss_case(linelist) # usa n_complete() para los recuentos[1] 69.12364# Porcentaje de filas que están completas (no faltan valores)
pct_complete_case(linelist) # usa n_complete() para los recuentos[1] 30.87636Visualización de faltantes
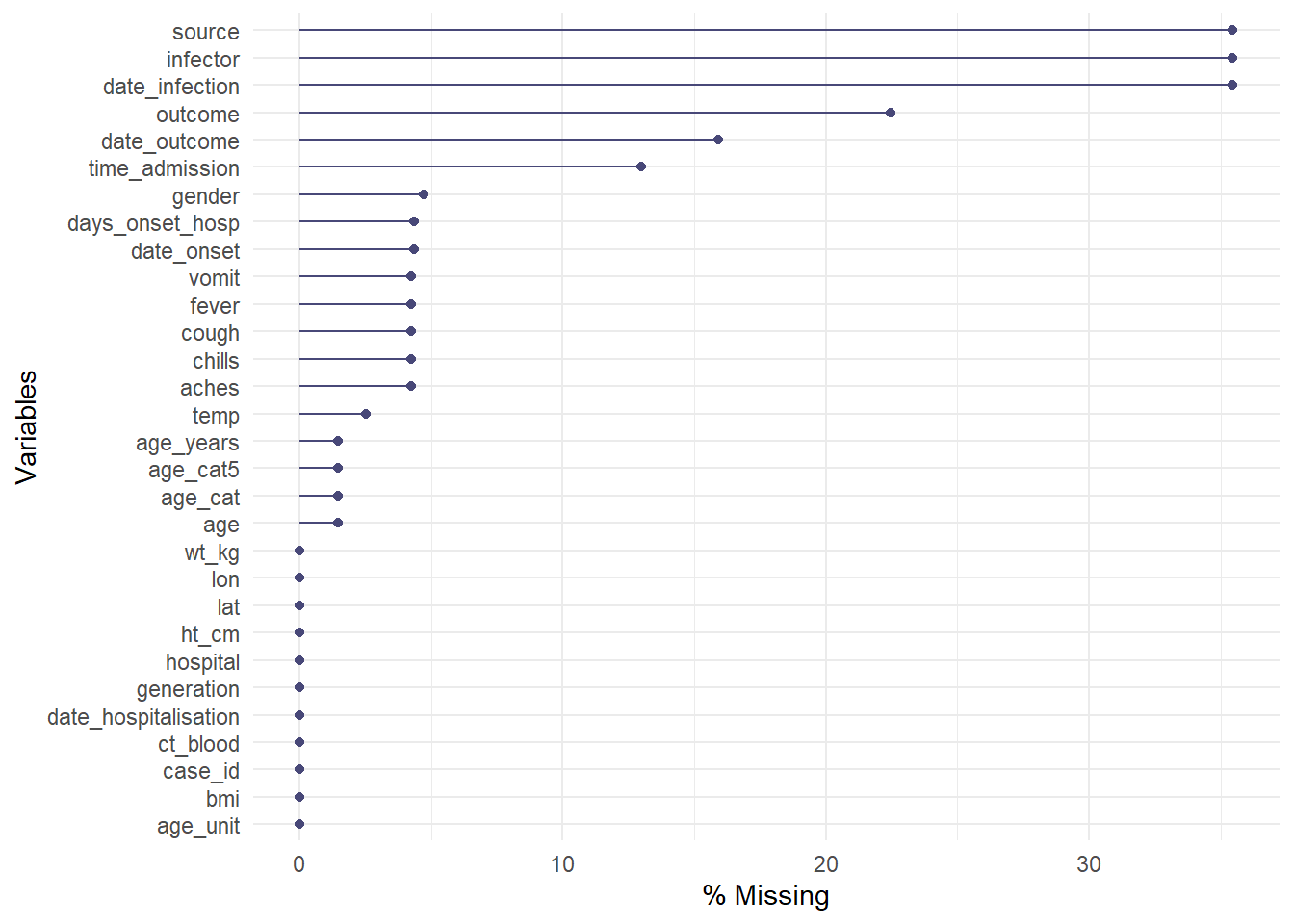
La función gg_miss_var() mostrará el número (o el %) de valores faltantes en cada columna. Algunos matices:
- Puedes añadir un nombre de columna (no entre comillas) al argumento
facet =para ver el gráfico por grupos - Por defecto, se muestran los recuentos en lugar de los porcentajes, cámbialo con
show_pct = TRUE - Puedes añadir etiquetas de eje y de título como para un
ggplot()normal con+ labs(...)
Aquí los datos están conectados con %>% en la función. El argumento facet = también se utiliza para dividir los datos.
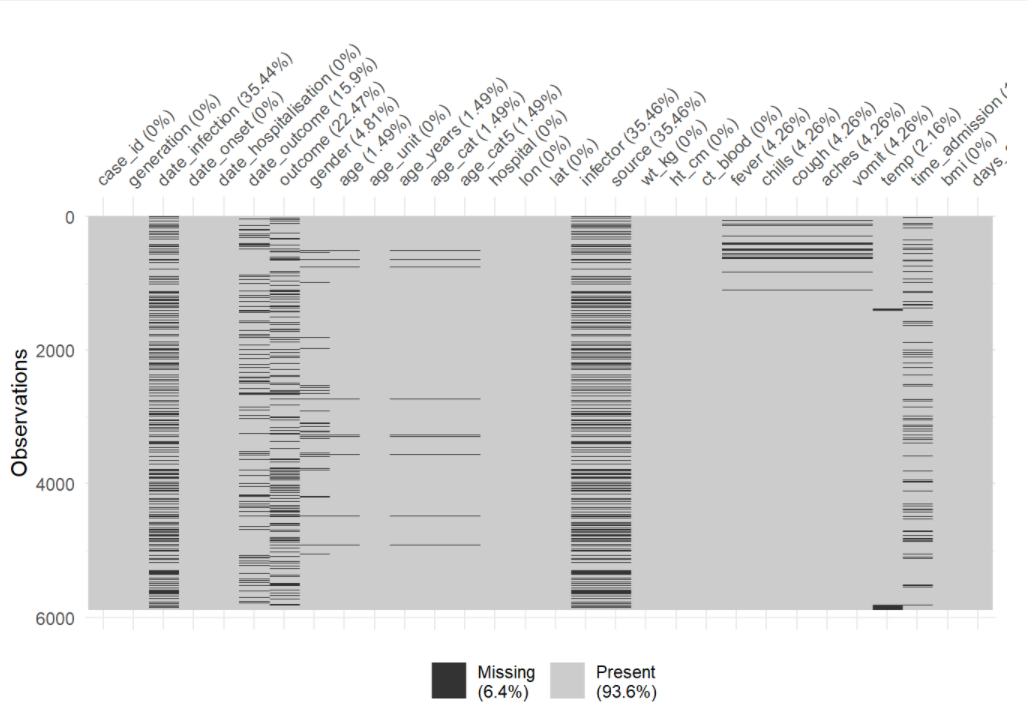
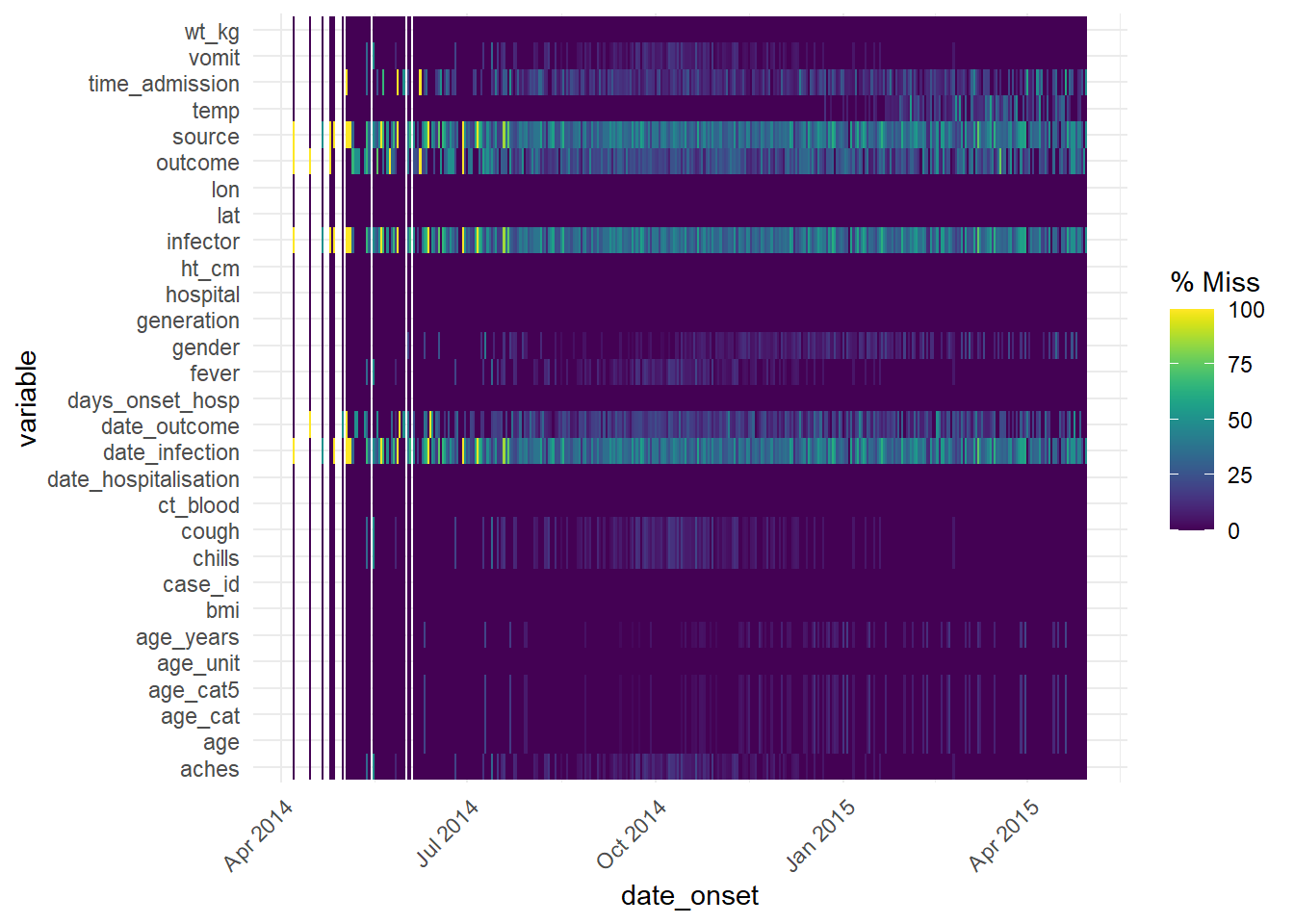
Puedes utilizar vis_miss() para visualizar el dataframe como un mapa de calor, mostrando si cada valor falta o no. También puedes select() determinadas columnas del dataframe y proporcionar sólo esas columnas a la función.
Explorar y visualizar las relaciones de datos faltantes
¿Cómo se visualiza algo que no existe? Por defecto, ggplot() elimina los puntos con valores faltantes de los gráficos.
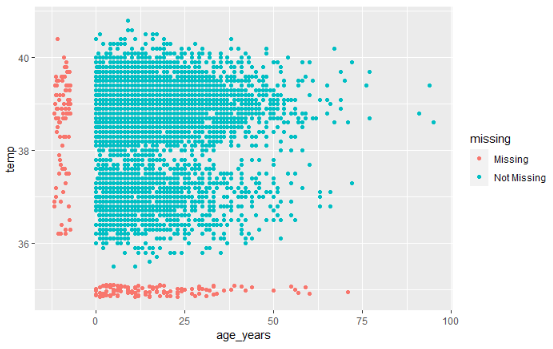
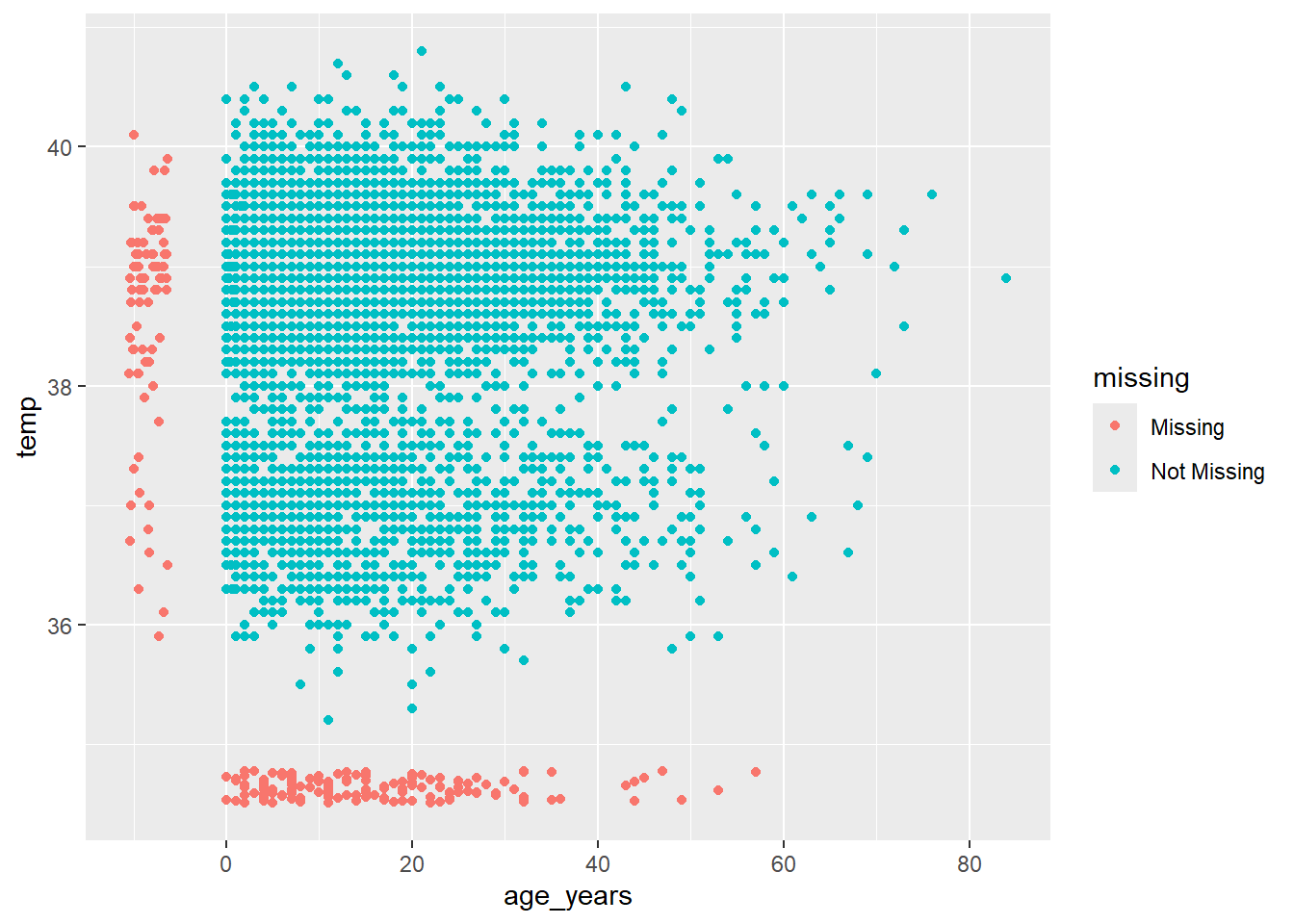
naniar ofrece una solución mediante geom_miss_point(). Al crear un gráfico de dispersión de dos columnas, los registros con uno de los valores ausentes y el otro valor presente se muestran estableciendo los valores ausentes en un 10% más bajo que el valor más bajo de la columna, y coloreándolos de forma distinta.
En el gráfico de dispersión que aparece a continuación, los puntos rojos son registros en los que el valor de una columna está presente pero falta el valor de la otra columna. Esto permite ver la distribución de los valores que faltan en relación con los valores que no faltan.
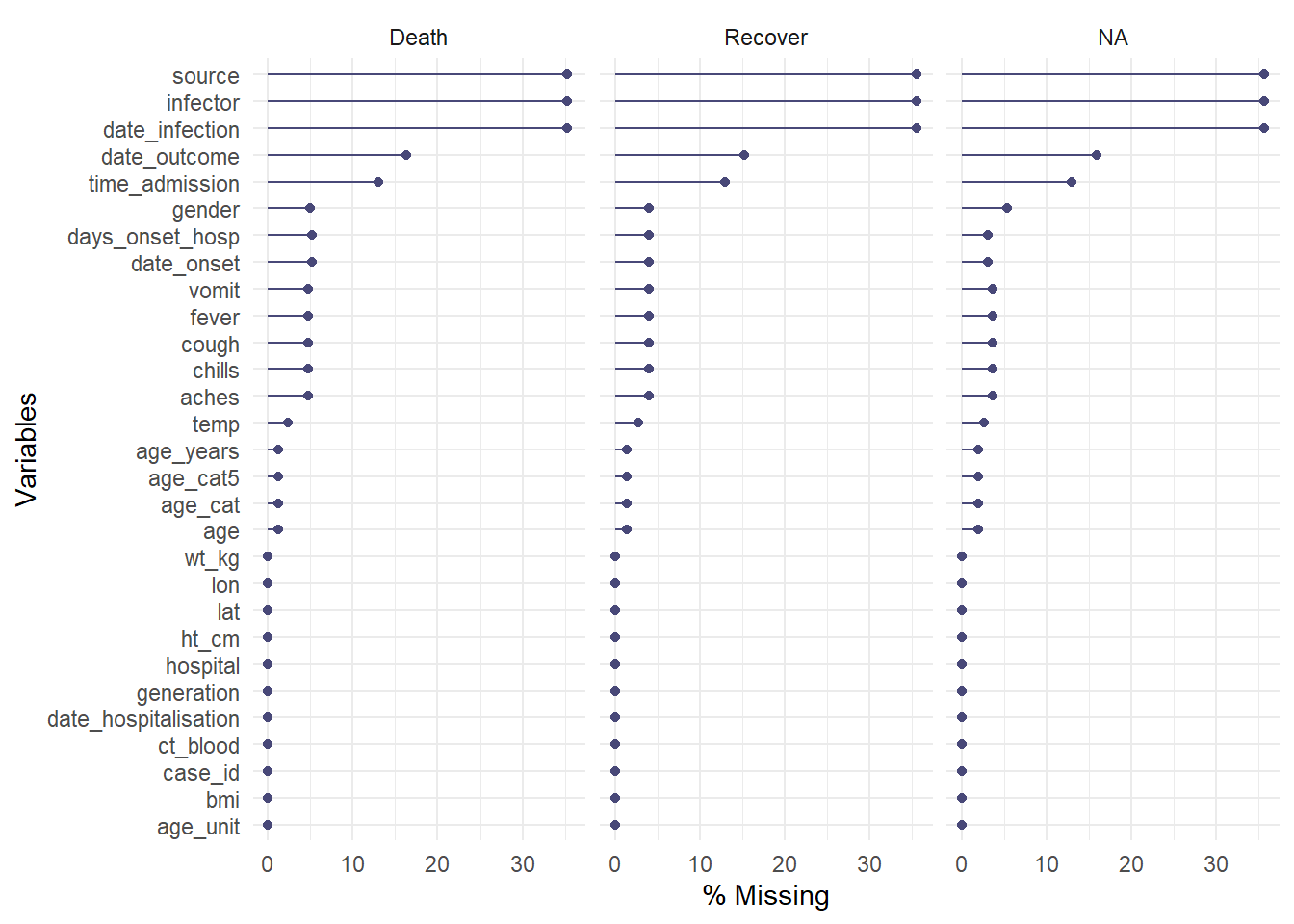
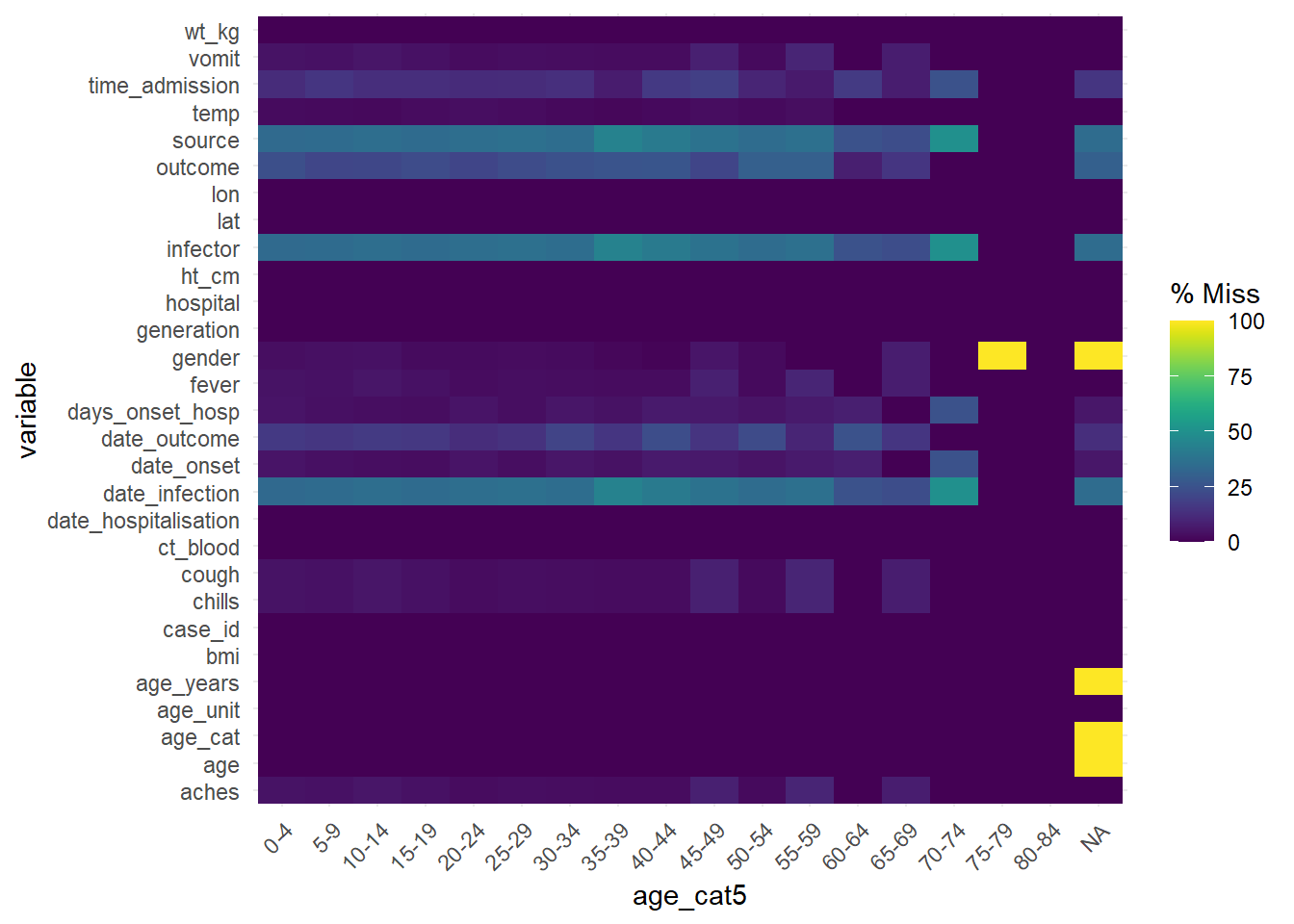
Para evaluar la ausencia en el dataframe estratificado por otra columna, puedes utilizar gg_miss_fct(), que devuelve un mapa de calor del porcentaje de ausencia en el dataframe por una columna de factor/categoría (o fecha):
gg_miss_fct(linelist, age_cat5)Warning: There was 1 warning in `mutate()`.
ℹ In argument: `age_cat5 = (function (x) ...`.
Caused by warning:
! `fct_explicit_na()` was deprecated in forcats 1.0.0.
ℹ Please use `fct_na_value_to_level()` instead.
ℹ The deprecated feature was likely used in the naniar package.
Please report the issue at <https://github.com/njtierney/naniar/issues>.Esta función también se puede utilizar con una columna de fechas para ver cómo ha cambiado la falta de datos en el tiempo:
“Sombra” de las columnas
Otra forma de visualizar la ausencia de valores en una columna es mediante una segunda columna que sea como una “sombra” de esta que puede crear naniar. bind_shadow() crea una columna binaria NA/no NA para cada columna existente, y vincula todas estas nuevas columnas al conjunto de datos original con el apéndice “_NA”. Esto duplica el número de columnas - ver más abajo:
shadowed_linelist <- linelist %>%
bind_shadow()
names(shadowed_linelist) [1] "case_id" "generation"
[3] "date_infection" "date_onset"
[5] "date_hospitalisation" "date_outcome"
[7] "outcome" "gender"
[9] "age" "age_unit"
[11] "age_years" "age_cat"
[13] "age_cat5" "hospital"
[15] "lon" "lat"
[17] "infector" "source"
[19] "wt_kg" "ht_cm"
[21] "ct_blood" "fever"
[23] "chills" "cough"
[25] "aches" "vomit"
[27] "temp" "time_admission"
[29] "bmi" "days_onset_hosp"
[31] "case_id_NA" "generation_NA"
[33] "date_infection_NA" "date_onset_NA"
[35] "date_hospitalisation_NA" "date_outcome_NA"
[37] "outcome_NA" "gender_NA"
[39] "age_NA" "age_unit_NA"
[41] "age_years_NA" "age_cat_NA"
[43] "age_cat5_NA" "hospital_NA"
[45] "lon_NA" "lat_NA"
[47] "infector_NA" "source_NA"
[49] "wt_kg_NA" "ht_cm_NA"
[51] "ct_blood_NA" "fever_NA"
[53] "chills_NA" "cough_NA"
[55] "aches_NA" "vomit_NA"
[57] "temp_NA" "time_admission_NA"
[59] "bmi_NA" "days_onset_hosp_NA" Estas “sombras” de las columnas pueden utilizarse para trazar la proporción de valores que faltan, por cualquier otra columna.
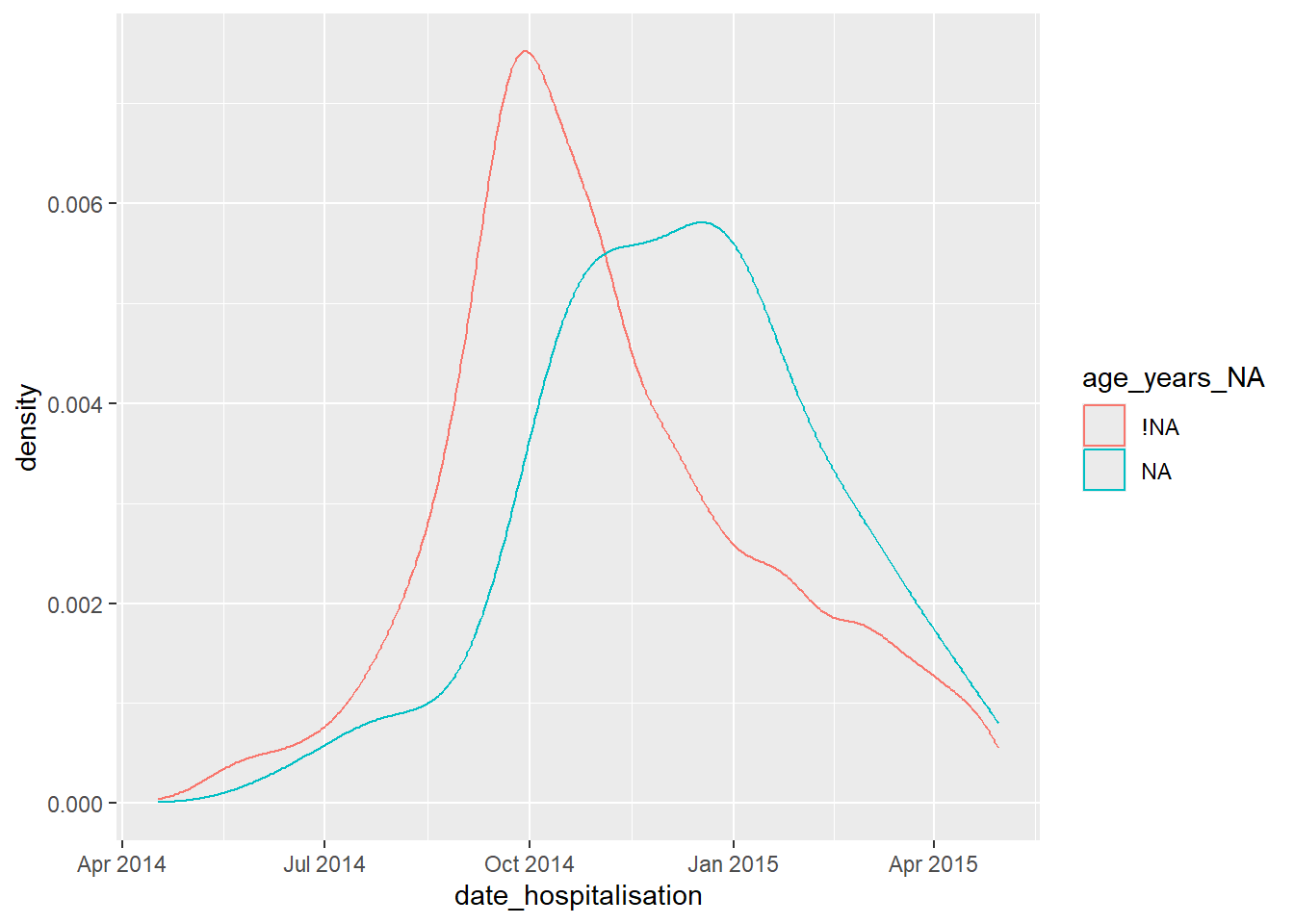
Por ejemplo, el siguiente gráfico muestra la proporción de registros que carecen de days_onset_hosp (número de días desde el inicio de los síntomas hasta la hospitalización), según el valor de ese registro en date_hospitalisation. Esencialmente, se está trazando la densidad de la columna del eje x, pero estratificando los resultados (color = ) por una columna de sombra de interés. Este análisis funciona mejor si el eje-x es una columna numérica o de fecha.
ggplot(data = shadowed_linelist, # dataframe con columnas sombreadas
mapping = aes(x = date_hospitalisation, # columna numérica o de fecha
colour = age_years_NA)) + # columna de sombra de interés
geom_density() # representa las curvas de densidadTambién puedes utilizar estas columnas “sombra” para estratificar un resumen estadístico, como se muestra a continuación:
linelist %>%
bind_shadow() %>% # crea la columna a mostrar
group_by(date_outcome_NA) %>% # columna sombreada para estratificar
summarise(across(
.cols = age_years, # variable de interés para los cálculos
.fns = list("mean" = mean, # estadísticas a calcular
"sd" = sd,
"var" = var,
"min" = min,
"max" = max),
na.rm = TRUE)) # otros argumentos para los cálculos estadísticosWarning: There was 1 warning in `summarise()`.
ℹ In argument: `across(...)`.
ℹ In group 1: `date_outcome_NA = !NA`.
Caused by warning:
! The `...` argument of `across()` is deprecated as of dplyr 1.1.0.
Supply arguments directly to `.fns` through an anonymous function instead.
# Previously
across(a:b, mean, na.rm = TRUE)
# Now
across(a:b, \(x) mean(x, na.rm = TRUE))# A tibble: 2 × 6
date_outcome_NA age_years_mean age_years_sd age_years_var age_years_min
<fct> <dbl> <dbl> <dbl> <dbl>
1 !NA 16.0 12.6 158. 0
2 NA 16.2 12.9 167. 0
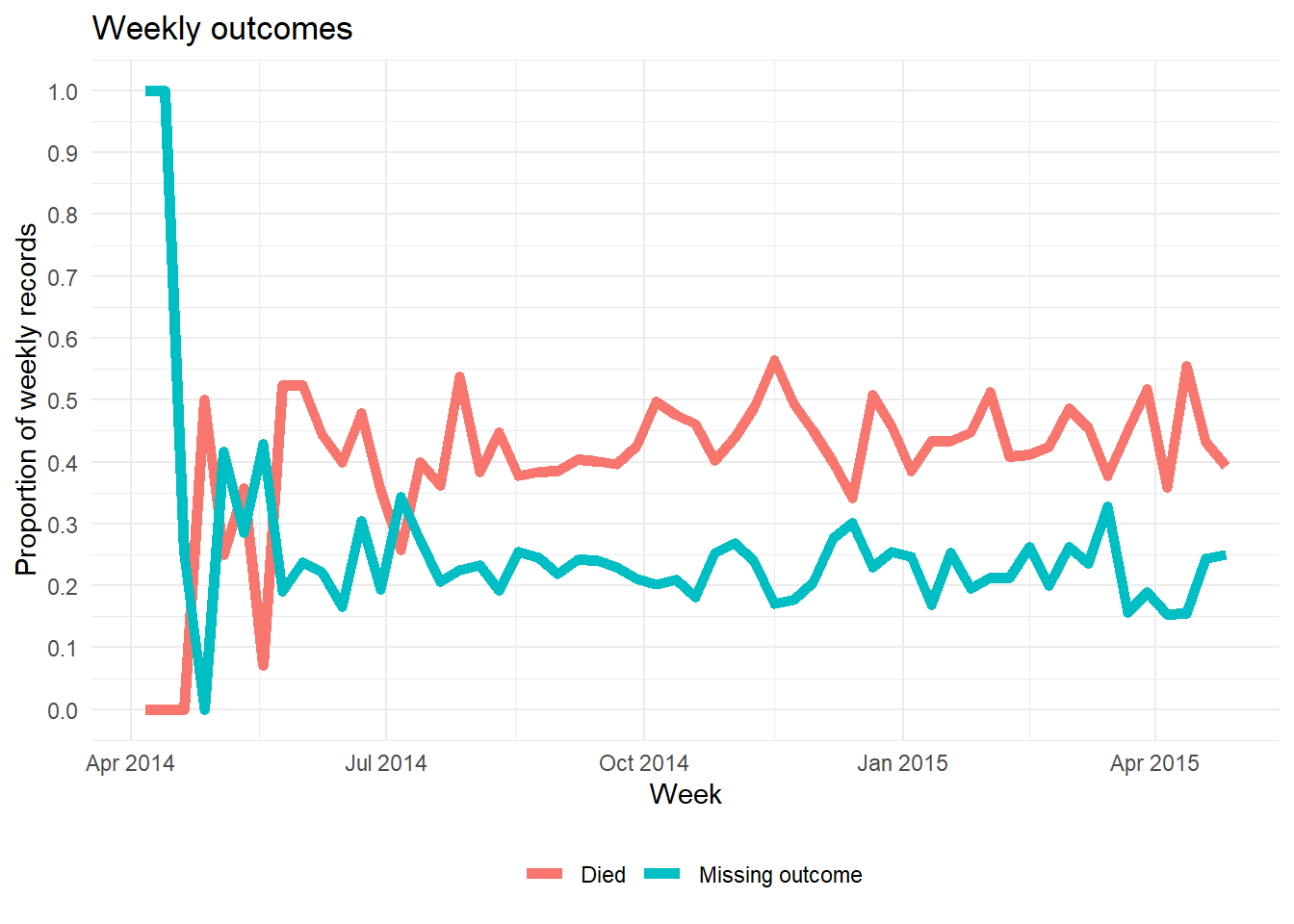
# ℹ 1 more variable: age_years_max <dbl>A continuación se muestra una forma alternativa de trazar la proporción de los valores de una columna que faltan a lo largo del tiempo. No implica naniar. Este ejemplo muestra el porcentaje de observaciones semanales que faltan).
- Agrega los datos en una unidad de tiempo útil (días, semanas, etc.), resumiendo la proporción de observaciones con
NA(y cualquier otro valor de interés) - Representa la proporción que falta como una línea usando
ggplot()
A continuación, tomamos linelist, añadimos una nueva columna para la semana, agrupamos los datos por semana y luego calculamos el porcentaje de registros de esa semana en los que falta el valor. (Nota: si se desea el porcentaje de 7 días el cálculo sería ligeramente diferente).
outcome_missing <- linelist %>%
mutate(week = lubridate::floor_date(date_onset, "week")) %>% # crea una nueva columna semana
group_by(week) %>% # agrupa las filas por semana
summarise( # resume cada semana
n_obs = n(), # número de registros
outcome_missing = sum(is.na(outcome) | outcome == ""), # número de registros en los que falta el valor
outcome_p_miss = outcome_missing / n_obs, # proporción de registros en los que falta el valor
outcome_dead = sum(outcome == "Death", na.rm=T), # número de registros como fallecidos
outcome_p_dead = outcome_dead / n_obs) %>% # proporción de registros como fallecidos
tidyr::pivot_longer(-week, names_to = "statistic") %>% # pivota a formato largo todas las columnas excepto semana, para ggplot
filter(stringr::str_detect(statistic, "_p_")) # conserva sólo los valores de proporcionesEntonces, representamos la proporción que falta como una línea, por semana. Si no estás familiarizado con el paquete de gráficas ggplot2, consulta la página de fundamentos de ggplot.
ggplot(data = outcome_missing)+
geom_line(
mapping = aes(x = week, y = value, group = statistic, color = statistic),
size = 2,
stat = "identity")+
labs(title = "Weekly outcomes",
x = "Week",
y = "Proportion of weekly records") +
scale_color_discrete(
name = "",
labels = c("Died", "Missing outcome"))+
scale_y_continuous(breaks = c(seq(0,1,0.1)))+
theme_minimal()+
theme(legend.position = "bottom")20.5 Uso de datos con valores faltantes
Filtrar las filas con valores faltantes
Para eliminar rápidamente las filas con valores faltantes, utiliza la función dplyr drop_na().
linelist original tiene nrow(linelist) filas. El número ajustado de filas se muestra a continuación:
linelist %>%
drop_na() %>% # elimina las filas con CUALQUIER valor faltante
nrow()[1] 1818Puedes especificar que se eliminen las filas que faltan en determinadas columnas:
linelist %>%
drop_na(date_onset) %>% # elimina las filas en las que falta date_onset
nrow()[1] 5632Puedes listar las columnas una tras otra, o utilizar las funciones de ayuda “tidyselect”:
linelist %>%
drop_na(contains("date")) %>% # elimina las filas a las que faltan valores en cualquier columna "fecha"
nrow()[1] 3029Manejo de NA en ggplot()
A menudo es conveniente informar del número de valores excluidos de un gráfico en un pie de foto. A continuación se muestra un ejemplo:
En ggplot(), puedes añadir labs() y dentro de él un caption =. En el pie, puedes usar str_glue() del paquete stringr para pegar los valores juntos en una frase de forma dinámica para que se ajusten a los datos. Un ejemplo es el siguiente:
- Observa el uso de
\npara una nueva línea. - Ten en cuenta que si varias columnas contribuyen a que los valores no se muestren (por ejemplo, la edad o el sexo si se reflejan en el gráfico), deberás filtrar también esas columnas para calcular correctamente el número no mostrado.
labs(
title = "",
y = "",
x = "",
caption = stringr::str_glue(
"n = {nrow(central_data)} from Central Hospital;
{nrow(central_data %>% filter(is.na(date_onset)))} cases missing date of onset and not shown.")) A veces, puede ser más fácil guardar la cadena como un objeto en comandos anteriores al comando ggplot(), y simplemente referenciar el objeto de cadena nombrado dentro de str_glue().
NA en los factores
Si tu columna de interés es un factor, utiliza fct_explicit_na() del paquete forcats para convertir los valores NA en un valor de carácter. Mira más detalles en la página de Factores. Por defecto, el nuevo valor es “(Missing)” pero puede ajustarse mediante el argumento na_level =.
pacman::p_load(forcats) # load package
linelist <- linelist %>%
mutate(gender = fct_explicit_na(gender, na_level = "Missing"))
levels(linelist$gender)[1] "f" "m" "Missing"20.6 Imputación
A veces, al analizar los datos, será importante “rellenar los huecos” e imputar los datos que faltan Aunque siempre se puede analizar simplemente unos datos después de eliminar todos los valores que faltan, esto puede causar problemas de muchas maneras. He aquí dos ejemplos:
Al eliminar todas las observaciones con valores faltantes o las variables con una gran cantidad de valores faltantes, podría reducir su potencia o la capacidad para realizar algunos tipos de análisis. Por ejemplo, como descubrimos antes, sólo una pequeña fracción de las observaciones de nuestro conjunto de datos de linelist no tiene valores faltantes en todas nuestras variables. Si elimináramos la mayor parte de nuestro conjunto de datos, perderíamos mucha información. Además, la mayoría de nuestras variables tienen una cierta cantidad de valores faltantes; para la mayoría de los análisis, probablemente no sea razonable eliminar todas las variables que tienen muchos valores faltantes.
Dependiendo de la razón por la que faltan datos, el análisis de los datos que no faltan podría conducir a resultados sesgados o engañosos. Por ejemplo, como hemos sabido antes, nos faltan datos de algunos pacientes sobre si han tenido algunos síntomas importantes como fiebre o tos. Pero, como una posibilidad, tal vez esa información no se registró para las personas que obviamente no estaban muy enfermas. En ese caso, si elimináramos estas observaciones, estaríamos excluyendo a algunas de las personas más sanas de nuestro conjunto de datos, lo que podría sesgar los resultados.
Es importante pensar en la razón por la que pueden faltar datos, además de ver cuántos faltan. Esto puede ayudarte a decidir la importancia de imputar los datos que faltan, así como el método de imputación de los datos que faltan que pueda ser mejor en esa situación.
Tipos de datos faltantes
A continuación se presentan tres tipos generales de datos faltantes:
Falta completamente al azar (MCAR Missing Completely at Random). Esto significa que no existe ninguna relación entre la probabilidad de que falten datos y cualquiera de las otras variables de los datos. La probabilidad de que falte es la misma para todos los casos. Pero, si tienes una fuerte razón para creer que tus datos son MCAR, analizar sólo los datos no ausentes sin imputar no sesgará los resultados (aunque puede perder algo de potencia). [PENDIENTE: considerar la discusión de las pruebas estadísticas para MCAR].
Falta al azar (MAR Missing at Random). Este nombre es, en realidad, un poco engañoso, ya que MAR significa que los datos faltan de forma sistemática y predecible en función del resto de la información que se tiene. Por ejemplo, puede que todas las observaciones de nuestro conjunto de datos con un valor ausente de fiebre no se hayan registrado porque se asumió que todos los pacientes con escalofríos y dolores tenían fiebre, por lo que nunca se les tomó la temperatura. Si es cierto, podríamos predecir fácilmente que cada observación que falta con escalofríos y dolores también tiene fiebre y utilizar esta información para imputar nuestros datos que faltan. En la práctica, esto es más bien un espectro. Quizá si un paciente tiene escalofríos y dolores, es más probable que también tenga fiebre si no se le toma la temperatura, pero no siempre. Esto sigue siendo predecible aunque no sea perfectamente predecible. Este es un tipo común de valores faltantes
Desaparición no aleatoria (MNAR Missing not at Random). A veces, también se denomina Falta no aleatoria (NMAR). Esto supone que la probabilidad de que falte un valor NO es sistemática ni predecible utilizando el resto de la información que tenemos, pero tampoco falta al azar. En esta situación, los datos faltan por razones desconocidas o por razones de las que no se tiene ninguna información. Por ejemplo, en nuestro conjunto de datos puede faltar información sobre la edad porque algunos pacientes muy mayores no saben o se niegan a decir su edad. En esta situación, los datos que faltan sobre la edad están relacionados con el propio valor (y, por tanto, no son aleatorios) y no son predecibles en función del resto de la información que tenemos. El MNAR es complejo y, a menudo, la mejor manera de afrontarlo es intentar recopilar más datos o información sobre el motivo por el que faltan los datos en lugar de intentar imputarlos.
En general, la imputación de datos MCAR suele ser bastante sencilla, mientras que la MNAR es muy difícil, si no imposible. Muchos de los métodos comunes de imputación de datos asumen MAR.
Paquetes útiles
Algunos paquetes útiles para la imputación de valores faltantes son Mmisc, missForest (que utiliza bosques aleatorios para imputar los valores faltantes) y mice (Imputación multivariada por ecuaciones encadenadas). Para esta sección sólo utilizaremos el paquete mice, que implementa una variedad de técnicas. El mantenedor del paquete mice ha publicado un libro en línea sobre la imputación Flexible de valores faltantes.
Este es el código para cargar el paquete mice :
pacman::p_load(mice)Imputación de la media
A veces, si estás haciendo un análisis simple o tienes una razón de peso para pensar que puede asumir MCAR, puedes simplemente establecer los valores numéricos que faltan a la media de esa variable. Tal vez podamos asumir que las mediciones de temperatura que faltan en nuestro conjunto de datos eran MCAR o eran simplemente valores normales. Aquí está el código para crear una nueva variable que reemplaza los valores de temperatura faltantes con el valor medio de la temperatura en nuestro conjunto de datos. Sin embargo, en muchas situaciones reemplazar los datos con la media puede conducir a un sesgo, así que ten cuidado.
linelist <- linelist %>%
mutate(temp_replace_na_with_mean = replace_na(temp, mean(temp, na.rm = T)))También puedes realizar un proceso similar para sustituir los datos categóricos por un valor específico. Para nuestro conjunto de datos, imagina que sabes que todas las observaciones con un valor faltante para tu resultado (que puede ser “Muerte” o “Recuperación”) son en realidad personas que han muerto (nota: esto no es realmente cierto para este conjunto de datos):
linelist <- linelist %>%
mutate(outcome_replace_na_with_death = replace_na(outcome, "Death"))Imputación de la regresión
Un método algo más avanzado consiste en utilizar algún tipo de modelo estadístico para predecir cuál es el valor que falta y sustituirlo por el valor predicho. Este es un ejemplo de creación de valores predichos para todas las observaciones en las que falta la temperatura, pero no la edad ni la fiebre, mediante una simple regresión lineal que utiliza el estado de la fiebre y la edad en años como predictores. En la práctica, es conveniente utilizar un modelo mejor que este tipo de enfoque simple.
simple_temperature_model_fit <- lm(temp ~ fever + age_years, data = linelist)
# utilizando nuestro modelo simple de temperatura para predecir valores sólo para las observaciones en las que falta temp.
predictions_for_missing_temps <- predict(simple_temperature_model_fit,
newdata = linelist %>% filter(is.na(temp))) O bien, utilizando el mismo enfoque de modelización a través del paquete mice para crear valores imputados para las observaciones de temperatura que faltan:
model_dataset <- linelist %>%
select(temp, fever, age_years)
temp_imputed <- mice(model_dataset,
method = "norm.predict",
seed = 1,
m = 1,
print = F)Warning: Number of logged events: 1temp_imputed_values <- temp_imputed$imp$tempEste es el mismo tipo de enfoque de algunos métodos más avanzados, como el uso del paquete missForest para sustituir los datos que faltan por valores predichos. En ese caso, el modelo de predicción es un bosque aleatorio en lugar de una regresión lineal. También se pueden utilizar otros tipos de modelos para hacer esto. Sin embargo, aunque este enfoque funciona bien con MCAR, debes tener un poco de cuidado si crees que MAR o MNAR describen con más precisión tu situación. La calidad de tu imputación dependerá de lo bueno que sea tu modelo de predicción e incluso con un modelo muy bueno la variabilidad de los datos imputados puede estar subestimada.
LOCF y BOCF
La última observación trasladada (LOCF) y la observación de referencia trasladada (BOCF) son métodos de imputación para datos de series temporales/longitudinales. La idea es tomar el valor observado anterior como reemplazo de los datos que faltan. Cuando faltan varios valores sucesivamente, el método busca el último valor observado.
La función fill() del paquete tidyr puede utilizarse para la imputación LOCF y BOCF (sin embargo, otros paquetes como HMISC, zoo y data.table también incluyen métodos para hacerlo). Para mostrar la sintaxis de fill(), crearemos un sencillo conjunto de datos de series temporales que contenga el número de casos de una enfermedad para cada trimestre de los años 2000 y 2001. Sin embargo, falta el valor del año para los trimestres posteriores al primero, por lo que tendremos que imputarlos. La unión fill() también se demuestra en la página Pivotar datos.
#crear nuestro conjunto de datos sencillo
disease <- tibble::tribble(
~quarter, ~year, ~cases,
"Q1", 2000, 66013,
"Q2", NA, 69182,
"Q3", NA, 53175,
"Q4", NA, 21001,
"Q1", 2001, 46036,
"Q2", NA, 58842,
"Q3", NA, 44568,
"Q4", NA, 50197)
#imputar los valores del año que faltan:
disease %>% fill(year)# A tibble: 8 × 3
quarter year cases
<chr> <dbl> <dbl>
1 Q1 2000 66013
2 Q2 2000 69182
3 Q3 2000 53175
4 Q4 2000 21001
5 Q1 2001 46036
6 Q2 2001 58842
7 Q3 2001 44568
8 Q4 2001 50197Nota: asegúrate de que tus datos están correctamente ordenados antes de utilizar la función fill(). fill() rellena por defecto “hacia abajo”, pero también puedes imputar valores en diferentes direcciones cambiando el parámetro .direction. Podemos hacer unos datos similares en el que el valor del año se registra sólo al final del año y falta para los trimestres anteriores:
#creando nuestro conjunto de datos ligeramente diferente
disease <- tibble::tribble(
~quarter, ~year, ~cases,
"Q1", NA, 66013,
"Q2", NA, 69182,
"Q3", NA, 53175,
"Q4", 2000, 21001,
"Q1", NA, 46036,
"Q2", NA, 58842,
"Q3", NA, 44568,
"Q4", 2001, 50197)
#imputando los valores del año que faltan en la dirección " up":
disease %>% fill(year, .direction = "up")# A tibble: 8 × 3
quarter year cases
<chr> <dbl> <dbl>
1 Q1 2000 66013
2 Q2 2000 69182
3 Q3 2000 53175
4 Q4 2000 21001
5 Q1 2001 46036
6 Q2 2001 58842
7 Q3 2001 44568
8 Q4 2001 50197En este ejemplo, LOCF y BOCF son claramente lo correcto, pero en situaciones más complicadas puede ser más difícil decidir si estos métodos son apropiados. Por ejemplo, es posible que falten valores de laboratorio para un paciente del hospital después del primer día. A veces, esto puede significar que los valores de laboratorio no cambiaron… ¡pero también podría significar que el paciente se recuperó y sus valores serían muy diferentes después del primer día! Utiliza estos métodos con precaución.
Imputación múltiple
El libro en línea que mencionamos antes, escrito por el autor del paquete mice contiene una explicación detallada de la imputación múltiple y de los motivos por los que conviene utilizarla. Pero, aquí hay una explicación básica del método:
Cuando se realiza una imputación múltiple, se crean múltiples conjuntos de datos con los valores faltantes imputados a valores de datos plausibles (dependiendo de los datos de tu investigación, puedes querer crear más o menos de estos conjuntos de datos imputados, pero el paquete mice establece el número por defecto en 5). La diferencia es que, en lugar de un valor único y específico, cada valor imputado se extrae de una distribución estimada (por lo que incluye cierta aleatoriedad). Como resultado, cada uno de estos conjuntos de datos tendrá valores imputados ligeramente diferentes (sin embargo, los datos no ausentes serán los mismos en cada uno de estos conjuntos de datos imputados). Todavía se utiliza algún tipo de modelo predictivo para hacer la imputación en cada uno de estos nuevos conjuntos de datos (mice tiene muchas opciones para los métodos de predicción, incluyendo Predictive Mean Matching, regresión logística y random forest), pero el paquete mice puede encargarse de muchos de los detalles del modelado.
Entonces, una vez que hayas creado estos nuevos conjuntos de datos imputados, puedes aplicar cualquier modelo estadístico o análisis que estuviera planeando hacer para cada uno de estos nuevos conjuntos de datos imputados y juntar los resultados de estos modelos. Esto funciona muy bien para reducir el sesgo tanto en MCAR como en muchas configuraciones de MAR y a menudo resulta en estimaciones de error estándar más precisas.
He aquí un ejemplo de aplicación del proceso de Imputación Múltiple para predecir la temperatura en nuestro conjunto de datos de linelist utilizando una edad y un estado de fiebre (nuestro conjunto de datos modelo simplificado de arriba):
# imputando valores faltantes para todas las variables de nuestro model_dataset, y creando 10 nuevos conjuntos de datos imputados
multiple_imputation = mice(
model_dataset,
seed = 1,
m = 10,
print = FALSE) Warning: Number of logged events: 1model_fit <- with(multiple_imputation, lm(temp ~ age_years + fever))
base::summary(mice::pool(model_fit)) term estimate std.error statistic df p.value
1 (Intercept) 3.703143e+01 0.0270863456 1.367162e+03 26.83673 1.583113e-66
2 age_years 3.867829e-05 0.0006090202 6.350905e-02 171.44363 9.494351e-01
3 feveryes 1.978044e+00 0.0193587115 1.021785e+02 176.51325 5.666771e-159En este caso, utilizamos el método de imputación por defecto de mice, que es el de Coincidencia de Medias Predictivas. A continuación, utilizamos estos conjuntos de datos imputados para estimar por separado y luego agrupar los resultados de las regresiones lineales simples en cada uno de estos conjuntos de datos. Hay muchos detalles que hemos pasado por alto y muchas configuraciones que puedes ajustar durante el proceso de Imputación Múltiple mientras utilizas el paquete mice. Por ejemplo, no siempre tendrá datos numéricos y podría necesitar utilizar otros métodos de imputación (puedes seguir utilizando el paquete mice para muchos otros tipos de datos y métodos). Pero, para un análisis más robusto cuando los datos faltantes son una preocupación significativa, la Imputación Múltiple es una buena solución que no siempre es mucho más trabajo que hacer un análisis de caso completo.
20.7 Recursos
Viñeta sobre el paquete naniar
Galería de visualizaciones de valores faltantes
Libro en línea sobre imputación múltiple en R por el mantenedor del paquete mice