28 GIS basics
28.1 Présentation
Les caracteristiques geospatiales de vos données peuvent fournir des informations capitales en situation de pandemie.En effet, ils permettent de repondent à des questions tels que:
Ou se trouve les zones à risques de la maladie
Comment les zones à risques evoluent dans le temps
L’accessibilité du plateau medical et la nécessité d’apporter des améliorations
L’accent mis sur le SIG à travers ces pages rend accessible toutes les compétences pertinentes pouvant être utilisées en cas de reponse pandemique.On va voir les methodes simples pour visualiser les donnees spatiales grace aux packages tmap et ggplot.Nous allons aussi passer en revue le package sf qui permet une manipulation des donnees de types vecteurs. Enfin on va aborder brievement les statistiques spatiales à travers les notions de dependance spatiale, l’autocorrelation spatiale et la regression spatiale en utilisant le package spdep
28.2 Mots clés
Quelques mots clés sont definis en bas.Pour une bonne initiation au SIG nous suggérons de suivre l’un des tutoriels proposés dans les références.
Systeme d’Information Geographique(SIG) - Un SIG est un outil informatique ou un environnement qui permet de regrouper, de manipuler, d’analyser et de visualiser les données spatiales
Logiciels SIG
Les logiciels populaires de SIG qui existent, fonctionnent sur la base d’une interface graphique qui interagit apres un clic de l’utlisateur.Ces outils peuvent etre utlisés sans aucun prerequis en programmation et ont comme avantage de pouvoir selectionner et placer des icones et des elements sur une carte manuellement.Les logiciels de SIG les plus connus sont :
ARCGIS - un logiciel commercial developpé par la compagnie ESRI.A ce jour il reste le logiciel de SIG le plus populaire.
QGIS - un logiciel de SIG libre d’utilisation qui fait pratiquement la meme choses que ARCGIS. On peut Telecharger QGIS par ici
Faire du SIG en utilisant R peut sembler frustant pour les premiers pas parce qu’à la place de pointer et cliquer , il faut maintenant ecrire des lignes de commande dans une interface (on a besoin de coder pour obtenir un resultat ).Mais ce changement de paradigme présente des avantages tels que l’automatisation et la reproductibilité dans la conception des carte et les analyses realisées.
Données spatiales
Les types de données utilisées en SIG sont de deux natures: les rasters et les vecteurs
Données vecteurs - Les données vecteurs dont l’utilisation est la plus courante dans l’environnement SIG sont dotés de proprietes géometriques par les sommets et les lignes reliant les sommets. Les données vecteurs largement utilisées peuvent être divisées en trois :
Points - le point est constitué d’un pair de coordonnnées (x,y) qui marque un lieu précis dans l’espace à travers le systeme de coordonnées. Il est la forme simple de représentation des donnees spatiales et peuvent etre utilisé pour situer le lieu d’apparition d’une maladie lors d’une pandémie (la maison du patient) ou un endroit (exemple : hôpital) sur une carte .
Lignes - Une ligne est composée de deux points reliés.Elle a une longueur et peut etre utilisée pour representer les routes ou les cours d’eaux
Polygones - Un polygones est une association minimum de 3 points reliés les uns aux autres .Les caractéristiques d’un polygone sont : le périmètre et l’aire.En pratique ils sont utilisés pour delmiter les contours d’une zone (village) ou une infractructures(hopital)
Données Rasters - A coté des données vecteurs on a des rasters qui sont des matrix constitués par des cellules contenant chacunes des informations par exemple l’altitude, la temperature, la pente, la couverture forestière etc. les rasters peuvent servir de fond de carte pour les donnees vecteurs
Visualisation des données spatiales
Pour afficher sur une carte des donnnées spatiales à partir d’un logiciel de SIG il est nécessaire de la part de l’utilisateur de connaitre l’emplacement des donnees .Lors qu’il nous arrive de manipuler des données vecteurs ce qui est d’ailleurs le plus frequents, les données sont stocker dans des fichiers shapefiles
Shapefiles - Un shapefile est un format de données vecteurs tres repandus dans le monde du SIG et il permet de stocker des donnees pouvant etre des points des lignes et des polygones.C’est en realite une collection ou un ensemnble de fichiers se terminant avec les extensions .shp,.shx et .dbf.Tout ces fichiers doivent etre presents dans un et unique dossiers pour obtenir une donnée shapefile fiable. L’ensemble de ces fichiers shapefile peuvent être comprimer en format zip pour un envoi à travers un mail ou un telechargement à partir d’un site web
Le shapefile contient des information sur un objet de la surface terreste modelisé et sa localisation géographique .Cela est important parce que bien que la terre est une geoide le système de coordonnées bidimentionnel au niveau des shapefiles permet de situer un objet sur la surface de la terre
Systéme de réference de coordonnées - Un CRS est un système de coordonnées utilisé pour localiser géographiquement un objet sur la surface de la terre .Il a quelques composantes clés:
Systeme de Coordonnées - Plusieurs systèmes de coordonnées existent mais il est important de savoir les coodonnées géographiques utilisées sont adossées à quel système.Les degres pour le longitude/latitude sont fréquents mais on note aussi l’usage des coordonnées UTM
Unités - Connaitre l’unité du système de coordonnées(degres ou decimal) est primordiale pour faire certaines analyses spatiales
Datum - C’est une modélisation de la terre revue au fil des annees, il est important de vérifier que les cartes utilisées ont le meme datum ou systeme geodesique
Projection - C’est l’utilisation d’équation mathematique pour projeter la forme geoide de la terre sur une surface plane
Rappellons que les données spatiales peuvent être manipulées sans avoir recours aux outils cartographiques ci-dessous
28.3 Débuter avec le SIG
Il y a quelques éléments clés que vous devrez avoir et auxquels vous devrez penser pour faire une carte. Ces éléments sont les suivants :
Un jeu de données - il peut s’agir d’un format de données spatiales (comme les shapefiles, comme indiqué ci-dessus) ou d’un format non spatial (par exemple, un simple csv).
-
Si votre ensemble de données n’est pas dans un format spatial, vous aurez également besoin d’un jeu données de référence. Les données de référence sont constituées de la représentation spatiale des données et des attributs associés, qui peuvent inclure des éléments contenant les informations relatives à l’emplacement et à l’adresse d’entités spécifiques.
Si vous travaillez avec des limites géographiques prédéfinies (par exemple, des régions administratives), les fichiers de forme de référence peuvent souvent être téléchargés gratuitement depuis une agence gouvernementale ou une organisation de partage de données. En cas de doute, un bon point de départ est de rechercher sur Google ” [régions] shapefile “.
Si vous avez des informations d’adresse, mais pas de latitude et de longitude, vous devrez peut-être utiliser un moteur de géocodage pour obtenir les données de référence spatiale pour vos enregistrements.
Une idée de la manière dont vous voulez présenter les informations de vos ensembles de données à votre public cible. Il existe de nombreux types de cartes, et il est important de réfléchir au type de carte qui correspond le mieux à vos besoins.
Types of maps for visualizing your data
Les types de cartes pour visualiser vos données
Carte chloroplethe - ce sont des cartes thematiques ou les couleurs l’ombrage et les motifs sont utilisés pour representer les valeurs des variables présentes dans les attributs en fonction des unités geograpphiques.Par exemple, une valeur plus grande peut être indiquée par une couleur plus foncée qu’une valeur plus petite. Ce type de carte est particulièrement utile pour visualiser une variable et son évolution dans des régions ou des zones géopolitiques définies.
carte de fréquentation de la densite des cas - C’est Un type de carte thematique ou l’intensité des couleurs est proportionnelle à la valeur de l’observation cela ne fait pas intervenir des zones ou des limites geographiques et adminitratives. Ce genre de carte est utilisé pour montrerles zones à risques ou les lieux à forte concentration de variables observées
Carte de densité de points - Une carte thematique qui utilise des points pour représenter la valeur du variable dans les données.Ce type de carte permet de visualiser les données avec un nuage de point afin d’identifier la presence de clusters ou foyers
Carte à symboles proportionnels (carte à symboles gradués) - une carte thématique similaire à une carte choroplèthe, mais au lieu d’utiliser une couleur pour indiquer la valeur d’un attribut, elle utilise un symbole (généralement un cercle) en relation avec la valeur. Par exemple, une valeur plus grande peut être indiquée par un symbole plus grand qu’une valeur plus petite. Ce type de carte est idéal lorsque vous souhaitez visualiser la taille ou la quantité de vos données dans des régions géographiques.
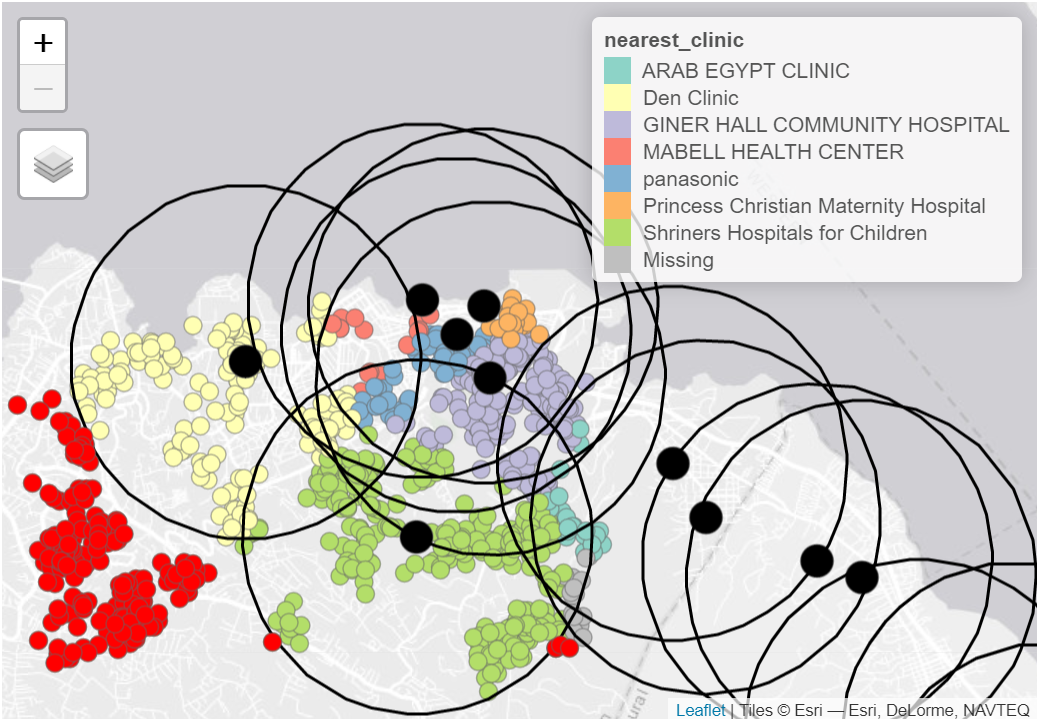
Vous pouvez également combiner plusieurs types de visualisations différentes pour montrer des schémas géographiques complexes. Par exemple, les cas (points) de la carte ci-dessous sont colorés en fonction de l’établissement de santé le plus proche (voir la légende). Les grands cercles rouges montrent les zones de couverture des établissements de santé d’un certain rayon, et les points rouges brillants les cas qui étaient en dehors de toute zone de desserte :
Remarque : L’objectif principal de cette page SIG est basé sur le contexte de la réponse aux épidémies sur le terrain. Par conséquent, le contenu de la page couvrira les manipulations, visualisations et analyses de données spatiales de base.
28.4 Preparation
Chargement packages
Ce morceau de code montre le chargement des paquets nécessaires aux analyses. Dans ce manuel, nous mettons l’accent sur p_load() de pacman, qui installe le paquet si nécessaire et le charge pour l’utiliser. Vous pouvez également charger les paquets installés avec library() de base R. Voir la page sur bases de R pour plus d’informations sur les paquets R.
pacman::p_load(
rio, # Pour importer les données
here, # Pour situer l'emplacement des donnes
tidyverse, # nettoyer manipuler et visualiser les données( inclure le package ggplot2)
sf, # manipuler les donnees spatiales avec le package sf
tmap, # pour produire des cartes simples, fonctionne pour les cartes interactives et statiques
janitor, # pour nettoyer les noms de colonnes
OpenStreetMap, # pour ajouter la carte de base OSM sur la carte ggplot
spdep # statistiques spatiales
) Vous pouvez consulter une vue d’ensemble de tous les paquets R qui traitent des données spatiales sur le site CRAN “Spatial Task View”.
Exemple de données de cas
À des fins de démonstration, nous travaillerons avec un échantillon aléatoire de 1000 cas provenant du dataframe linelist de l’épidémie d’Ebola simulée (d’un point de vue computationnel, travailler avec moins de cas est plus facile à afficher dans ce manuel). Si vous souhaitez suivre le processus, cliquez pour télécharger la linelist “propre” (en tant que fichier .rds).
Comme nous prenons un échantillon aléatoire de cas, vos résultats peuvent être légèrement différents de ce qui est démontré ici lorsque vous exécutez les codes par vous-même.
Importez les données avec la fonction import() du paquet rio (elle gère de nombreux types de fichiers comme .xlsx, .csv, .rds - voir la page Importation et exportation pour plus de détails).
# importer le jeu de données linelist nettoyé
linelist <- import("linelist_cleaned.rds") Ensuite, nous sélectionnons un échantillon aléatoire de 1000 lignes en utilisant sample() de base R.
# générer 1000 numéros de ligne aléatoires, à partir du nombre de lignes de la linelist
sample_rows <- sample(nrow(linelist), 1000)
# creons un sous-ensembles de linelist pour ne garder que les lignes de l'échantillon, et toutes les colonnes
linelist <- linelist[sample_rows,]Maintenant nous voulons convertir cette linelist qui est de classe dataframe, en un objet de classe “sf” (caractéristiques spatiales). Étant donné que la linelist a deux colonnes “lon” et “lat” représentant la longitude et la latitude de la résidence de chaque cas, cela sera facile.
Nous utilisons le package sf (caractéristiques spatiales) et sa fonction st_as_sf() pour créer le nouvel objet que nous appelons linelist_sf. Ce nouvel objet ressemble essentiellement à la linelist, mais les colonnes lon et lat ont été désignées comme des colonnes de coordonnées, et un système de référence de coordonnées (CRS) a été attribué pour l’affichage des points. 4326 identifie nos coordonnées comme étant basées sur le Système géodésique mondial 1984 (WGS84) - qui est la norme pour les coordonnées GPS.
# Creons un objet sf
linelist_sf <- linelist %>%
sf::st_as_sf(coords = c("lon", "lat"), crs = 4326)Voici à quoi ressemble le dataframe original linelist. Dans cette démonstration, nous n’utiliserons que la colonne date_onset et geometry (qui a été construite à partir des champs de longitude et de latitude ci-dessus et qui est la dernière colonne du dataframe).
DT::datatable(head(linelist_sf, 10), rownames = FALSE, options = list(pageLength = 5, scrollX=T), class = 'white-space: nowrap' )shapefiles des limites administratives
Sierra Leone: shapefiles des limites administratives
Par avance, nous avons téléchargé toutes les limites administratives de la Sierra Leone à partir du Humanitarian Data Exchange (HDX) site Web ici. Alternativement, vous pouvez télécharger ces données et toutes les autres données d’exemple pour ce manuel via notre package R, comme expliqué dans la page Télécharger le manuel et les données.
Nous allons maintenant procéder comme suit pour enregistrer le shapefile Admin Level 3 dans R :
- Importer les shapefiles
- Nettoyer les noms des colonnes
- Filtrer les lignes pour ne garder que les zones d’intérêt.
Pour importer un fichier shapefile, nous utilisons la fonction read_sf() de sf. Le chemin du fichier est fourni via here(). - Dans notre cas, le fichier se trouve dans notre projet R dans les sous-dossiers “data”, “gis” et “shp”, avec le nom de fichier “sle_adm3.shp” (voir les pages sur Importation et exportation et Projets R pour plus d’informations). Vous devrez fournir votre propre chemin de fichier.
Ensuite, nous utilisons clean_names() du package janitor pour normaliser les noms de colonnes du fichier shapefile. Nous utilisons également filter() pour ne conserver que les lignes dont le nom d’administrateur est “Western Area Urban” ou “Western Area Rural”.
# ADM3 level clean
sle_adm3 <- sle_adm3_raw %>%
janitor::clean_names() %>% # normaliser les noms de colonnes
filter(admin2name %in% c("Western Area Urban", "Western Area Rural")) # filtre pour garder certaines zonesVous pouvez voir ci-dessous à quoi ressemble le fichier shapefile après importation et nettoyage. Faites défiler vers la droite pour voir s’il y a des colonnes avec le niveau d’administration 0 (pays), le niveau d’administration 1, le niveau d’administration 2, et enfin le niveau d’administration 3. Chaque niveau a un nom de caractère et un identifiant unique “pcode”. Le pcode se développe avec chaque niveau d’administration croissant, par exemple SL (Sierra Leone) -> SL04 (Western) -> SL0410 (Western Area Rural) -> SL040101 (Koya Rural).
Population data
Sierra Leone : Population par ADM3
Ces données peuvent être téléchargées sur HDX (lien ici) ou via notre package R epirhandbook comme expliqué dans la page Télécharger le manuel et les données. Nous utilisons import() pour charger le fichier .csv. Nous passons également le fichier importé à clean_names() pour standardiser la syntaxe des noms de colonnes.
# Population par ADM3
sle_adm3_pop <- import(here("data", "gis", "population", "sle_admpop_adm3_2020.csv")) %>%
janitor::clean_names()Voici à quoi ressemble le fichier de la population. Faites défiler vers la droite pour voir comment chaque juridiction a des colonnes avec la population “masculine”, la population “féminine”, la population “totale”, et la répartition de la population en colonnes par groupe d’âge.
Infractructures sanitaires
Sierra Leone : Données sur les établissements de santé provenant d’OpenStreetMap.
Encore une fois, nous avons téléchargé les emplacements des établissements de santé à partir de HDX ici ou via les instructions dans la page [Télécharger le manuel et les données](#download_book_data.
Nous importons le shapefile des points des établissements avec read_sf(), nettoyons à nouveau les noms des colonnes, et filtrons ensuite pour ne garder que les points étiquetés comme “hôpital”, “clinique”, ou “médecins”.
# Les donnees shapefiles de OSM sur les infractuctures sanitaires
sle_hf <- sf::read_sf(here("data", "gis", "shp", "sle_hf.shp")) %>%
janitor::clean_names() %>%
filter(amenity %in% c("hospital", "clinic", "doctors"))Voici le dataframe obtenu defile jusqu’a en bas pour voir le nom de l’infrastructures sanitaires et les coordonnées geographiques à travers la colonne geometry.
28.5 Visualiser les coordonnées
La manière la plus simple de visualiser des coordonnées X-Y (longitude/latitude, points), dans ce cas de figure, est de les dessiner sous forme de points directement à partir de l’objet linelist_sf que nous avons créé dans la section de préparation.
Le package tmap offre des capacités de cartographie simples à la fois pour le mode statique (mode “plot”) et interactif (mode “view”) avec seulement quelques lignes de code. La syntaxe de tmap est similaire à celle de ggplot2, de sorte que les commandes sont ajoutées les unes aux autres avec +. Vous trouverez plus de détails dans cette vignette.
- Définir le mode tmap. Dans ce cas, nous utiliserons le mode “plot”, qui produit des sorties statiques.
tmap_mode("plot") # choisir soit "view" ou "plot"Ci-dessous, seulement les points sont affichés. tm_shape() prend comme argument : l’objet linelist_sf. Ensuite , Nous ajoutons des points via tm_dots(), en spécifiant la taille et la couleur. Comme linelist_sf est un objet sf, nous avons déjà désigné les deux colonnes qui contiennent les coordonnées lat/long et le système de référence des coordonnées (CRS) :
les points seulement ne sont pas assez informatifs. Nous devons donc également cartographier les limites administratives :
Apres cela, nous utilisons tm_shape() (voir documentation) mais au lieu de fournir le shapefile des points ou les cas sont localisés, nous fournissons le shapefile des limites administratives (polygones).
Avec l’argument bbox = (bbox signifie “bounding box”) nous pouvons spécifier l’emprise des coordonnées. Nous allons d’abord visualiser la carte sans l’emprise bbox, et ensuite avec l’emprise de l’objet.
# Uniquement les frontieres administratives (polygones)
tm_shape(sle_adm3) + # Shapefiles des limites administratives
tm_polygons(col = "#F7F7F7")+ # afficher les polygones en gris clairs
tm_borders(col = "#000000", # parametrer la couleur des bordures et l'epaisseur des lignes
lwd = 2) +
tm_text("admin3name") # le texte de la colonne à afficher pour chaque polygone
#Comme ci-dessus, mais avec un zoom à partir de l'emprise
tm_shape(sle_adm3,
bbox = c(-13.3, 8.43, # sommet
-13.2, 8.5)) + # sommet
tm_polygons(col = "#F7F7F7") +
tm_borders(col = "#000000", lwd = 2) +
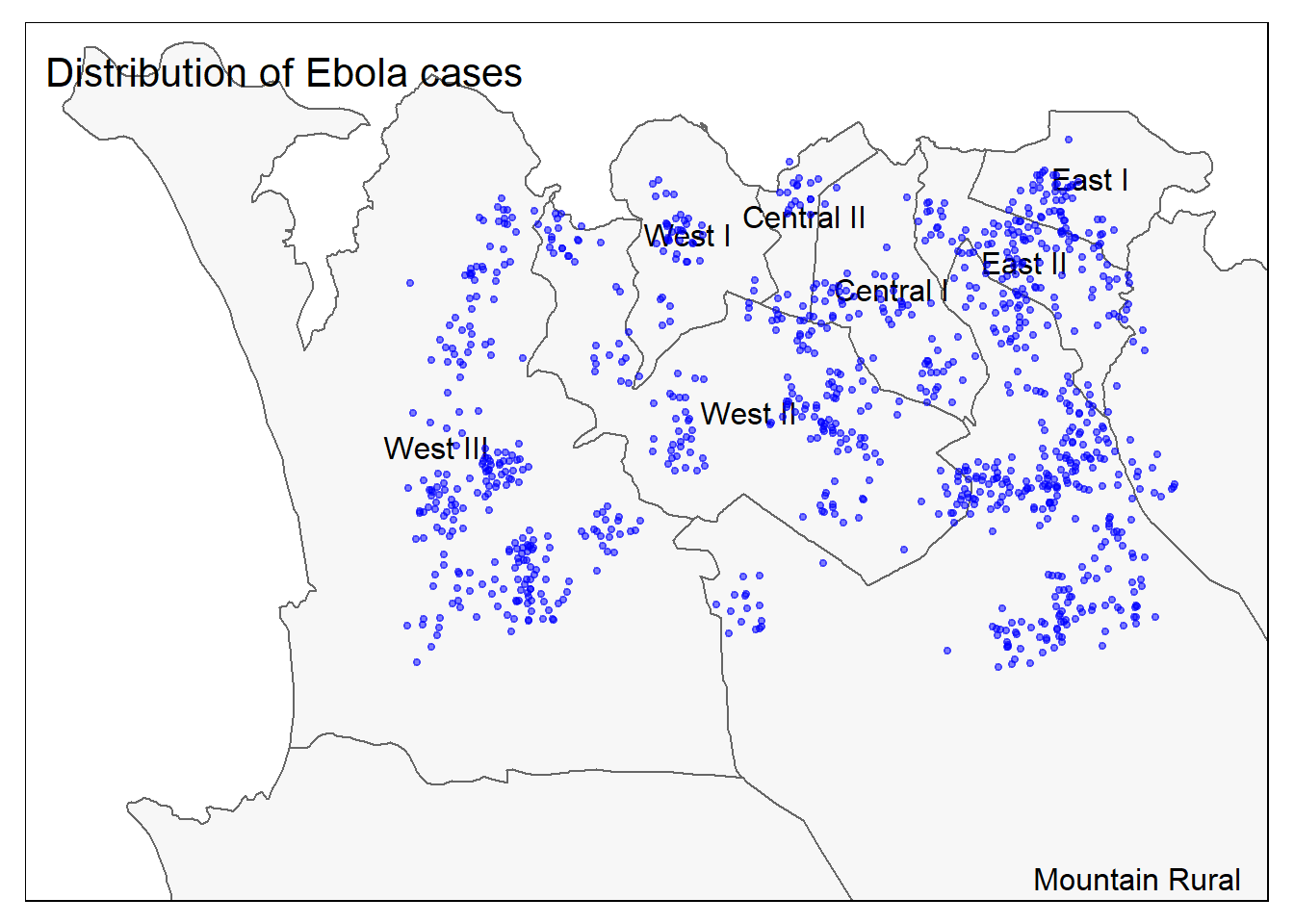
tm_text("admin3name")Et maintenant les points et les polygones ensemble :
# tous ensemble
tm_shape(sle_adm3, bbox = c(-13.3, 8.43, -13.2, 8.5)) + #
tm_polygons(col = "#F7F7F7") +
tm_borders(col = "#000000", lwd = 2) +
tm_text("admin3name")+
tm_shape(linelist_sf) +
tm_dots(size=0.08, col='blue', alpha = 0.5) +
tm_layout(title = "Distribution of Ebola cases") # donner un titre à la cartePour lire une bonne comparaison des options de mappage dans R, consultez cet article de blog.
28.6 Jointures spatiales
Vous êtes peut-être familier avec la jonction de données d’un jeu de données à un autre. Plusieurs méthodes sont présentées à la page Joindre des données de ce manuel. Une jointure spatiale a un objectif similaire mais exploite les relations spatiales. Au lieu de compter sur des valeurs communes dans les colonnes pour faire correspondre correctement les observations, vous pouvez utiliser leurs relations spatiales, comme le fait qu’une observation soit dans une autre, ou le plus proche voisin d’une autre, ou dans un buffer d’un certain rayon d’une autre, etc.
Le package sf offre diverses méthodes de jointures spatiales. Vous trouverez plus de documentation sur la méthode st_join et les types de jointures spatiales dans cette référence.
Points dans le polygone
Attribuer spatialement des unités administratives aux cas
Voici une question intéressante : la liste de cas ne contient aucune information sur les unités administratives des cas. Bien qu’il soit idéal de collecter ces informations au cours de la phase initiale de collecte des données, nous pouvons également attribuer des unités administratives aux cas individuels sur la base de leurs relations spatiales (c’est-à-dire l’intersection d’un point avec un polygone).
Ci-dessous, nous allons croiser spatialement les emplacements de nos cas (points) avec les limites d’ADM3 (polygones) :
- Commencer par l’objet linelist (points)
- Jointure spatiale aux limites, en définissant le type de jointure comme etant “st_intersects”.
- Utilisez
select()pour ne garder que certaines des colonnes de la nouvelle limite administrative.
linelist_adm <- linelist_sf %>%
# joindre le fichier des limites administratives à linelist sur la base de l'intersection spatiale.
sf::st_join(sle_adm3, join = st_intersects)Toutes les colonnes de sle_adms ont été ajoutées à la linelist ! Chaque cas a maintenant des colonnes détaillant les niveaux administratifs dont il fait partie. Dans cet exemple, nous voulons seulement garder deux des nouvelles colonnes (niveau administratif 3), donc nous sélectionnons() les anciens noms de colonnes et seulement les deux supplémentaires d’intérêt :
linelist_adm <- linelist_sf %>%
# joindre le fichier des limites administratives au fichier linelist, sur la base de l'intersection spatiale.
sf::st_join(sle_adm3, join = st_intersects) %>%
# Conservez les anciens noms de colonnes et deux nouveaux noms admin d'intérêt
select(names(linelist_sf), admin3name, admin3pcod)Ci-dessous, à des fins d’affichage, vous pouvez voir les dix premiers cas et les limites administratives de niveau 3(ADM3) qui y ont été rattachées, en fonction de l’endroit où le point a croisé les formes polygonales.
# Vous verrez maintenant les noms ADM3 attachés à chaque cas.
linelist_adm %>% select(case_id, admin3name, admin3pcod)Simple feature collection with 1000 features and 3 fields
Geometry type: POINT
Dimension: XY
Bounding box: xmin: -13.2701 ymin: 8.447961 xmax: -13.20576 ymax: 8.491748
Geodetic CRS: WGS 84
First 10 features:
case_id admin3name admin3pcod geometry
3873 8ab204 East I SL040203 POINT (-13.21179 8.485098)
4500 2314ad Central II SL040202 POINT (-13.23624 8.488154)
1622 ab27c7 West III SL040208 POINT (-13.25969 8.455222)
2772 430f99 East III SL040205 POINT (-13.20595 8.462697)
5822 e9315f Mountain Rural SL040102 POINT (-13.21879 8.462427)
393 2aedd6 West III SL040208 POINT (-13.25856 8.453381)
4300 89a9a9 West III SL040208 POINT (-13.26543 8.46738)
3565 6bec10 West II SL040207 POINT (-13.24752 8.468575)
3214 8f7caa East II SL040204 POINT (-13.21347 8.481946)
3101 8e4378 West II SL040207 POINT (-13.24149 8.476931)Nous pouvons maintenant décrire nos cas par unité administrative - ce que nous ne pouvions pas faire avant la jointure spatiale !
# Créer un nouveau dataframe contenant le nombre de cas par unité administrative.
case_adm3 <- linelist_adm %>% # commencer avec linelist avec de nouveaux admin colonnes
as_tibble() %>% # convert en format tibble pour un meilleur affichage
group_by(admin3pcod, admin3name) %>% # regrouper par unite admin, à la fois par le nom et le pcode
summarise(cases = n()) %>% # utilisons la fonction summarize et comptons les lignes
arrange(desc(cases)) # arrangement par ordre decroissant
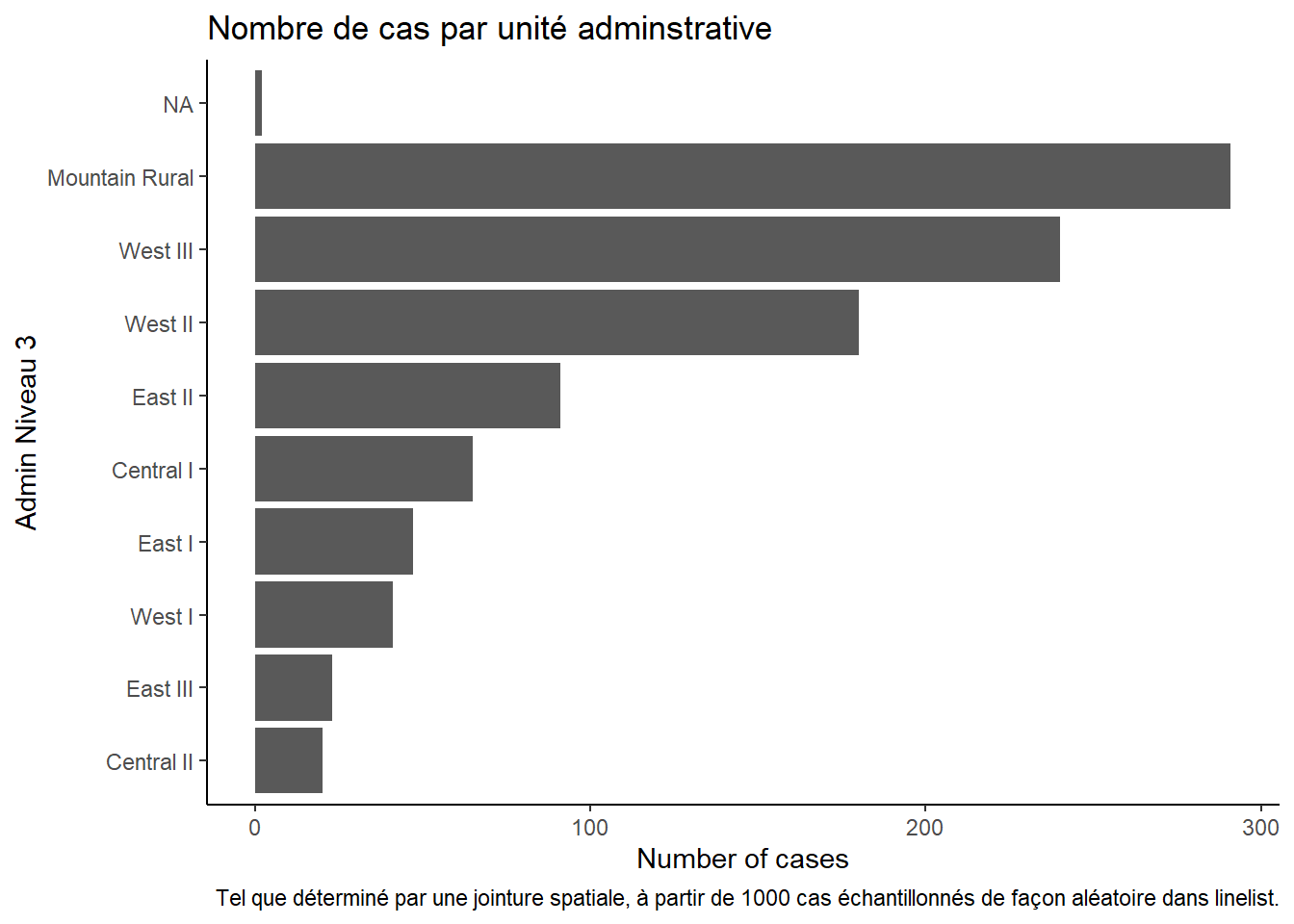
case_adm3# A tibble: 10 × 3
# Groups: admin3pcod [10]
admin3pcod admin3name cases
<chr> <chr> <int>
1 SL040102 Mountain Rural 291
2 SL040208 West III 240
3 SL040207 West II 180
4 SL040204 East II 91
5 SL040201 Central I 65
6 SL040203 East I 47
7 SL040206 West I 41
8 SL040205 East III 23
9 SL040202 Central II 20
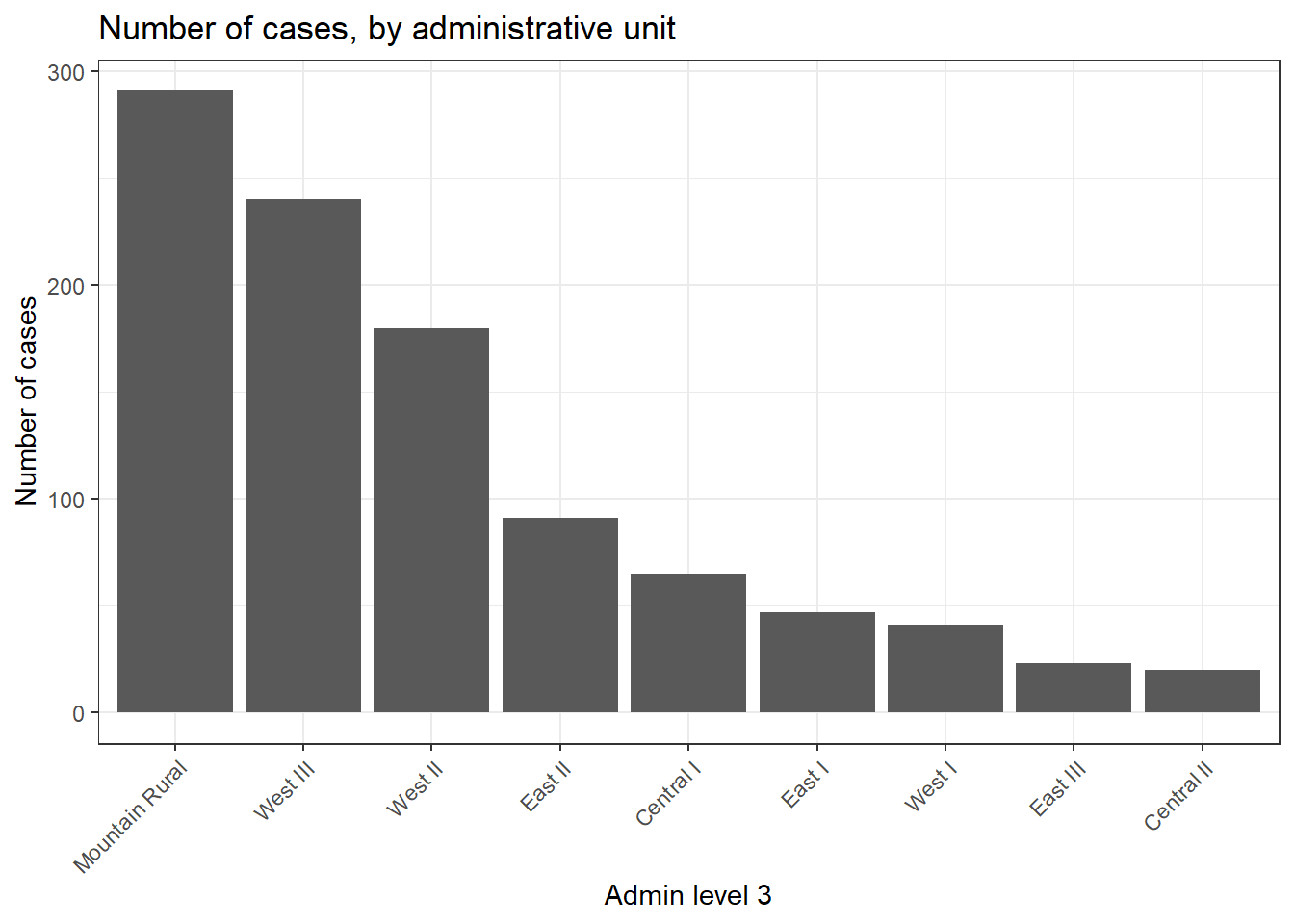
10 <NA> <NA> 2Nous pouvons également créer un diagramme en barres du nombre de cas par unité administrative.
Dans cet exemple, nous commençons le ggplot() avec la linelist_adm, afin de pouvoir appliquer des fonctions de facteurs comme fct_infreq() qui ordonne les barres par fréquence (voir la page sur les Facteurs pour des conseils).
ggplot(
data = linelist_adm, # Debuter avec linelist qui contient les infos sur les unites admin
mapping = aes(
x = fct_rev(fct_infreq(admin3name))))+ # L'axe des x est constitué d'unités administratives, classées par fréquence (inversée).
geom_bar()+ # créer des barres, la hauteur est le nombre de lignes
coord_flip()+ # retournement des axes X et Y pour une lecture plus aisée des unités d'admin
theme_classic()+ # simplifier le fond
labs( # titres et les labels
x = "Admin Niveau 3",
y = "Number of cases",
title = "Nombre de cas par unité adminstrative",
caption = "Tel que déterminé par une jointure spatiale, à partir de 1000 cas échantillonnés de façon aléatoire dans linelist."
)Le voisin le plus proche
Trouver l’établissement de santé le plus proche / la zone de captage
Il peut être utile de savoir où sont situés les établissements de santé par rapport aux foyers de maladies.
Nous pouvons utiliser la méthode de jointure st_nearest_feature de la fonction st_join() (package sf) pour visualiser l’établissement de santé le plus proche des cas individuels.
- Nous commençons avec le fichier de forme linelist
linelist_sf.
- Nous joignons spatialement avec
sle_hf, qui est les emplacements des établissements de santé et des cliniques (points)
# Établissement de santé le plus proche de chaque cas
linelist_sf_hf <- linelist_sf %>% # commencons avec linelist shapefile
st_join(sle_hf, join = st_nearest_feature) %>% # données de la clinique la plus proche jointes aux données du cas
select(case_id, osm_id, name, amenity) %>% # conserver les colonnes d'intérêt, notamment l'identifiant, le nom, le type et la géométrie de l'établissement de santé
rename("nearest_clinic" = "name") # renommer pour plus de clartéNous pouvons voir ci-dessous (les 50 premières lignes) que chaque cas a maintenant des données sur la clinique/hôpital le plus proche.
Nous pouvons voir que “Den Clinic” est l’établissement de santé le plus proche pour environ 30 % des cas.
# Nombre de cas par établissement de santé
hf_catchment <- linelist_sf_hf %>% # commencer par linelist comprenant les données de la clinique la plus proche
as.data.frame() %>% # convertir de shapefile à dataframe
count(nearest_clinic, # compter les lignes par "nom" (de la clinique)
name = "case_n") %>% # assign new counts column as "case_n"
arrange(desc(case_n)) # classer par ordre décroissant
hf_catchment # Afficher sur la console nearest_clinic case_n
1 Den Clinic 379
2 Shriners Hospitals for Children 345
3 GINER HALL COMMUNITY HOSPITAL 160
4 panasonic 42
5 Princess Christian Maternity Hospital 27
6 ARAB EGYPT CLINIC 22
7 <NA> 17
8 MABELL HEALTH CENTER 8Pour visualiser les résultats, nous pouvons utiliser tmap - cette fois-ci en mode interactif pour une visualisation plus facile.
tmap_mode("view") # Utiliser tmap en mode interactive
# Visualiser les points ou sont localisés les cas et cliniques
tm_shape(linelist_sf_hf) + # visualiser les cas
tm_dots(size=0.08, # cas colorés par la clinique la plus proche
col='nearest_clinic') +
tm_shape(sle_hf) + # tracer les cliniques en gros points noirs
tm_dots(size=0.3, col='black', alpha = 0.4) +
tm_text("name") + # superposition du nom de l'installation
tm_view(set.view = c(-13.2284, 8.4699, 13), # ajuster le zoom (coordonnées du centre, zoom)
set.zoom.limits = c(13,14))+
tm_layout(title = "Cas, colorés par la clinique la plus proche")Buffer
Nous pouvons également explorer combien de cas sont situés à moins de 2,5 km (~30 minutes) de distance de marche de l’établissement de santé le plus proche.
Remarque : pour des calculs de distance plus précis, il est préférable de reprojeter votre objet sf dans le système de projection de la carte locale, tel que l’UTM (Terre projetée sur une surface plane). Dans cet exemple, pour des raisons de simplicité, nous nous en tiendrons au système de coordonnées géograhpiques World Geodetic System (WGS84) (la Terre est représentée par une surface sphérique / ronde, les unités sont donc en degrés décimaux). Nous utiliserons une conversion générale de : 1 degré décimal = ~111km.
Pour plus d’informations sur les projections cartographiques et les systèmes de coordonnées, consultez cet article esri. Ce blog traite des différents types de projection cartographique et de la manière de choisir une projection appropriée en fonction de la zone d’intérêt et du contexte de votre carte / analyse.
Tout d’abord, créez un tampon circulaire d’un rayon de ~2,5 km autour de chaque établissement de santé. Ceci est fait avec la fonction st_buffer() de tmap. Parce que l’unité de la carte est en degrés décimaux lat/long, c’est ainsi que “0,02” est interprété. Si le système de coordonnées de votre carte est en mètres, le nombre doit être fourni en mètres.
sle_hf_2k <- sle_hf %>%
st_buffer(dist=0.02) # degrés décimaux se traduisant par environ 2,5 km Ci-dessous, nous traçons les zones tampons elles-mêmes, avec les valeurs de :
tmap_mode("plot")
# Creons une zone tampon circulaire
tm_shape(sle_hf_2k) +
tm_borders(col = "black", lwd = 2)+
tm_shape(sle_hf) + # materialiser les installations cliniques avec de gros points rouges
tm_dots(size=0.3, col='black') Ensuite, nous intersectons ces tampons avec les cas (points) en utilisant st_join() et le type de jointure de st_intersects. C’est-à-dire que les données de la zone tampon sont jointes aux points qu’ils croisent.
# Intersecter les cas observés avec la zone tampon
linelist_sf_hf_2k <- linelist_sf_hf %>%
st_join(sle_hf_2k, join = st_intersects, left = TRUE) %>%
filter(osm_id.x==osm_id.y | is.na(osm_id.y)) %>%
select(case_id, osm_id.x, nearest_clinic, amenity.x, osm_id.y)Maintenant, nous pouvons compter les résultats : nrow(linelist_sf_hf_2k[is.na(linelist_sf_hf_2k$osm_id.y),]) sur 1000 cas ne recoupent aucun tampon (cette valeur est manquante), et vivent donc à plus de 30 minutes de marche de l’établissement de santé le plus proche.
# Cas qui n'ont pas été croisés avec l'un des tampons de l'établissement de santé
linelist_sf_hf_2k %>%
filter(is.na(osm_id.y)) %>%
nrow()[1] 1000Nous pouvons visualiser les résultats de telle sorte que les cas qui n’ont intersecté aucun tampon apparaissent en rouge.
tmap_mode("view")
# Affichez d'abord les cas en points
tm_shape(linelist_sf_hf) +
tm_dots(size=0.08, col='nearest_clinic') +
# tracer les installations cliniques en gros points noirs
tm_shape(sle_hf) +
tm_dots(size=0.3, col='black')+
# Superposez ensuite les zones tampons des établissements de santé sous forme de polylignes.
tm_shape(sle_hf_2k) +
tm_borders(col = "black", lwd = 2) +
# Highlight cases that are not part of any health facility buffers
# en points noirs
tm_shape(linelist_sf_hf_2k %>% filter(is.na(osm_id.y))) +
tm_dots(size=0.1, col='red') +
tm_view(set.view = c(-13.2284,8.4699, 13), set.zoom.limits = c(13,14))+
# ajouter le titre
tm_layout(title = "Nombre de Cas par zone couverture des cliniques ")les autres jointures spatiales
Les valeurs alternatives pour l’argument join comprennent (de la documentation)
- st_contains_properly
- st_contains
- st_covered_by
- st_covers
- st_crosses
- st_disjoint
- st_equals_exact
- st_equals
- st_is_within_distance
- st_nearest_feature
- st_overlaps
- st_touches
- st_within
28.7 Choropleth maps
Les cartes choroplèthes peuvent être utiles pour visualiser vos données par zone prédéfinie, généralement une unité administrative ou une zone de santé. Dans le cadre de la réponse aux épidémies, cela peut aider à cibler l’allocation des ressources pour des zones spécifiques présentant des taux d’incidence élevés, par exemple.
Maintenant que nous avons les noms des unités administratives attribués à tous les cas (voir la section sur les jointures spatiales, ci-dessus), nous pouvons commencer à cartographier le nombre de cas par zone (cartes choroplèthes).
Puisque nous disposons également des données de population par ADM3, nous pouvons ajouter ces informations à la table case_adm3 créée précédemment.
Nous commençons avec le cadre de données créé à l’étape précédente case_adm3, qui est un tableau récapitulatif de chaque unité administrative et de son nombre de cas.
- Les données de population
sle_adm3_popsont jointes à l’aide d’unleft_join()de dplyr sur la base des valeurs communes à la colonneadmin3pcoddans le dataframecase_adm3, et à la colonneadm_pcodedans le dataframesle_adm3_pop. Voir la page Joindre des données).
-
select()est appliqué au nouveau dataframe, pour ne garder que les colonnes utiles -totalest la population totale.
- Les cas pour 10.000 habitants sont calculés comme une nouvelle colonne avec
mutate().
# Ajouter les données de la population et calculer les cas pour 10K de population
case_adm3 <- case_adm3 %>%
left_join(sle_adm3_pop, #ajouter des colonnes à partir du jeu données pop
by = c("admin3pcod" = "adm3_pcode")) %>% # jointure basee sur les valeurs communes à ces deux colonnes
select(names(case_adm3), total) %>% # ne conserver que les colonnes importantes, notamment la population totale
mutate(case_10kpop = round(cases/total * 10000, 3)) # créer une nouvelle colonne avec le taux de cas pour 10000, arrondi à 3 décimales
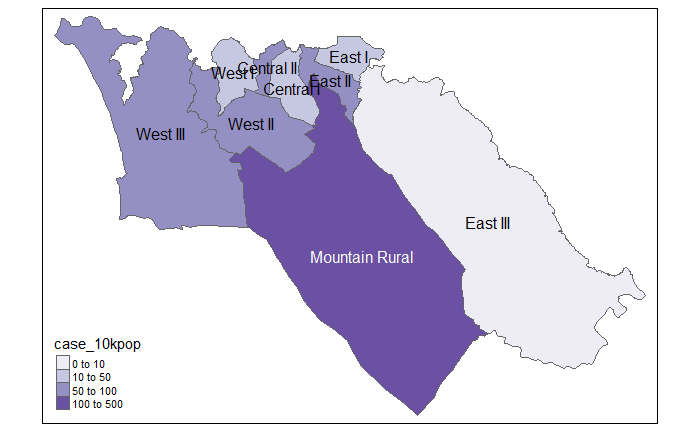
case_adm3 # imprimer sur la console pour l'affichage# A tibble: 10 × 5
# Groups: admin3pcod [10]
admin3pcod admin3name cases total case_10kpop
<chr> <chr> <int> <int> <dbl>
1 SL040102 Mountain Rural 291 33993 85.6
2 SL040208 West III 240 210252 11.4
3 SL040207 West II 180 145109 12.4
4 SL040204 East II 91 99821 9.12
5 SL040201 Central I 65 69683 9.33
6 SL040203 East I 47 68284 6.88
7 SL040206 West I 41 60186 6.81
8 SL040205 East III 23 500134 0.46
9 SL040202 Central II 20 23874 8.38
10 <NA> <NA> 2 NA NA faire une Jointure cette table au shapefile ADM3 polygones pour la cartographie.
case_adm3_sf <- case_adm3 %>% # Commencer par les cas et classer par unité administrative
left_join(sle_adm3, by="admin3pcod") %>% # jointure aux données shapefile par colonne commune
select(objectid, admin3pcod, # ne conserver que certaines colonnes d'intérêt
admin3name = admin3name.x, # nettoyer le nom de ce column
admin2name, admin1name,
cases, total, case_10kpop,
geometry) %>% # conserver la géométrie pour que les polygones puissent être cartographier
drop_na(objectid) %>% # supprimer les lignes vides
st_as_sf() # convertir en shapefileCartographier le resultat
# tmap mode
tmap_mode("plot") # carte statique
# visualiser les polygones
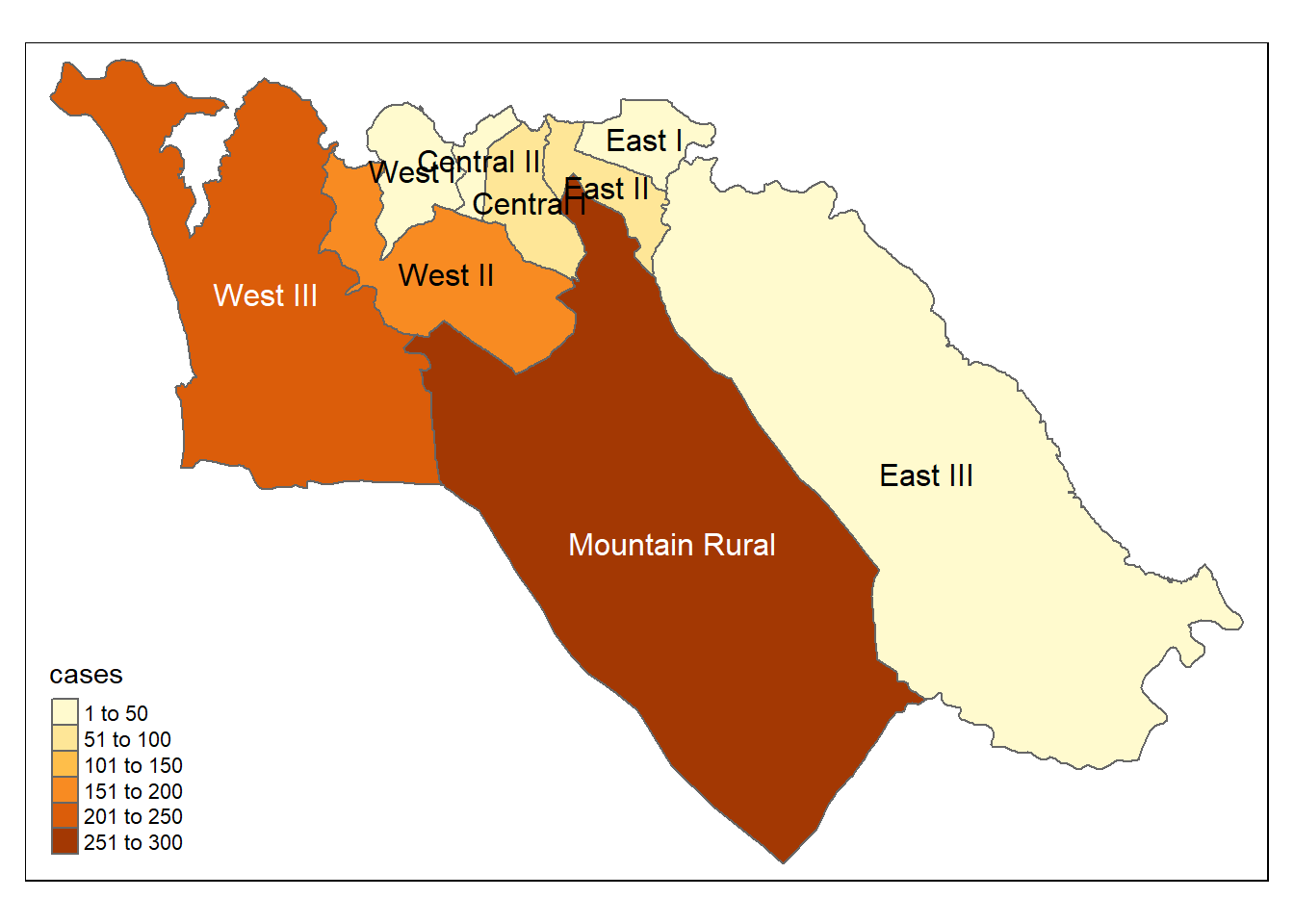
tm_shape(case_adm3_sf) +
tm_polygons("cases") + # colorier en fonction du nombre de cas
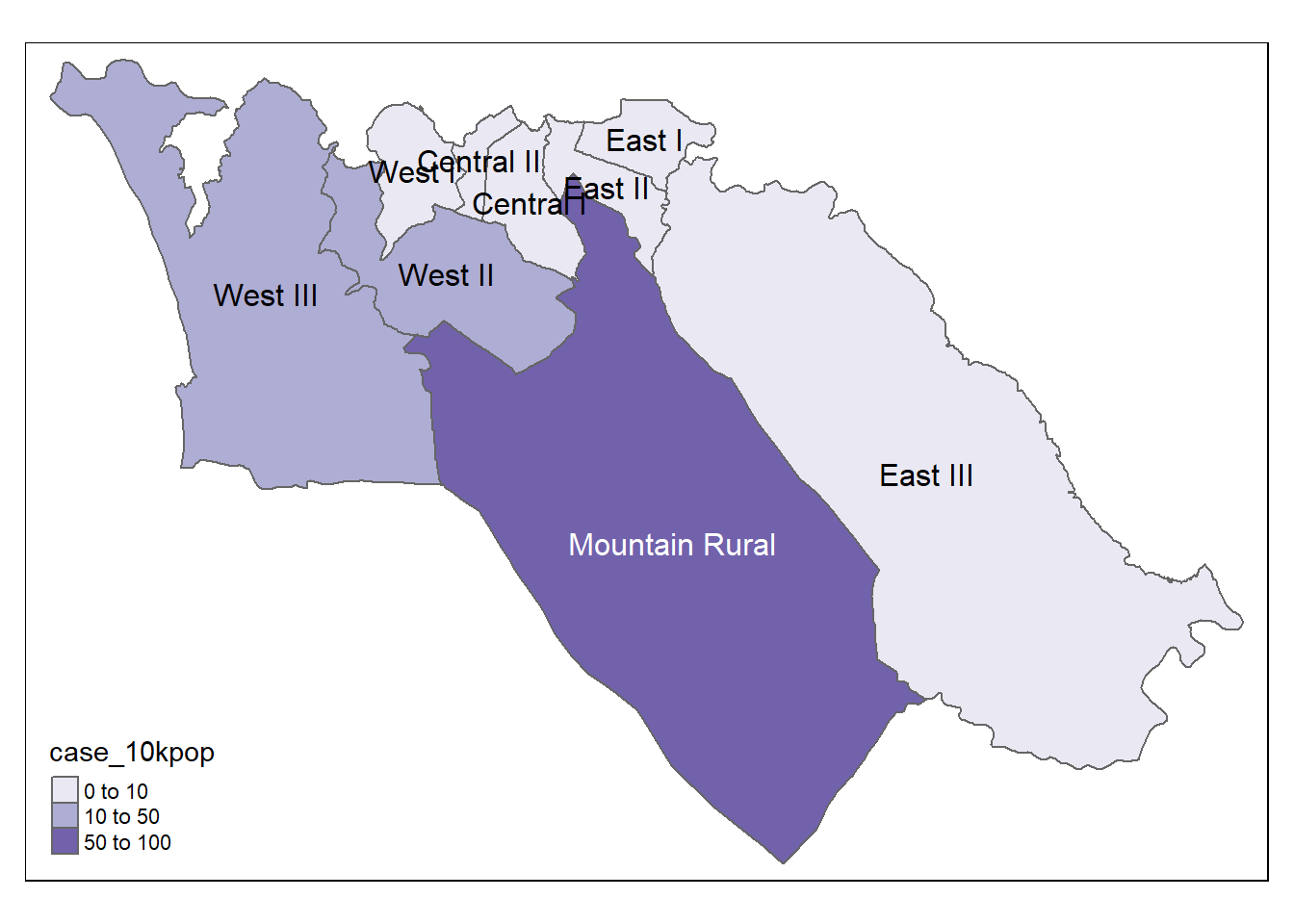
tm_text("admin3name") # afficher les nomsNous pouvons cartographier le taux d’incidence
# Cases per 10K population
tmap_mode("plot") # mode affiche statique
# plot
tm_shape(case_adm3_sf) + # visualiser lespolygons
tm_polygons("case_10kpop", # colorier en fonction du colonnes contenant le pourcentages des cas
breaks=c(0, 10, 50, 100), # définir des points de rupture pour les couleurs
palette = "Purples" # utiliser une palette de couleurs violettes
) +
tm_text("admin3name") # afficher le texte28.8 Cartographie avec ggplot2
Si vous êtes déjà familiarisé avec l’utilisation de ggplot2, vous pouvez utiliser ce package pour créer des cartes statiques de vos données. La fonction geom_sf() dessinera différents objets en fonction des caractéristiques (points, lignes ou polygones) présentes dans vos données. Par exemple, vous pouvez utiliser geom_sf() dans un ggplot() utilisant des données sf avec une géométrie polygonale pour créer une carte choroplèthe.
Pour illustrer comment cela fonctionne, nous pouvons commencer avec le fichier de forme ADM3 polygones que nous avons utilisé plus tôt. Rappelez-vous qu’il s’agit des régions de niveau administratif 3 de la Sierra Leone :
sle_adm3Simple feature collection with 12 features and 19 fields
Geometry type: MULTIPOLYGON
Dimension: XY
Bounding box: xmin: -13.29894 ymin: 8.094272 xmax: -12.91333 ymax: 8.499809
Geodetic CRS: WGS 84
# A tibble: 12 × 20
objectid admin3name admin3pcod admin3ref_n admin2name admin2pcod admin1name
* <dbl> <chr> <chr> <chr> <chr> <chr> <chr>
1 155 Koya Rural SL040101 Koya Rural Western A… SL0401 Western
2 156 Mountain Ru… SL040102 Mountain R… Western A… SL0401 Western
3 157 Waterloo Ru… SL040103 Waterloo R… Western A… SL0401 Western
4 158 York Rural SL040104 York Rural Western A… SL0401 Western
5 159 Central I SL040201 Central I Western A… SL0402 Western
6 160 East I SL040203 East I Western A… SL0402 Western
7 161 East II SL040204 East II Western A… SL0402 Western
8 162 Central II SL040202 Central II Western A… SL0402 Western
9 163 West III SL040208 West III Western A… SL0402 Western
10 164 West I SL040206 West I Western A… SL0402 Western
11 165 West II SL040207 West II Western A… SL0402 Western
12 167 East III SL040205 East III Western A… SL0402 Western
# ℹ 13 more variables: admin1pcod <chr>, admin0name <chr>, admin0pcod <chr>,
# date <date>, valid_on <date>, valid_to <date>, shape_leng <dbl>,
# shape_area <dbl>, rowcacode0 <chr>, rowcacode1 <chr>, rowcacode2 <chr>,
# rowcacode3 <chr>, geometry <MULTIPOLYGON [°]>Nous pouvons utiliser la fonction left_join() de dplyr pour ajouter les données que nous souhaitons mapper à l’objet shapefile. Dans ce cas, nous allons utiliser le cadre de données case_adm3 que nous avons créé plus tôt pour résumer le nombre de cas par région administrative ; cependant, nous pouvons utiliser cette même approche pour mapper n’importe quelle donnée stockée dans un cadre de données.
sle_adm3_dat <- sle_adm3 %>%
inner_join(case_adm3, by = "admin3pcod") # inner join = retenir seulement si dans les deux objets de données
select(sle_adm3_dat, admin3name.x, cases) # affiche les variables sélectionnées sur la consoleSimple feature collection with 9 features and 2 fields
Geometry type: MULTIPOLYGON
Dimension: XY
Bounding box: xmin: -13.29894 ymin: 8.384533 xmax: -13.12612 ymax: 8.499809
Geodetic CRS: WGS 84
# A tibble: 9 × 3
admin3name.x cases geometry
<chr> <int> <MULTIPOLYGON [°]>
1 Mountain Rural 291 (((-13.21496 8.474341, -13.21479 8.474289, -13.21465 8.4…
2 Central I 65 (((-13.22646 8.489716, -13.22648 8.48955, -13.22644 8.48…
3 East I 47 (((-13.2129 8.494033, -13.21076 8.494026, -13.21013 8.49…
4 East II 91 (((-13.22653 8.491883, -13.22647 8.491853, -13.22642 8.4…
5 Central II 20 (((-13.23154 8.491768, -13.23141 8.491566, -13.23144 8.4…
6 West III 240 (((-13.28529 8.497354, -13.28456 8.496497, -13.28403 8.4…
7 West I 41 (((-13.24677 8.493453, -13.24669 8.493285, -13.2464 8.49…
8 West II 180 (((-13.25698 8.485518, -13.25685 8.485501, -13.25668 8.4…
9 East III 23 (((-13.20465 8.485758, -13.20461 8.485698, -13.20449 8.4…Pour réaliser un graphique en colonnes du nombre de cas par région, en utilisant ggplot2, nous pourrions alors appeler geom_col() comme suit :
ggplot(data=sle_adm3_dat) +
geom_col(aes(x=fct_reorder(admin3name.x, cases, .desc=T), # réorganiser l'axe des x par ordre décroissant 'cases'
y=cases)) + # l'axe des y est le nombre de cas par région
theme_bw() +
labs( # définir le texte de la figure
title="Number of cases, by administrative unit",
x="Admin level 3",
y="Number of cases"
) +
guides(x=guide_axis(angle=45)) # angle des étiquettes de l'axe des x de 45 degrés pour un meilleur ajustementSi nous voulons utiliser ggplot2 pour réaliser une carte choroplèthe du nombre de cas, nous pouvons utiliser une syntaxe similaire pour appeler la fonction geom_sf() :
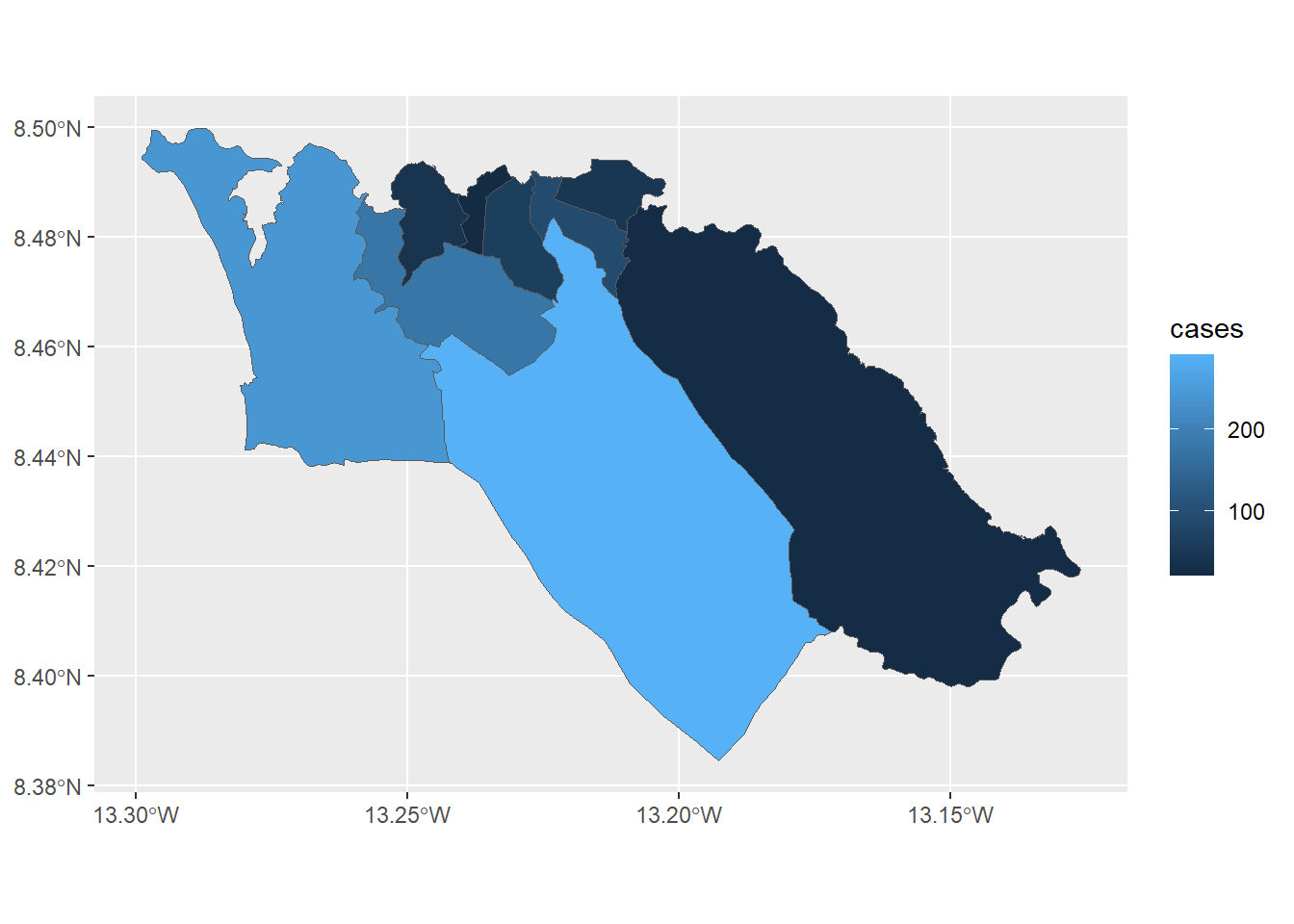
ggplot(data=sle_adm3_dat) +
geom_sf(aes(fill=cases)) # donnons une variable d'entrée à fill pour varier selon le nombre de casNous pouvons ensuite personnaliser l’apparence de notre carte en utilisant une grammaire cohérente dans ggplot2, par exemple :
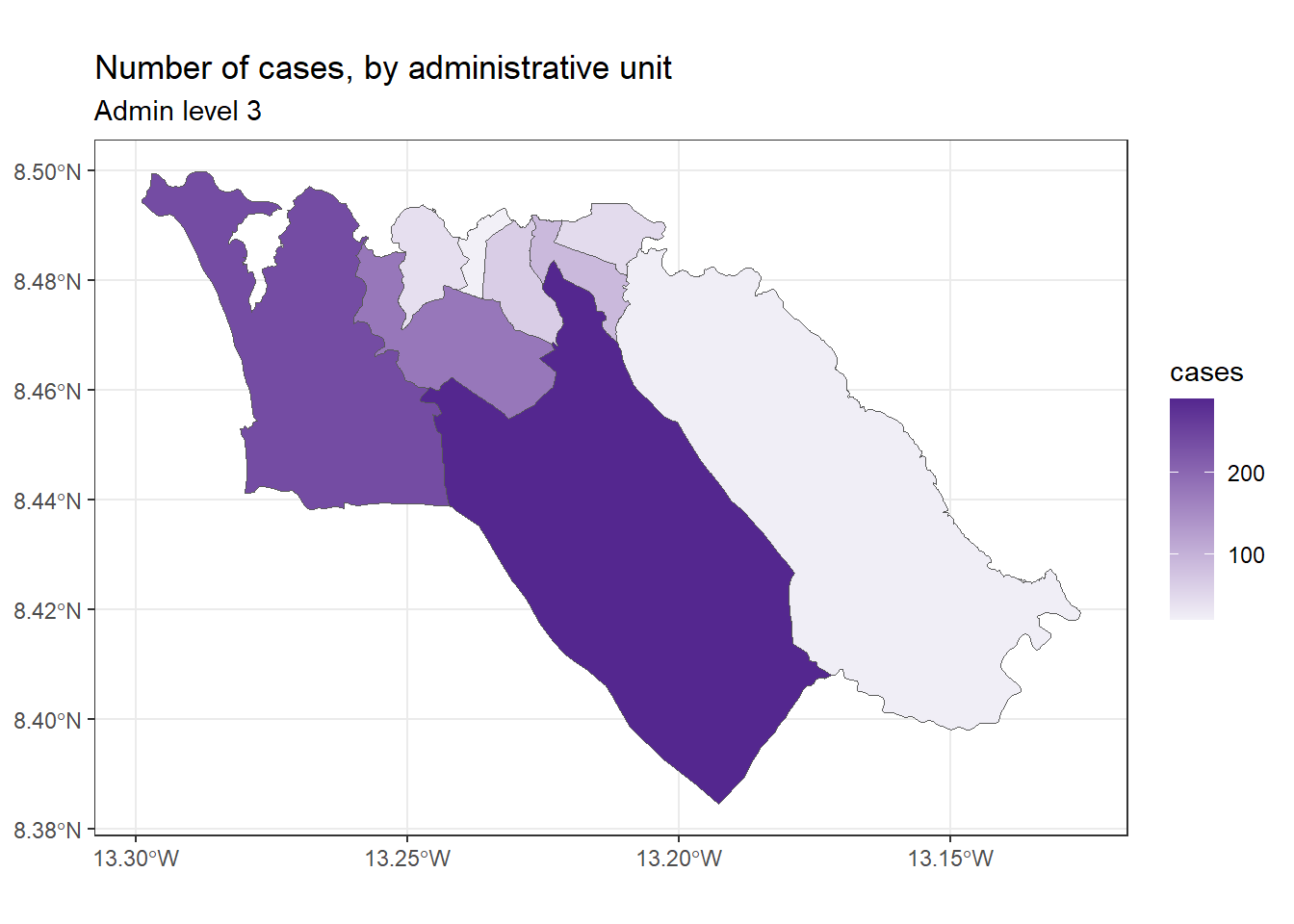
ggplot(data=sle_adm3_dat) +
geom_sf(aes(fill=cases)) +
scale_fill_continuous(high="#54278f", low="#f2f0f7") + # changeons le gradient de couleur
theme_bw() +
labs(title = "Number of cases, by administrative unit", # mettre du texte dans le graphe
subtitle = "Admin level 3"
)Pour les utilisateurs de R qui sont à l’aise avec ggplot2, geom_sf() offre une implémentation simple et directe qui convient aux visualisations cartographiques de base. Pour en savoir plus, lisez la vignette geom_sf() ou le livre ggplot2.
28.8.1 Basemaps
OpenStreetMap
Nous décrivons ci-dessous comment obtenir un plan de base pour une carte ggplot2 en utilisant les fonctionnalités d’OpenStreetMap. Les méthodes alternatives incluent l’utilisation de ggmap qui nécessite un enregistrement gratuit auprès de Google (détails).
OpenStreetMap est un projet collaboratif visant à créer une carte du monde librement modifiable. Les données de géolocalisation sous-jacentes (par exemple, l’emplacement des villes, des routes, des caractéristiques naturelles, des aéroports, des écoles, des hôpitaux, des routes, etc.) sont considérées comme le principal résultat du projet.
Tout d’abord, nous chargeons le paquet OpenStreetMap, à partir duquel nous allons obtenir notre carte de base.
Ensuite, nous créons l’objet map, que nous définissons en utilisant la fonction openmap() du paquet OpenStreetMap (documentation). Nous fournissons les éléments suivants :
-
upperLeftetlowerRightDeux paires de coordonnées spécifiant les limites du carreau de la carte de base.
- Dans ce cas, nous avons mis les valeurs max et min des lignes de la linelist, afin que la carte réponde dynamiquement aux données.
-
zoom =(si null il est déterminé automatiquement)
-
type =quel type de carte de base - nous avons listé plusieurs possibilités ici et le code utilise actuellement la première ([1]) “osm”.
-
mergeTiles =nous avons choisi TRUE pour que les tuiles de base soient toutes fusionnées en une seule.
# charger package
pacman::p_load(OpenStreetMap)
# Ajustez la carte de base par le couple de coordonnées lat/long. Choisir le type de tuile
map <- openmap(
upperLeft = c(max(linelist$lat, na.rm=T), max(linelist$lon, na.rm=T)), # limits of basemap tile
lowerRight = c(min(linelist$lat, na.rm=T), min(linelist$lon, na.rm=T)),
zoom = NULL,
type = c("osm", "stamen-toner", "stamen-terrain", "stamen-watercolor", "esri","esri-topo")[1])Si nous traçons cette carte de base maintenant, en utilisant autoplot.OpenStreetMap() du paquet OpenStreetMap, vous voyez que les unités sur les axes ne sont pas des coordonnées de latitude/longitude. Il utilise un système de coordonnées différent. Pour afficher correctement les résidences du cas (qui sont stockées en lat/long), cela doit être changé.
Ainsi, nous voulons convertir la carte en latitude/longitude avec la fonction openproj() du paquet OpenStreetMap. Nous fournissons la carte de base map et nous fournissons également le système de référence de coordonnées (CRS) que nous voulons. Nous le faisons en fournissant la chaîne de caractères “proj.4” pour la projection WGS 1984, mais vous pouvez également fournir le CRS d’autres manières. (voir cette page pour mieux comprendre ce qu’est une chaîne proj.4)
# Projection WGS84
map_latlon <- openproj(map, projection = "+proj=longlat +ellps=WGS84 +datum=WGS84 +no_defs")Maintenant, lorsque nous créons le tracé, nous voyons que les axes contiennent des coordonnées de latitude et de longitude. Le système de coordonnées a été converti. Maintenant, nos cas seront tracés correctement s’ils sont superposés !
# la representation graphique d'une carte doit se faire en utilisant "autoplot" afin de pouvoir travailler avec ggplot.
autoplot.OpenStreetMap(map_latlon)Consultez les tutoriels ici et ici pour plus d’informations.
28.9 Cartes thermiques de densité avec contours
Nous décrivons ci-dessous comment réaliser une carte thermique de densité des cas, sur une carte de base, en commençant par une liste de lignes (une ligne par cas).
- Créer une tuile basemap à partir d’OpenStreetMap, comme décrit ci-dessus.
- Tracez les cas de
linelisten utilisant les colonnes de latitude et de longitude.
- Convertir les points en une carte thermique de densité avec
stat_density_2d()de ggplot2,
Lorsque nous avons une carte de base avec des coordonnées de latitude et de longitude, nous pouvons tracer nos cas par-dessus en utilisant les coordonnées de latitude et de longitude de leur résidence.
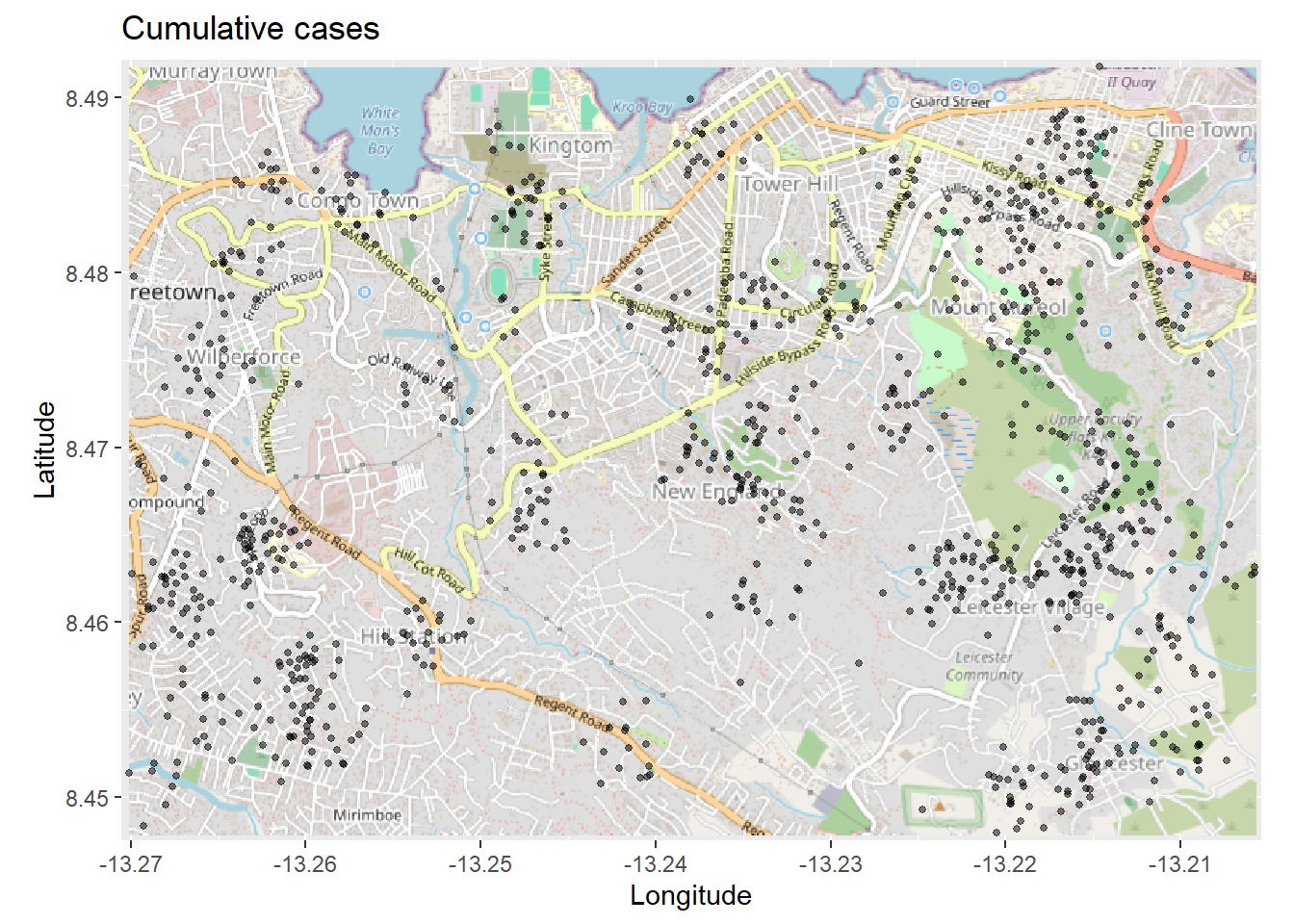
En s’appuyant sur la fonction autoplot.OpenStreetMap() pour créer la carte de base, les fonctions de ggplot2 s’ajouteront facilement par-dessus, comme le montre geom_point() ci-dessous :
## la representation graphique d'une carte doit se faire en utilisant "autoplot" afin de pouvoir travailler avec ggplot
autoplot.OpenStreetMap(map_latlon)+ # commercer avec basemap
geom_point( # ajouter des points xy à partir des colonnes lon et lat de la linelist
data = linelist,
aes(x = lon, y = lat),
size = 1,
alpha = 0.5,
show.legend = FALSE) + # abandonner entièrement la légende
labs(x = "Longitude", # titres et etiquettes
y = "Latitude",
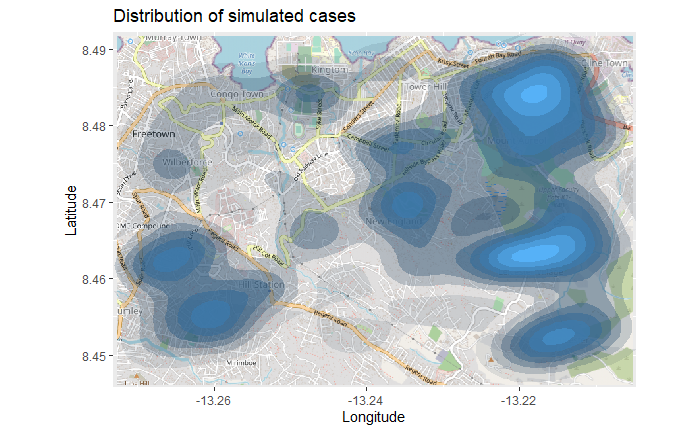
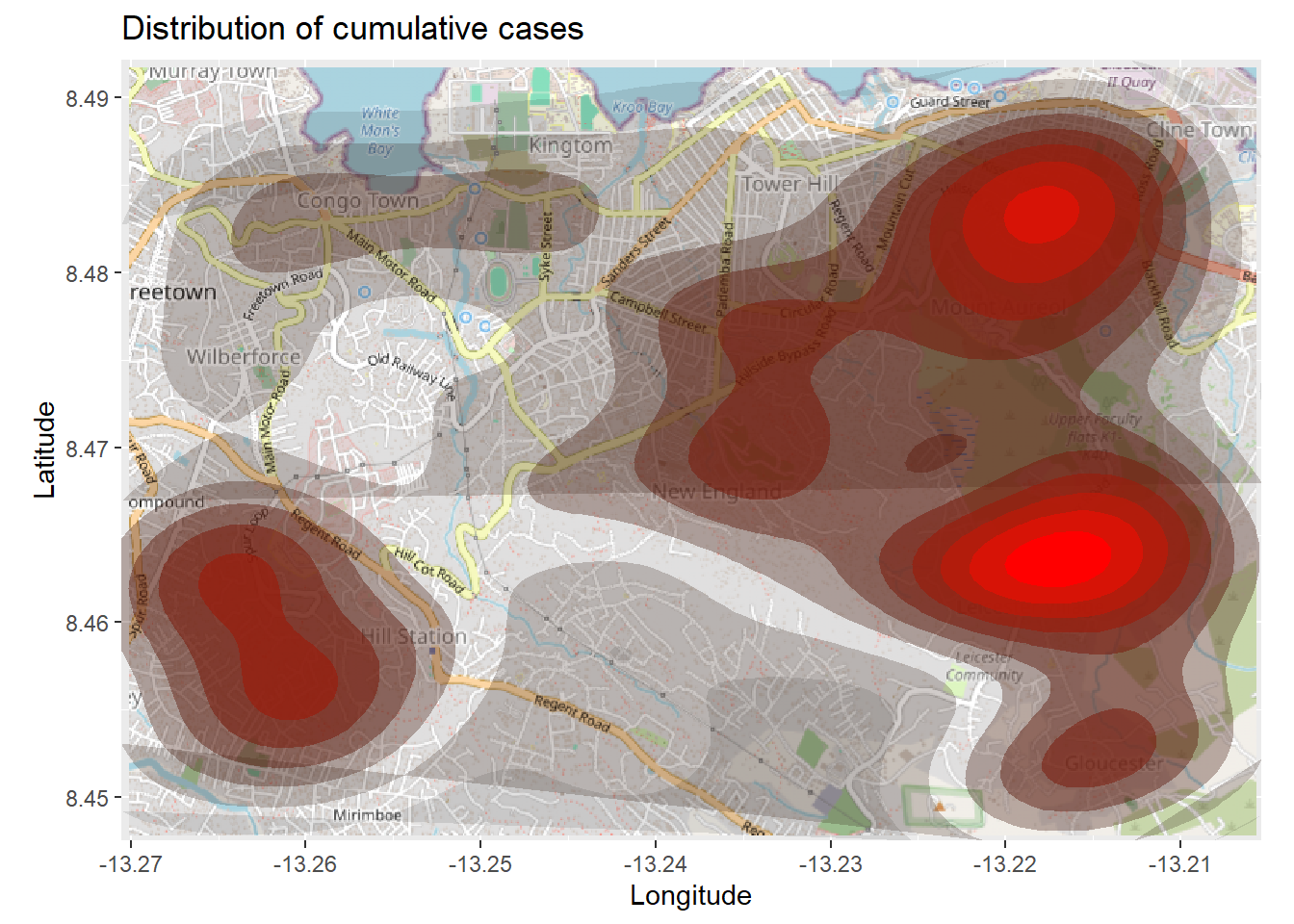
title = "Cumulative cases")La carte ci-dessus peut être difficile à interpréter, surtout si les points se chevauchent. Vous pouvez donc tracer une carte de densité en 2d en utilisant la fonction ggplot2 stat_density_2d(). Vous utilisez toujours les coordonnées lat/lon de la linelist, mais une estimation de densité à noyau 2D est effectuée et les résultats sont affichés avec des lignes de contour - comme une carte topographique. Lisez la documentation complète ici.
# commencer par la carte de base
autoplot.OpenStreetMap(map_latlon)+
# ajouter le graphique de densité
ggplot2::stat_density_2d(
data = linelist,
aes(
x = lon,
y = lat,
fill = ..level..,
alpha = ..level..),
bins = 10,
geom = "polygon",
contour_var = "count",
show.legend = F) +
# spécifier l'échelle de couleurs
scale_fill_gradient(low = "black", high = "red")+
# etiquettes
labs(x = "Longitude",
y = "Latitude",
title = "Distribution of cumulative cases")Carte thermique des séries chronologiques
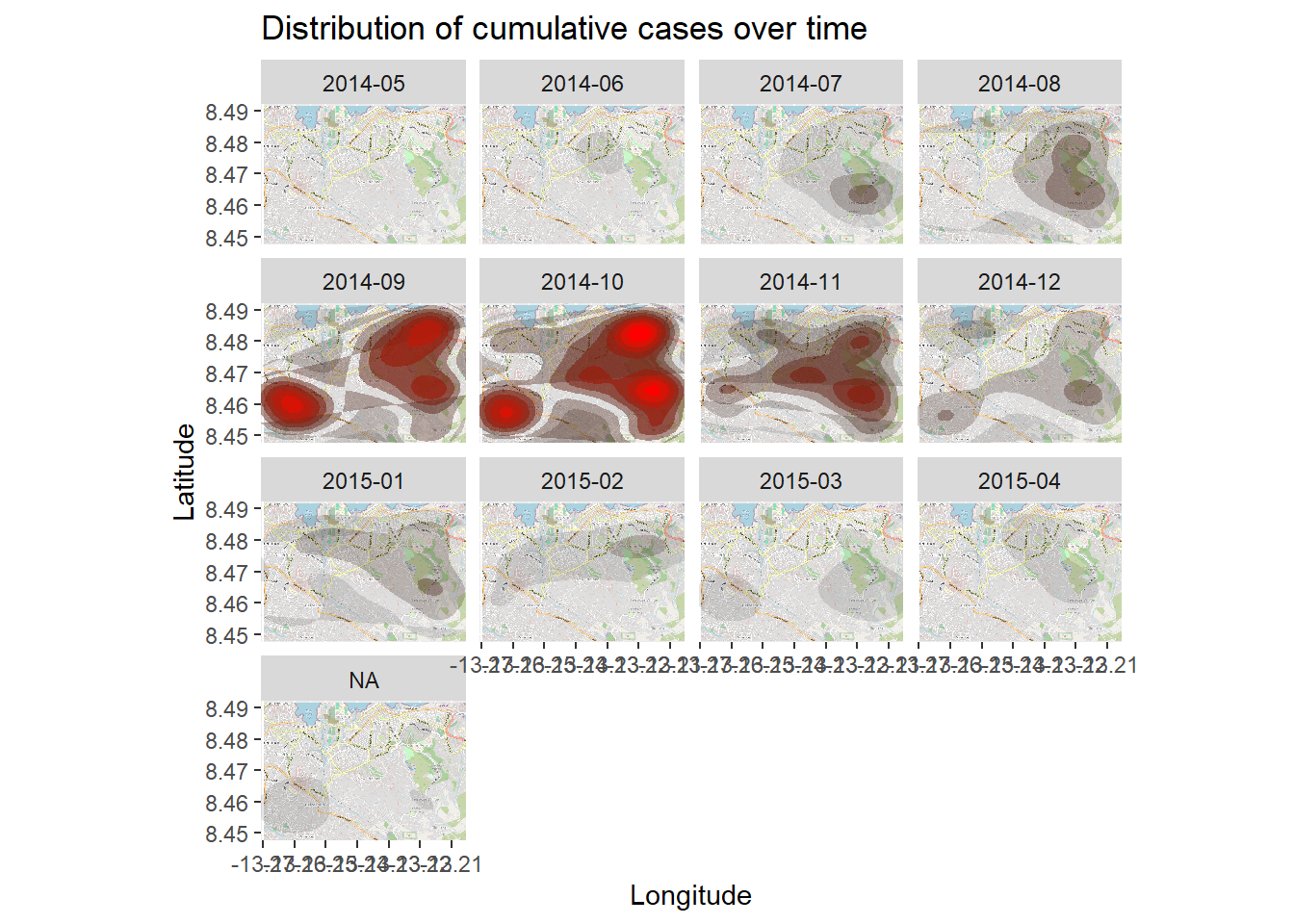
La carte thermique de densité ci-dessus montre les cas cumulés. Nous pouvons examiner l’épidémie dans le temps et l’espace en facettant la carte de chaleur en fonction du mois d’apparition des symptômes, tel qu’il est dérivé de la liste des lignes.
Nous commençons dans la linelist, en créant une nouvelle colonne avec l’année et le mois d’apparition des symptômes. La fonction format() de base R change la façon dont une date est affichée. Dans ce cas, nous voulons “YYYY-MM”.
# Extrait mois de l'apparition
linelist <- linelist %>%
mutate(date_onset_ym = format(date_onset, "%Y-%m"))
# Examiner les valeurs
table(linelist$date_onset_ym, useNA = "always")
2014-05 2014-06 2014-07 2014-08 2014-09 2014-10 2014-11 2014-12 2015-01 2015-02
9 15 42 86 179 208 128 93 77 44
2015-03 2015-04 <NA>
47 29 43 Maintenant, nous introduisons simplement le facettage via ggplot2 à la carte thermique de densité. facet_wrap() est appliqué, en utilisant les nouvelles colonnes comme lignes. Nous avons fixé le nombre de colonnes de facettes à 3 pour plus de clarté.
# packages
pacman::p_load(OpenStreetMap, tidyverse)
# commencer avec basemap
autoplot.OpenStreetMap(map_latlon)+
# ajouter le graphique de densité
ggplot2::stat_density_2d(
data = linelist,
aes(
x = lon,
y = lat,
fill = ..level..,
alpha = ..level..),
bins = 10,
geom = "polygon",
contour_var = "count",
show.legend = F) +
# spécifier l'échelle de couleurs
scale_fill_gradient(low = "black", high = "red")+
# etiquettes
labs(x = "Longitude",
y = "Latitude",
title = "Distribution of cumulative cases over time")+
# facetter le graphique par mois-année de début d'activité
facet_wrap(~ date_onset_ym, ncol = 4) 28.10 Statistiques spatiales
Jusqu’à présent, la plupart de nos discussions ont porté sur la visualisation des données spatiales. Dans certains cas, vous pouvez également être intéressé par l’utilisation des statistiques spatiales pour mesurer la force des relations spatiales des attributs dans vos données. Cette section donne un bref aperçu de certains concepts clés des statistiques spatiales et suggère quelques ressources utiles à explorer si vous souhaitez effectuer des analyses spatiales plus poussées.
Les relations spatiales
Avant de pouvoir calculer toute statistique spatiale, nous devons spécifier les relations entre les objets de nos données. Il existe de nombreuses façons de conceptualiser les relations spatiales, mais un modèle simple et couramment appliqué est celui de la adjacence - plus précisément, nous nous attendons à une relation géographique entre les zones qui partagent une frontière ou qui sont “voisines” les unes des autres.
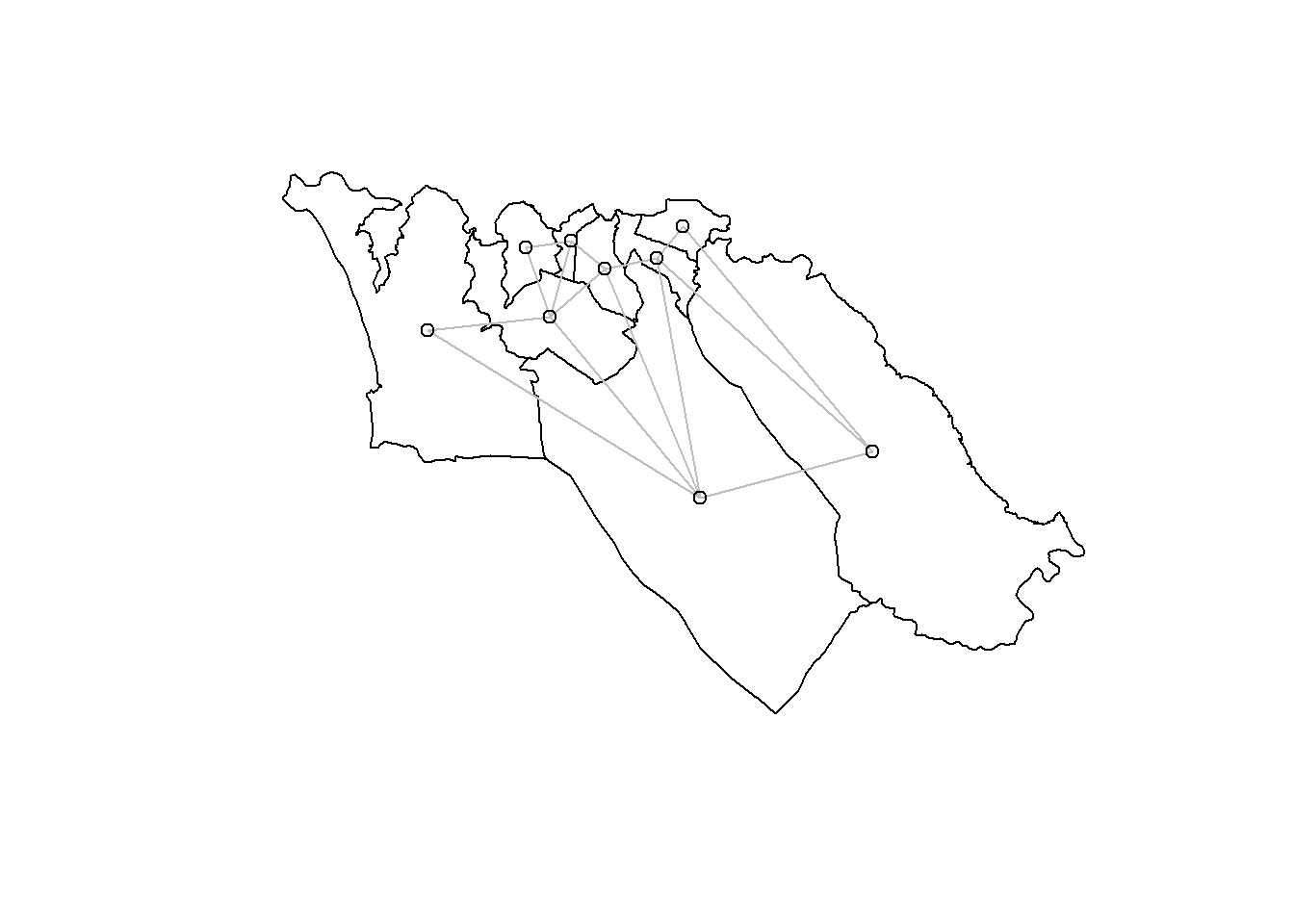
Nous pouvons quantifier les relations d’adjencence entre les polygones des régions administratives dans les données sle_adm3 que nous avons utilisées avec le package spdep. Nous allons spécifier une contiguïté queen, ce qui signifie que les voisins possèdent au moins un segment de frontière commune.les voisins possèdent au moins un segment de frontière commune Dans notre cas, avec des polygones irréguliers, la distinction est triviale, mais dans certains cas, le choix entre la reine et la tour peut ne pas etre evident
sle_nb <- spdep::poly2nb(sle_adm3_dat, queen=T) #Extraction de la liste des voisins
sle_adjmat <- spdep::nb2mat(sle_nb) # créer une matrice résumant les relations entre voisins
sle_listw <- spdep::nb2listw(sle_nb) # créer l'objet listw (liste de poids) -- nous en aurons besoin plus tard
sle_nbNeighbour list object:
Number of regions: 9
Number of nonzero links: 30
Percentage nonzero weights: 37.03704
Average number of links: 3.333333 round(sle_adjmat, digits = 2) [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9]
1 0.00 0.20 0.00 0.20 0.00 0.2 0.00 0.20 0.20
2 0.25 0.00 0.00 0.25 0.25 0.0 0.00 0.25 0.00
3 0.00 0.00 0.00 0.50 0.00 0.0 0.00 0.00 0.50
4 0.25 0.25 0.25 0.00 0.00 0.0 0.00 0.00 0.25
5 0.00 0.33 0.00 0.00 0.00 0.0 0.33 0.33 0.00
6 0.50 0.00 0.00 0.00 0.00 0.0 0.00 0.50 0.00
7 0.00 0.00 0.00 0.00 0.50 0.0 0.00 0.50 0.00
8 0.20 0.20 0.00 0.00 0.20 0.2 0.20 0.00 0.00
9 0.33 0.00 0.33 0.33 0.00 0.0 0.00 0.00 0.00
attr(,"call")
spdep::nb2mat(neighbours = sle_nb)La matrice affiché ci-dessus montre les relations entre les 9 régions de nos données sle_adm3. Un score de 0 indique que deux régions ne sont pas voisines, tandis que toute valeur différente de 0 indique une relation de voisinage. Les valeurs de la matrice sont normalisées afin que chaque région ait un poids total par ligne egale à 1.
Une meilleure façon de faire ressortir ces relations de voisinage est de les reprensenter graphiquement :
plot(sle_adm3_dat$geometry) + # visualiser les limites de la region
spdep::plot.nb(sle_nb,as(sle_adm3_dat, 'Spatial'), col='grey', add=T) # ajout des relation de voisinageNous avons utilisé une approche d’adjacence pour identifier les polygones voisins ; les voisins que nous avons identifiés sont aussi parfois appelés voisins basés sur la contiguïté. Mais ce n’est qu’une façon de choisir les régions qui sont censées avoir une relation géographique. Les approches alternatives les plus courantes pour identifier les relations géographiques génèrent des voisins basés sur la distance ; brièvement, il s’agit de :
K-plus proches voisins - Sur la base de la distance entre les centroïdes (le centre géographiquement pondéré de chaque région polygonale), sélectionnez les n régions les plus proches comme voisines. Un seuil de proximité de distance maximale peut également être fixé. Dans spdep, vous pouvez utiliser
knearneigh()(voir documentation).Distance threshold neighbors - Sélectionne les voisins sur la base d’une un seuil de distance definie. Dans spdep, ces relations de voisinage peuvent être identifiées en utilisant
dnearneigh()(voir documentation).
Autocorrélation spatiale
La première loi de la géographie de Tobler, souvent citée, stipule que “Tout interagit avec tout, mais deux objets proches ont plus de chance de le faire que deux objets éloignés”. En épidémiologie, cela signifie souvent que la probabilité d’obtenir un résultat sanitaire particulier dans une région donnée est plus liée au resultats des régions voisines qu’à celui des régions éloignées. Ce concept a été formalisé sous le nom d’autocorrélation spatiale - la propriété statistique selon laquelle les objets spatiaux ayant des valeurs similaires sont localisées dans l’espace. Les mesures statistiques de l’autocorrélation spatiale peuvent être utilisées pour quantifier la dépendance spatiale dans vos données, localiser l’endroit où ont trouve des patterns, et identifier les patterns d’autocorrélation spatiale entre des variables distinctes dans vos données. Cette section donne un aperçu de certaines mesures courantes d’autocorrélation spatiale et de la façon de les calculer dans R.
I de Moran - Il s’agit d’une statistique globale de synthèse de la corrélation entre la valeur d’une variable dans une région et les valeurs de cette même variable dans les régions voisines. La statistique I de Moran est généralement comprise entre -1 et 1. Une valeur de 0 n’indique aucun modèle de corrélation spatiale, tandis que des valeurs plus proches de 1 ou de -1 indiquent une autocorrélation spatiale plus forte (valeurs similaires proches) ou une dispersion spatiale (valeurs dissemblables proches), respectivement.
Par exemple, nous allons calculer une statistique I de Moran pour quantifier l’autocorrélation spatiale dans les cas d’Ebola que nous avons cartographiés plus tôt (rappelez-vous, il s’agit d’un sous-ensemble de cas provenant du cadre de données de la linelist épidémique simulée). Le paquet spdep possède une fonction, moran.test, qui peut faire ce calcul pour nous :
moran_i <-spdep::moran.test(sle_adm3_dat$cases, # vecteur numérique avec la variable d'intérêt
listw=sle_listw) # listw object résumant les relations de voisinage
moran_i # print les résultats du test I de Moran
Moran I test under randomisation
data: sle_adm3_dat$cases
weights: sle_listw
Moran I statistic standard deviate = 1.8096, p-value = 0.03518
alternative hypothesis: greater
sample estimates:
Moran I statistic Expectation Variance
0.25087348 -0.12500000 0.04314513 La sortie de la fonction moran.test() nous montre une statistique de Moran I de round(moran_i$estimate[1],2). Cela indique la présence d’une autocorrélation spatiale dans nos données - plus précisément, que les régions présentant un nombre similaire de cas d’Ebola sont susceptibles d’être proches les unes des autres. La valeur p fournie par moran.test() est générée par comparaison avec l’espérance sous l’hypothèse nulle d’absence d’autocorrélation spatiale, et peut être utilisée si vous avez besoin de rapporter les résultats d’un test d’hypothèse formel.
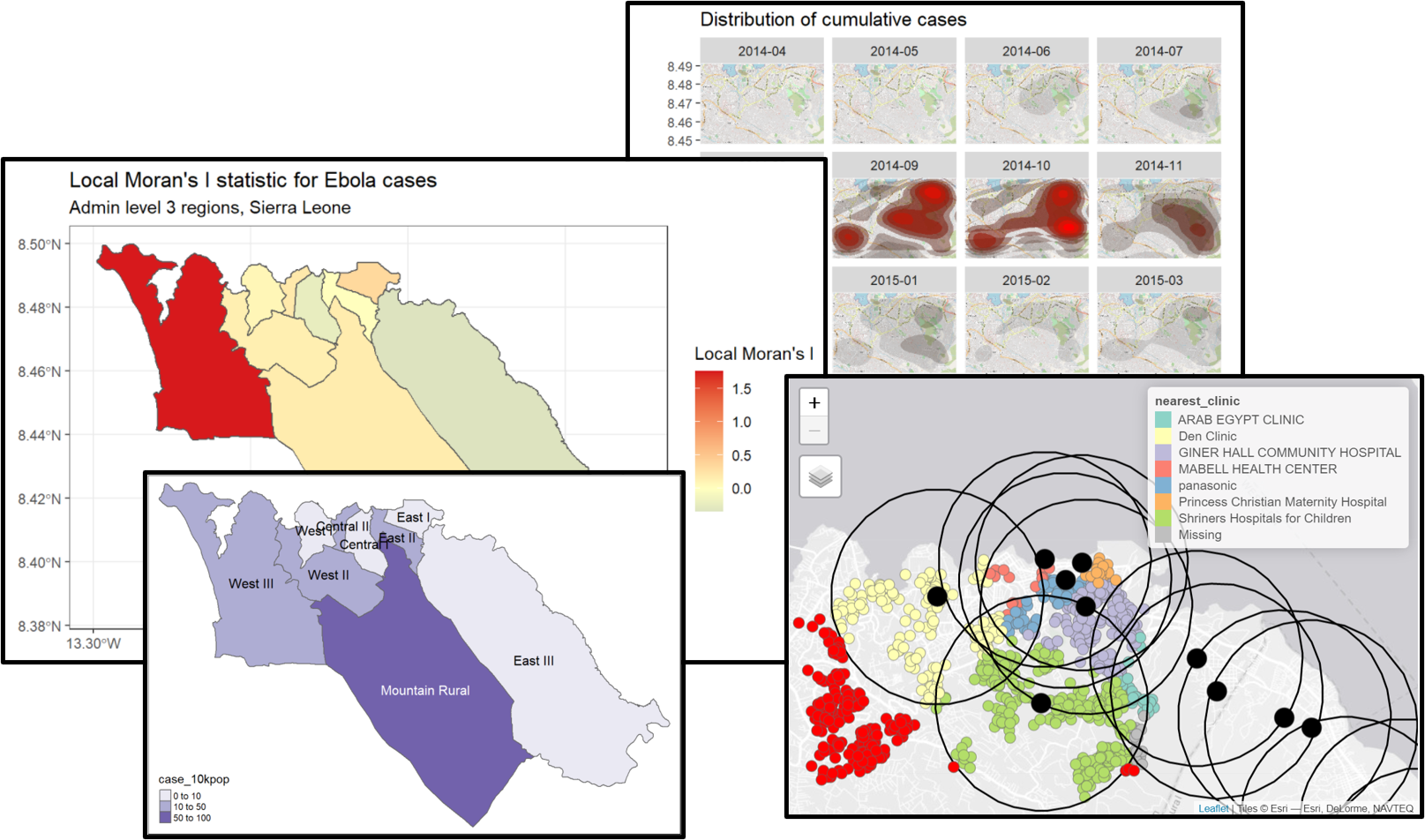
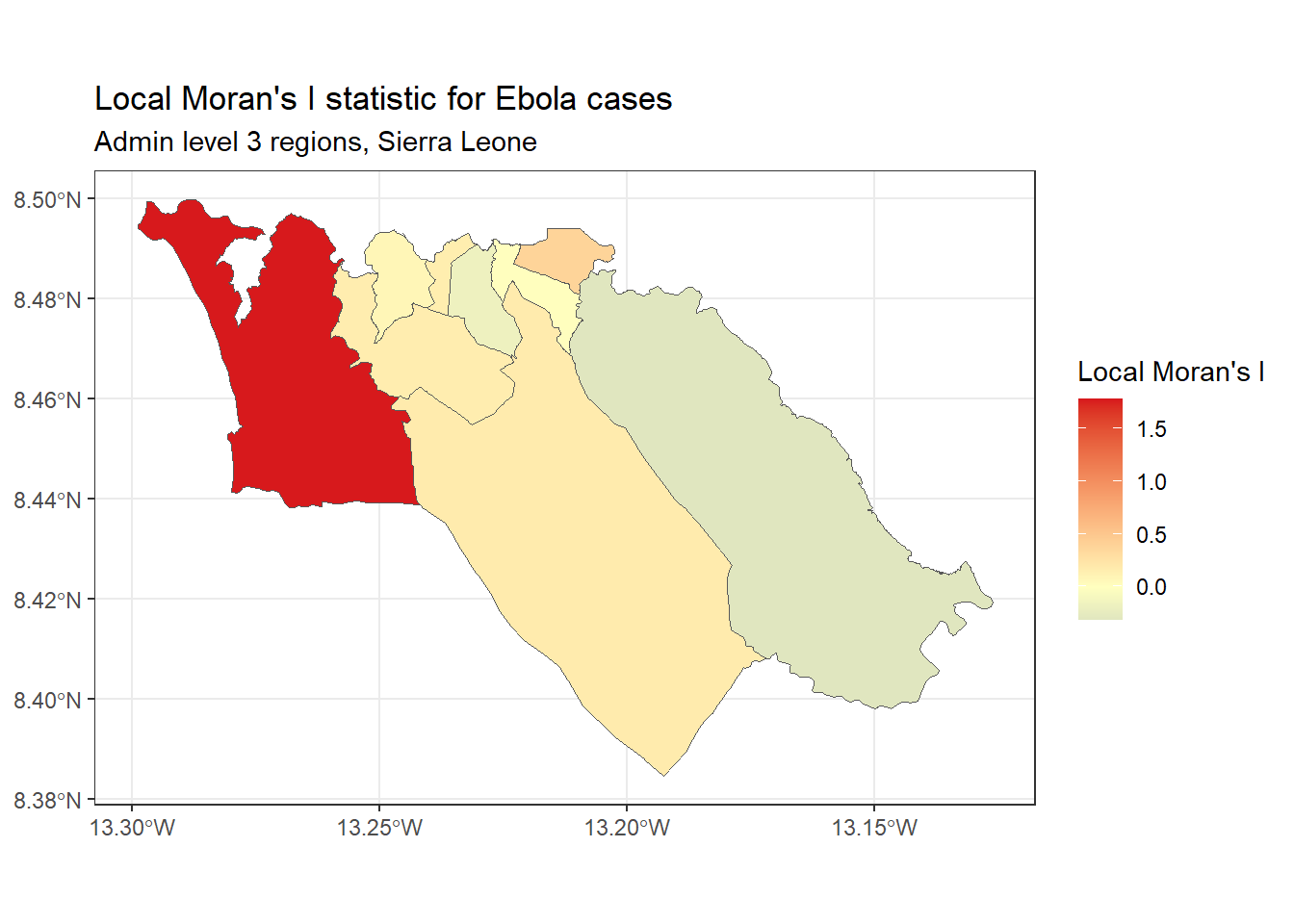
Local Moran’s I - Nous pouvons décomposer la statistique (globale) de Moran I calculée ci-dessus pour identifier l’autocorrélation spatiale localisée, c’est-à-dire pour identifier des groupes spécifiques dans nos données. Cette statistique, qui est parfois appelée Indicateur local d’association spatiale (LISA), résume l’étendue de l’autocorrélation spatiale autour de chaque région individuelle. Elle peut être utile pour trouver les points “chauds” et “froids” sur la carte.
Pour montrer un exemple, nous pouvons calculer et cartographier le I de Moran local pour les comptes de cas d’Ebola utilisés ci-dessus, avec la fonction local_moran() de spdep :
# calculer le I de Moran local
local_moran <- spdep::localmoran(
sle_adm3_dat$cases, # variable d'interet
listw=sle_listw # listw object avec la ponderation de voisinage
)
# joindre les results à un jeu de données sf
sle_adm3_dat<- cbind(sle_adm3_dat, local_moran)
# cartographier
ggplot(data=sle_adm3_dat) +
geom_sf(aes(fill=Ii)) +
theme_bw() +
scale_fill_gradient2(low="#2c7bb6", mid="#ffffbf", high="#d7191c",
name="Local Moran's I") +
labs(title="Local Moran's I statistic for Ebola cases",
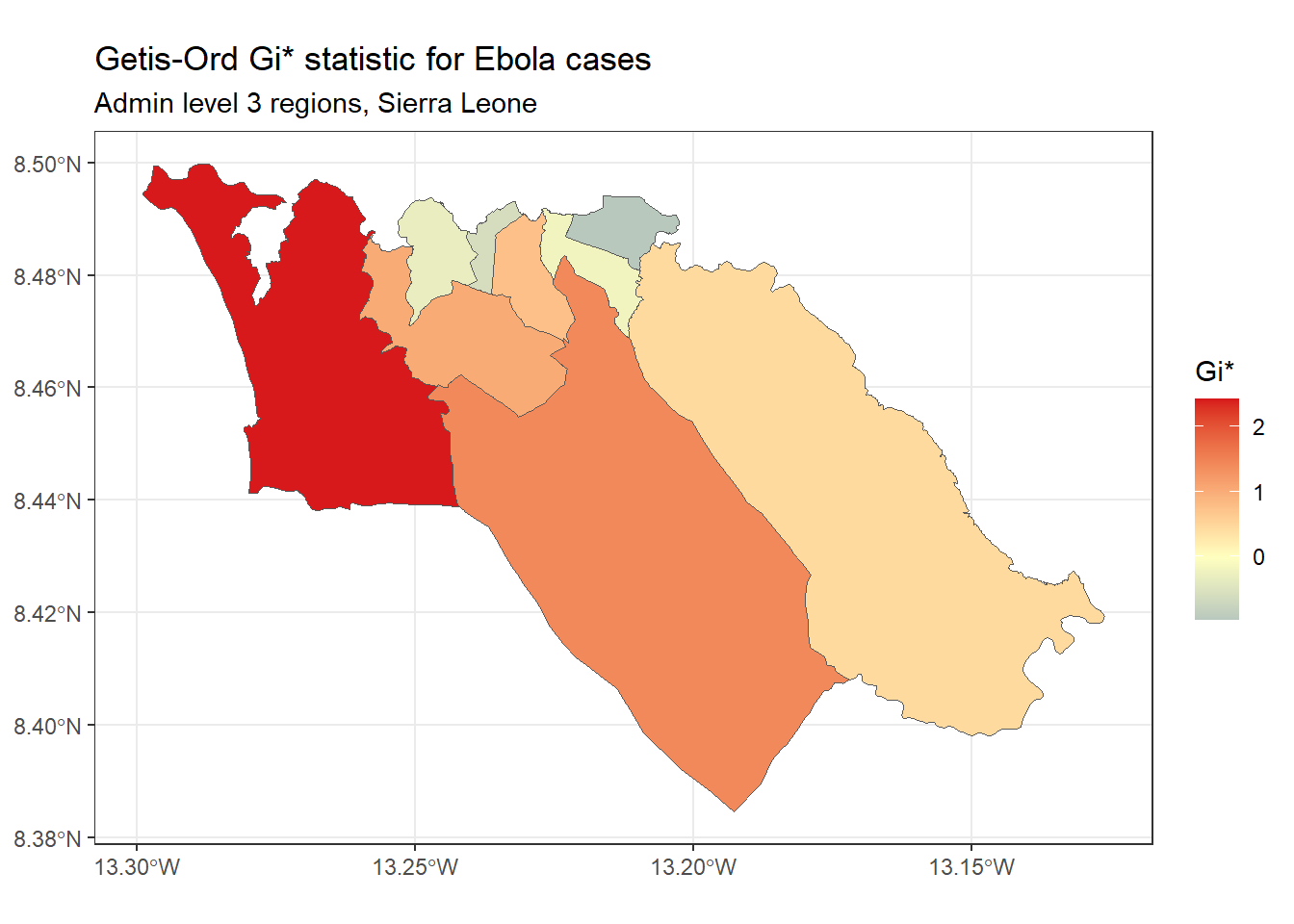
subtitle="Admin level 3 regions, Sierra Leone")Getis-Ord Gi* - Il s’agit d’une autre statistique couramment utilisée pour l’analyse des points chauds ; en grande partie, la popularité de cette statistique est liée à son utilisation dans l’outil d’analyse des points chauds d’ArcGIS. Elle est basée sur l’hypothèse que, généralement, la différence de valeur d’une variable entre des régions voisines devrait suivre une distribution normale. Elle utilise une approche de type z-score pour identifier les régions qui ont des valeurs significativement plus élevées (point chaud) ou significativement plus basses (point froid) d’une variable spécifiée, par rapport à leurs voisins.
Nous pouvons calculer et cartographier la statistique Gi* en utilisant la fonction localG() de spdep :
# Effectuer une analyse G locale
getis_ord <- spdep::localG(
sle_adm3_dat$cases,
sle_listw
)
# joindre les résultats aux données SF
sle_adm3_dat$getis_ord <- as.numeric(getis_ord)
# afficher la carte
ggplot(data=sle_adm3_dat) +
geom_sf(aes(fill=getis_ord)) +
theme_bw() +
scale_fill_gradient2(low="#2c7bb6", mid="#ffffbf", high="#d7191c",
name="Gi*") +
labs(title="Getis-Ord Gi* statistic for Ebola cases",
subtitle="Admin level 3 regions, Sierra Leone")Comme vous pouvez le constater, la carte des Getis-Ord Gi* est légèrement différente de la carte des Moran locaux que j’ai produite précédemment. Cela reflète le fait que la méthode utilisée pour calculer ces deux statistiques est légèrement différente ; celle que vous devez utiliser dépend de votre cas d’utilisation spécifique et de la question de recherche qui vous intéresse.
Test L de Lee - Il s’agit d’un test statistique de corrélation spatiale bivariée. Il vous permet de vérifier si la configuration spatiale d’une variable donnée x est similaire à la configuration spatiale d’une autre variable, y, dont on suppose qu’elle est liée spatialement à x.
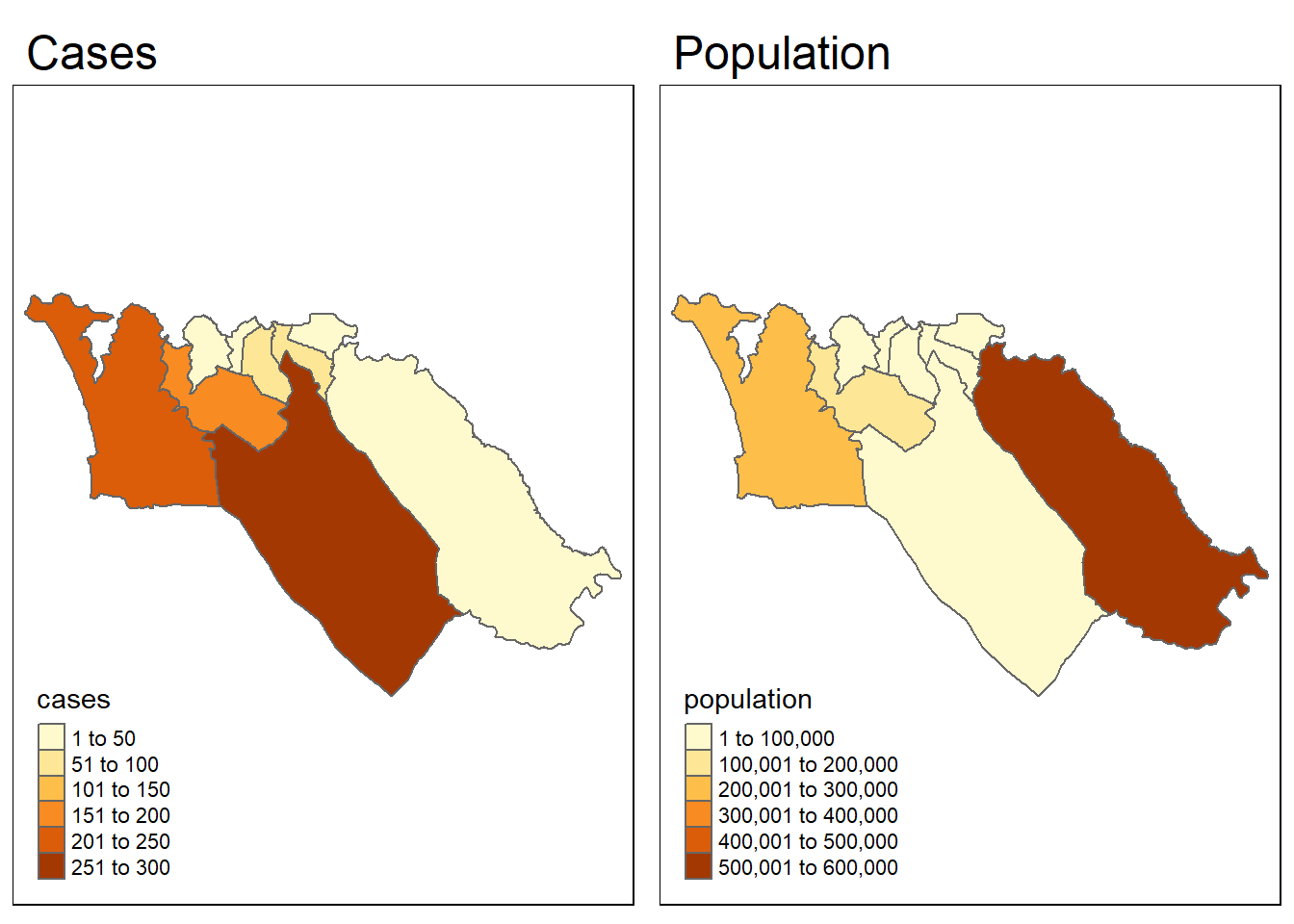
Pour donner un exemple, testons si la configuration spatiale des cas d’Ebola de l’épidémie simulée est corrélée à la configuration spatiale de la population. Pour commencer, nous devons avoir une variable population dans nos données sle_adm3. Nous pouvons utiliser la variable total du dataframe sle_adm3_pop que nous avons chargé précédemment.
sle_adm3_dat <- sle_adm3_dat %>%
rename(population = total) # renommer 'total' en 'population'Nous pouvons rapidement visualiser les configurations spatiales des deux variables côte à côte, pour voir si elles se ressemblent :
tmap_mode("plot")
cases_map <- tm_shape(sle_adm3_dat) + tm_polygons("cases") + tm_layout(main.title="Cases")
pop_map <- tm_shape(sle_adm3_dat) + tm_polygons("population") + tm_layout(main.title="Population")
tmap_arrange(cases_map, pop_map, ncol=2) # arranger en facettes 2x1Visuellement, les modèles semblent dissemblables. Nous pouvons utiliser la fonction lee.test() de spdep pour tester statistiquement si le modèle d’autocorrélation spatiale des deux variables est lié. La statistique L sera proche de 0 s’il n’y a pas de corrélation entre les modèles, proche de 1 s’il y a une forte corrélation positive (c’est-à-dire que les modèles sont similaires), et proche de -1 s’il y a une forte corrélation négative (c’est-à-dire que les modèles sont inverses).
lee_test <- spdep::lee.test(
x=sle_adm3_dat$cases, # variable 1 à comparer
y=sle_adm3_dat$population, # variable 2 à comparer
listw=sle_listw # listw objet avec les poids des voisins
)
lee_test
Lee's L statistic randomisation
data: sle_adm3_dat$cases , sle_adm3_dat$population
weights: sle_listw
Lee's L statistic standard deviate = -1.0406, p-value = 0.851
alternative hypothesis: greater
sample estimates:
Lee's L statistic Expectation Variance
-0.15123600 -0.03567982 0.01233238 Le résultat ci-dessus montre que la statistique L de Lee pour nos deux variables était round(lee_test$estimate[1],2), ce qui indique une faible corrélation négative. Cela confirme notre évaluation visuelle selon laquelle le schéma des cas et la population ne sont pas liés l’un à l’autre, et fournit la preuve que le schéma spatial des cas n’est pas strictement le résultat de la densité de population dans les zones à haut risque.
La statistique de Lee L peut être utile pour faire ce genre d’inférences sur la relation entre des variables distribuées dans l’espace ; cependant, pour décrire la nature de la relation entre deux variables de manière plus détaillée, ou pour ajuster les facteurs de confusion, des techniques de régression spatiale seront nécessaires. Celles-ci sont décrites brièvement dans la section suivante.
Régression spatiale
Vous pouvez souhaiter faire des inférences statistiques sur les relations entre les variables de vos données spatiales. Dans ce cas, il est utile d’envisager des techniques de régression spatiale, c’est-à-dire des approches de la régression qui prennent explicitement en compte l’organisation spatiale des unités dans vos données. Voici quelques raisons pour lesquelles vous pouvez avoir besoin d’envisager des modèles de régression spatiale, plutôt que des modèles de régression standard tels que les GLM :
Les modèles de régression standard supposent que les résidus sont indépendants les uns des autres. En présence d’une forte autocorrélation spatiale, les résidus d’un modèle de régression standard sont susceptibles d’être également autocorrélés dans l’espace, violant ainsi cette hypothèse. Cela peut entraîner des problèmes d’interprétation des résultats du modèle, auquel cas un modèle spatial serait préférable.
Les modèles de régression supposent aussi généralement que l’effet d’une variable x est constant sur toutes les observations. Dans le cas d’une hétérogénéité spatiale, les effets que nous souhaitons estimer peuvent varier dans l’espace, et nous pouvons être intéressés par la quantification de ces différences. Dans ce cas, les modèles de régression spatiale offrent plus de flexibilité pour estimer et interpréter les effets.
Les détails des approches de régression spatiale dépassent le cadre de ce manuel. Cette section donnera plutôt un aperçu des modèles de régression spatiale les plus courants et de leurs utilisations, et vous renverra à des références qui pourront vous être utiles si vous souhaitez approfondir ce domaine.
Modèles d’erreurs spatiales - Ces modèles supposent que les termes d’erreur entre les unités spatiales sont corrélés, auquel cas les données violeraient les hypothèses d’un modèle MCO standard. Les modèles d’erreur spatiaux sont aussi parfois appelés modèles autorégressifs simultanés (SAR). Ils peuvent être ajustés en utilisant la fonction errorsarlm() du paquet spatialreg (fonctions de régression spatiale qui faisaient autrefois partie de spdep).
Modèles de décalage spatial - Ces modèles supposent que la variable dépendante pour une région i est influencée non seulement par la valeur des variables indépendantes dans i, mais aussi par les valeurs de ces variables dans les régions voisines de i. Comme les modèles à erreurs spatiales, les modèles à décalage spatial sont aussi parfois décrits comme des modèles autorégressifs simultanés (SAR). Ils peuvent être ajustés en utilisant la fonction lagsarlm() du paquet spatialreg.
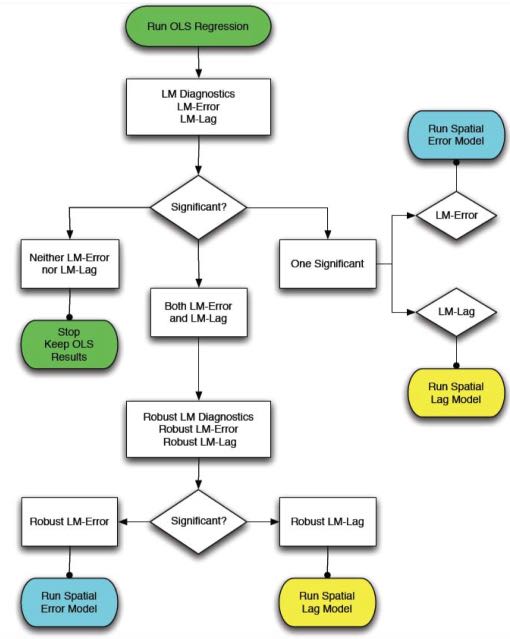
Le paquet spdep contient plusieurs tests de diagnostic utiles pour décider entre les modèles MCO standard, les modèles à décalage spatial et les modèles à erreur spatiale. Ces tests, appelés Diagnostics du multiplicateur de Lagrange, peuvent être utilisés pour identifier le type de dépendance spatiale de vos données et choisir le modèle le plus approprié. La fonction lm.LMtests() peut être utilisée pour calculer tous les tests du multiplicateur de Lagrange. Anselin (1988) fournit également un diagramme de flux utile pour décider du modèle de régression spatiale à utiliser en fonction des résultats des tests du multiplicateur de Lagrange :
Modèles hiérarchiques bayésiens - Les approches bayésiennes sont couramment utilisées pour certaines applications de l’analyse spatiale, le plus souvent pour la cartographie des maladies. Elles sont préférables dans les cas où les données de cas sont peu distribuées (par exemple, dans le cas d’un résultat rare) ou statistiquement “bruyantes”, car elles peuvent être utilisées pour générer des estimations “lissées” du risque de maladie en tenant compte du processus spatial latent sous-jacent. Cela peut améliorer la qualité des estimations. Elles permettent également à l’enquêteur de préspécifier (via le choix de l’antériorité) les modèles de corrélation spatiale complexes qui peuvent exister dans les données, ce qui peut rendre compte de la variation spatialement dépendante et indépendante des variables indépendantes et dépendantes. Dans R, les modèles hiérarchiques bayésiens peuvent être ajustés à l’aide du paquet CARbayes (voir vignette) ou de R-INLA (voir site web et manuel). R peut également être utilisé pour appeler un logiciel externe qui effectue des estimations bayésiennes, comme JAGS ou WinBUGS.
28.11 Resources
R Simple Features and sf package vignette
R tmap package vignette
Spatial Data in R (EarthLab course)
Applied Spatial Data Analysis in R textbook
SpatialEpiApp - a Shiny app that is downloadable as an R package, allowing you to provide your own data and conduct mapping, cluster analysis, and spatial statistics.
An Introduction to Spatial Econometrics in R workshop