Warning in epicontacts::make_epicontacts(linelist = linelist, contacts =
contacts, : Cycle(s) detected in the contact network: this may be unwanted37 感染連鎖
37.1 概略
感染経路や接触者の追跡データを扱い、分析し、可視化するための主要なツールは、RECON 社の人々によって開発された epicontacts パッケージです。 ノードにカーソルを合わせて詳細を表示したり、ドラッグして移動したり、クリックして下流の症例を強調するなど、以下の動的なプロットを試してみてください。
37.2 準備
パッケージを読み込み
まず、データの読み込みや操作に必要な標準パッケージを読み込みます。 このハンドブックでは、pacman パッケージの p_load() を使うことを強調しています。 p_load() は、必要に応じてパッケージをインストールし、そして使用するためにパッケージを読み込みます。R の base パッケージから library() を使用してパッケージを読み込めます。R のパッケージについての詳細は R の基礎 のページを参照してください。
pacman::p_load(
rio, # ファイルをインポート
here, # ファイルの位置
tidyverse, # データ管理 + ggplot2 作図
remotes # github からのパッケージインストール
)開発版の epicontacts パッケージが必要になります。 pacman パッケージの p_install_github() 関数を使って GitHub からインストールできます。 以下のコマンドは一度だけ実行すればよく、パッケージを使用するたびに実行する必要はありません(その後は通常通り p_load() を使用できます)。
pacman::p_install_gh("reconhub/epicontacts@timeline")データをインポート
エボラ出血熱の流行をシミュレートしたデータセットをインポートします。データをダウンロードして順を追って見たい方は、ハンドブックとデータのダウンロード章の説明をご覧ください。データは rio パッケージの import() 関数を利用してインポートしましょう。データをインポートする様々な方法については、インポートとエクスポートの章をご覧ください。
# linelist をインポート
linelist <- import("linelist_cleaned.xlsx")linelist の最初の 50 行を以下に表示します。特に注目すべきは、case_id, generation, infector, source の列です。
epicontacts オブジェクトを作成
次に、epicontacts オブジェクトを作成する必要がありますが、これには 2 種類のデータが必要です。
- 症例を文書化したラインリスト。列は変数で、行は固有の症例に対応します。
- 固有の ID に基づいて症例間のリンクを定義するエッジリスト(これらは接触、伝播イベントなどになります)。
すでに linelist があるので、症例間、特に ID 間のエッジリストを作成するだけです。 infector 列と case_id 列をリンクすることで、linelist から感染リンクを抽出できます。 この時点で、「エッジのプロパティ」を追加できます。 エッジプロパティは、症例自体ではなく、2 つの症例間のリンクを記述するあらゆる変数を意味します。 例として、感染イベントの場所を記述する location 変数と、接触の持続時間を日単位で記述する duration 変数をエッジプロパティへ追加してみます。
以下のコードでは、dplyr パッケージの transmute 関数は mutate 関数と似ていますが、関数内で指定した列のみを保持する点が異なります。 drop_na 関数は、指定された列が NA 値を持つ行をフィルタリングします。ここでは、感染者が判明している行のみを保持します。
## contacts を生成
contacts <- linelist %>%
transmute(
infector = infector,
case_id = case_id,
location = sample(c("Community", "Nosocomial"), n(), TRUE),
duration = sample.int(10, n(), TRUE)
) %>%
drop_na(infector)これで、make_epicontacts 関数を使って、epicontacts オブジェクトを作成できます。 linelist のどの列が症例の一意の識別子を指しているか、また contacts のどの列が各リンクに関係する症例の一意の識別子を指しているかを指定する必要があります。 リンクについては、感染が感染者から(from)症例へと(to)向かうという方向性を持っているので、それに合わせて from と to の引数を指定する必要があります。 今後の操作に影響するように directed 引数を TRUE に設定します。
## epicontacts オブジェクトを生成
epic <- make_epicontacts(
linelist = linelist,
contacts = contacts,
id = "case_id",
from = "infector",
to = "case_id",
directed = TRUE
)Warning in make_epicontacts(linelist = linelist, contacts = contacts, id =
"case_id", : Cycle(s) detected in the contact network: this may be unwantedepicontacts オブジェクトを表示してみると、linelist の case_id の列名が id に、case_id と infector の列名が from と to に変更されています。 この変更により一貫性が確保され、その後の処理、視覚化、分析の操作が容易になります。
## epicontacts オブジェクトを表示
epic
/// Epidemiological Contacts //
// class: epicontacts
// 5,888 cases in linelist; 3,800 contacts; directed
// linelist
# A tibble: 5,888 × 30
id generation date_infection date_onset date_hospitalisation date_outcome
<chr> <dbl> <date> <date> <date> <date>
1 5fe599 4 2014-05-08 2014-05-13 2014-05-15 NA
2 8689b7 4 NA 2014-05-13 2014-05-14 2014-05-18
3 11f8ea 2 NA 2014-05-16 2014-05-18 2014-05-30
4 b8812a 3 2014-05-04 2014-05-18 2014-05-20 NA
5 893f25 3 2014-05-18 2014-05-21 2014-05-22 2014-05-29
6 be99c8 3 2014-05-03 2014-05-22 2014-05-23 2014-05-24
7 07e3e8 4 2014-05-22 2014-05-27 2014-05-29 2014-06-01
8 369449 4 2014-05-28 2014-06-02 2014-06-03 2014-06-07
9 f393b4 4 NA 2014-06-05 2014-06-06 2014-06-18
10 1389ca 4 NA 2014-06-05 2014-06-07 2014-06-09
# ℹ 5,878 more rows
# ℹ 24 more variables: outcome <chr>, gender <chr>, age <dbl>, age_unit <chr>,
# age_years <dbl>, age_cat <fct>, age_cat5 <fct>, hospital <chr>, lon <dbl>,
# lat <dbl>, infector <chr>, source <chr>, wt_kg <dbl>, ht_cm <dbl>,
# ct_blood <dbl>, fever <chr>, chills <chr>, cough <chr>, aches <chr>,
# vomit <chr>, temp <dbl>, time_admission <chr>, bmi <dbl>,
# days_onset_hosp <dbl>
// contacts
# A tibble: 3,800 × 4
from to location duration
<chr> <chr> <chr> <int>
1 f547d6 5fe599 Nosocomial 2
2 f90f5f b8812a Community 8
3 11f8ea 893f25 Nosocomial 7
4 aec8ec be99c8 Community 8
5 893f25 07e3e8 Nosocomial 5
6 133ee7 369449 Community 1
7 996f3a 2978ac Nosocomial 3
8 133ee7 57a565 Community 9
9 37a6f6 fc15ef Nosocomial 4
10 9f6884 2eaa9a Nosocomial 4
# ℹ 3,790 more rows37.3 操作
サブセット化
epicontacts オブジェクトの subset() メソッドは、特に、linelist のプロパティ(“ノード属性”)と contacts データベースのプロパティ(“エッジ属性”)に基づいてネットワークをフィルタリングできます。 これらの値は、名前付きリストとして、それぞれの引数に渡す必要があります。 例えば、以下のコードでは、感染日が 2014 年 4 月から 7 月の間(日付は範囲で指定)で、病院内で発生した感染リンクを持つ男性症例のみを linelist に残しています。
sub_attributes <- subset(
epic,
node_attribute = list(
gender = "m",
date_infection = as.Date(c("2014-04-01", "2014-07-01"))
),
edge_attribute = list(location = "Nosocomial")
)
sub_attributes
/// Epidemiological Contacts //
// class: epicontacts
// 69 cases in linelist; 1,925 contacts; directed
// linelist
# A tibble: 69 × 30
id generation date_infection date_onset date_hospitalisation date_outcome
<chr> <dbl> <date> <date> <date> <date>
1 5fe599 4 2014-05-08 2014-05-13 2014-05-15 NA
2 893f25 3 2014-05-18 2014-05-21 2014-05-22 2014-05-29
3 2978ac 4 2014-05-30 2014-06-06 2014-06-08 2014-06-15
4 57a565 4 2014-05-28 2014-06-13 2014-06-15 NA
5 fc15ef 6 2014-06-14 2014-06-16 2014-06-17 2014-07-09
6 99e8fa 7 2014-06-24 2014-06-28 2014-06-29 2014-07-09
7 f327be 6 2014-06-14 2014-07-12 2014-07-13 2014-07-14
8 90e5fe 5 2014-06-18 2014-07-13 2014-07-14 2014-07-16
9 a47529 5 2014-06-13 2014-07-17 2014-07-18 2014-07-26
10 da8ecb 5 2014-06-20 2014-07-18 2014-07-20 2014-08-01
# ℹ 59 more rows
# ℹ 24 more variables: outcome <chr>, gender <chr>, age <dbl>, age_unit <chr>,
# age_years <dbl>, age_cat <fct>, age_cat5 <fct>, hospital <chr>, lon <dbl>,
# lat <dbl>, infector <chr>, source <chr>, wt_kg <dbl>, ht_cm <dbl>,
# ct_blood <dbl>, fever <chr>, chills <chr>, cough <chr>, aches <chr>,
# vomit <chr>, temp <dbl>, time_admission <chr>, bmi <dbl>,
# days_onset_hosp <dbl>
// contacts
# A tibble: 1,925 × 4
from to location duration
<chr> <chr> <chr> <int>
1 f547d6 5fe599 Nosocomial 2
2 11f8ea 893f25 Nosocomial 7
3 893f25 07e3e8 Nosocomial 5
4 996f3a 2978ac Nosocomial 3
5 37a6f6 fc15ef Nosocomial 4
6 9f6884 2eaa9a Nosocomial 4
7 4802b1 bbfa93 Nosocomial 3
8 a75c7f 7f5a01 Nosocomial 3
9 8e104d ddddee Nosocomial 8
10 b799eb bc2adf Nosocomial 10
# ℹ 1,915 more rowsthin 関数を使って、引数に what = "linelist" を設定することで、contacts で見つかった症例を含むように linelist をフィルタリングしたり、引数に what = "contacts" を設定することで、contact で見つかった症例を含むように contacts をフィルタリングしたりできます。 以下のコードでは、epicontacts オブジェクトをさらにフィルタリングして、上記でフィルタリングした 4 月から 7 月の間に感染した男性の症例を含む感染リンクのみを保持しています。 この仕様に合うのは、2 つの既知の感染リンクだけであることがわかります。
sub_attributes <- thin(sub_attributes, what = "contacts")
nrow(sub_attributes$contacts)[1] 4ネットワークは、ノードやエッジの属性によるサブセットに加えて、特定のノードに接続されているコンポーネントのみを含むように切り取れます。 cluster_id 引数は、症例 ID のベクトルを取り、それらの ID に直接または間接的にリンクされた個人の linelist を返します。 以下のコードでは、2ae019 と 71577a を含むクラスタに合計 13 件の linelist が関与していることがわかります。
sub_id <- subset(epic, cluster_id = c("2ae019","71577a"))
nrow(sub_id$linelist)[1] 13また、epicontacts オブジェクトの subset() メソッドでは、cs, cs_min, cs_maxという引数を使って、クラスタのサイズでフィルタリングできます。 以下のコードでは、10 件以上のクラスタにリンクしている症例のみを保持しており、そのクラスタ内には 271 件のラインリストの症例があることがわかります。
sub_cs <- subset(epic, cs_min = 10)
nrow(sub_cs$linelist)[1] 271ID にアクセス
get_id() は、データセットに含まれる症例 ID の情報を取得するもので、 以下のようにパラメータを指定できます。
- linelist:linelist データの ID
- contacts:contacts データセットに含まれる ID(“from” と “to” の組み合わせ)
- from:contacts データセットの “from” 列の ID
- to:contacts データセットの “to” 列に含まれる ID
- all:いずれかのデータセットに含まれる ID
- common:contacts データセットと linelist の両方に出現する ID
例えば、contacts データセットの最初の 10 個の ID は何でしょうか?
contacts_ids <- get_id(epic, "contacts")
head(contacts_ids, n = 10) [1] "f547d6" "f90f5f" "11f8ea" "aec8ec" "893f25" "133ee7" "996f3a" "37a6f6"
[9] "9f6884" "4802b1"linelist と contacts の両方に何個の ID があるでしょうか?
length(get_id(epic, "common"))[1] 435237.4 可視化
基本のプロット
epicontacts オブジェクトの可視化はすべて plot 関数で行います。 まず、subset 関数を使って、発症日が 2014 年 6 月の症例のみを含むように epicontacts オブジェクトをフィルタリングし、thin を使って、それらの症例にリンクしている contacts のみを含めます。
## epicontacts オブジェクトをサブセット化
sub <- epic %>%
subset(
node_attribute = list(date_onset = c(as.Date(c("2014-06-30", "2014-06-01"))))
) %>%
thin("contacts")そして、次のように簡単に基本的な動的なプロットを作成できます。
## epicontacts オブジェクトをプロット
plot(
sub,
width = 700,
height = 700
)ノードをドラッグして移動したり、ノードにカーソルを合わせて詳細情報を表示したり、ノードをクリックして接続された症例を強調表示したりできます。
このプロットをさらに変更するための引数は数多くあります。 ここでは主なものを説明しますが、関数の引数の完全な説明を得るには、?vis_epicontacts(epicontacts オブジェクトで plot を使用したときに呼び出される関数)経由でドキュメントをチェックしてください。
ノード属性を可視化
ノードの色、ノードの形、ノードの大きさは、node_color, node_shape, node_size という引数を用いて、linelist の任意の列にマッピングできます。 これは ggplot2 パッケージでおなじみの aes シンタックスに似ています。
ノードの色、形、サイズは以下のように指定できます。
色
col_pal引数を介して、以下のように各色を手動で指定するための名前付きリストを提供できます。またはcolorRampPalette(c("black", "red", "orange"))のようなカラーパレット関数を提供することで、指定された色の間のグラデーションを提供できます。形
shapes引数に名前付きリストを渡して、node_shape引数で指定された linelist の列にあるユニークな要素ごとに 1 つの形を指定します。利用可能な形についてはcodeawesomeを参照してください。サイズ
size_range引数にノードの大きさの範囲を渡します。
以下では、色は結果を、形は性別を、大きさは年齢を表す例を示しています。
plot(
sub,
node_color = "outcome",
node_shape = "gender",
node_size = "age",
col_pal = c(Death = "firebrick", Recover = "green"),
shapes = c(f = "female", m = "male"),
size_range = c(40, 60),
height = 700,
width = 700
)エッジ属性を可視化
エッジの色、幅、線種は、edge_color, edge_width, edge_linetype の各引数を用いて、コンタクトデータフレームの任意の列にマッピングできます。 エッジの色と幅は以下のように指定できます。
色 は
edge_col_palで、col_palと同様の方法で指定します。幅
width_range引数にノードの大きさの範囲を渡すことで指定します。
以下に例を示します。
plot(
sub,
node_color = "outcome",
node_shape = "gender",
node_size = 'age',
col_pal = c(Death = "firebrick", Recover = "green"),
shapes = c(f = "female", m = "male"),
size_range = c(40, 60),
edge_color = 'location',
edge_linetype = 'location',
edge_width = 'duration',
edge_col_pal = c(Community = "orange", Nosocomial = "purple"),
width_range = c(1, 3),
height = 700,
width = 700
)時間軸
また、x_axis の引数を linelist の列にマッピングすることで、ネットワークを時間軸に沿って可視化できます。 下の例では、x 軸は症状が現れた日付を表しています。 また、矢印が大きすぎないように arrow_size 引数を指定し、図が見づらくならないように label = FALSE を設定しています。
plot(
sub,
x_axis = "date_onset",
node_color = "outcome",
col_pal = c(Death = "firebrick", Recover = "green"),
arrow_size = 0.5,
node_size = 13,
label = FALSE,
height = 700,
width = 700
)多数存在する追加の引数によって、このネットワークの外観を時間軸に沿ってより明確にできます。 これらの引数については、?vis_temporal_interactive で確認できます(x_axis を指定した epicontacts オブジェクトで plot を使用したときに呼び出される関数)。 以下、いくつか引数の例を紹介します。
伝播ツリーの形状を指定
伝播ツリー (transmission tree) の分岐の形状には大きく分けて 2 種類あり、network_shape 引数で指定できます。 1 つ目は、上図のような「枝分かれ」(branching)型で、2 つのノードを直線のエッジで接続します。 これは最も直感的な表現ですが、密に接続されたネットワークではエッジが重なってしまう可能性があります。 2 つ目の形状は「直角」(rectangle)で、これは系統樹のような木を表現します。 例えば、以下のようになります。
plot(
sub,
x_axis = "date_onset",
network_shape = "rectangle",
node_color = "outcome",
col_pal = c(Death = "firebrick", Recover = "green"),
arrow_size = 0.5,
node_size = 13,
label = FALSE,
height = 700,
width = 700
)各症例のノードは position_dodge の引数を色々と試すことで、ユニークな垂直方向の位置を割り当てできます。 unlinked_pos 引数を使って、接続されていない症例(つまり、報告された contacts がない症例)の位置を指定します。
plot(
sub,
x_axis = "date_onset",
network_shape = "rectangle",
node_color = "outcome",
col_pal = c(Death = "firebrick", Recover = "green"),
position_dodge = TRUE,
unlinked_pos = "bottom",
arrow_size = 0.5,
node_size = 13,
label = FALSE,
height = 700,
width = 700
)子ノードに対する親ノードの相対的な位置は、parent_pos 引数で指定できます。 デフォルトでは、親ノードは中央に配置されますが、下に配置したり(parent_pos = 'bottom')、上に配置したり(parent_pos = 'top')できます。
plot(
sub,
x_axis = "date_onset",
network_shape = "rectangle",
node_color = "outcome",
col_pal = c(Death = "firebrick", Recover = "green"),
parent_pos = "top",
arrow_size = 0.5,
node_size = 13,
label = FALSE,
height = 700,
width = 700
)プロットと図を保存
VisNetwork パッケージの visSave 関数を使うと、プロットを動的な自己完結型の html ファイルとして保存できます。
plot(
sub,
x_axis = "date_onset",
network_shape = "rectangle",
node_color = "outcome",
col_pal = c(Death = "firebrick", Recover = "green"),
parent_pos = "top",
arrow_size = 0.5,
node_size = 13,
label = FALSE,
height = 700,
width = 700
) %>%
visNetwork::visSave("network.html")これらのネットワーク出力を画像として保存するのは、残念ながらあまり簡単ではなく、ファイルを html として保存した後、webshot パッケージを使ってこのファイルのスクリーンショットを撮る必要があります。 以下のコードでは、上記で保存した html ファイルを PNG に変換しています。
webshot(url = "network.html", file = "network.png")タイムライン
また、各症例の X 軸に表示されているネットワークのタイムラインを表示できます。 これは、例えば、症例の位置や結果が出るまでの時間を可視化するために使用できます。 タイムラインを生成するには、症例 ID、「イベント」の開始日、「イベント」の終了日を示す少なくとも3つの列からなるデータフレームを作成する必要があります。 また、他の列をいくつでも追加し、タイムラインのノードとエッジのプロパティにマッピングできます。 以下のコードでは、症状が出た日から結果が出た日までのタイムラインを作成し、ノードの形と色を定義するために使用する結果と病院の変数を保持しています。 複数の病院間で転院した場合など、症例ごとに複数のタイムライン行・イベントを持つことができることに注意してください。
## timeline を生成
timeline <- linelist %>%
transmute(
id = case_id,
start = date_onset,
end = date_outcome,
outcome = outcome,
hospital = hospital
)上記のタイムラインオブジェクトを timeline 引数に渡します。 タイムラインのアトリビュートをタイムラインのノードの色、形、サイズにマッピングする方法は、前のセクションで定義したのと同じですが、各タイムラインの開始ノードと終了ノードの 2 つのノードがあり、それぞれ別の引数を持ちます。 例えば、tl_start_node_color はどのタイムラインの列が開始ノードの色にマッピングされるかを定義し、tl_end_node_shape はどのタイムラインの列が終了ノードの形にマッピングされるかを定義します。 また、色、幅、線種、ラベルを tl_edge_* 引数でタイムラインの edge にマッピングもできます。
引数についての詳しい説明は、?vis_temporal_interactive (epicontactsオブジェクトをプロットするときに呼ばれる関数)を参照してください。 各引数は以下のコードでも注釈がついています。
## shapes を定義
shapes <- c(
f = "female",
m = "male",
Death = "user-times",
Recover = "heartbeat",
"NA" = "question-circle"
)
## colours を定義
colours <- c(
Death = "firebrick",
Recover = "green",
"NA" = "grey"
)
## プロットを作成
plot(
sub,
## x 軸を発症日に指定
x_axis = "date_onset",
## ネットワーク形状に rectangular を使用
network_shape = "rectangle",
## 症例ノードの形状に gender 列を割り当て
node_shape = "gender",
## ノードの色にはどの列も割り当てない。
## デフォルトでは node id に割り当てられており、色スキームが滅茶苦茶になってしまうので、重要
node_color = NULL,
## 症例 node の大きさを 30 にする。(これは文字列ではありません。
## node_size は列に割り当てていないので、実際のノード の大きさと解釈されます。)
node_size = 30,
## 感染 link の大きさを 4 にする。(これは文字列ではありません。
## edge_width は列に割り当てていないので、実際の edge の大きさと解釈されます。)
edge_width = 4,
## timeline オブジェクトを渡す
timeline = timeline,
## timeline オブジェクトのノードの末端形状を outcome 列に割り当て
tl_end_node_shape = "outcome",
## ノード末端の大きさを 15 にする。(これは文字列ではありません。
## 引数は列に割り当てていないので、実際のノードの大きさと解釈されます。)
tl_end_node_size = 15,
## timeline edge の色を hospital 列に割り当て
tl_edge_color = "hospital",
## timeline エッジの大きさを 2 にする。(これは文字列ではありません。
## 引数は列に割り当てていないので、実際のエッジの大きさと解釈されます。)
tl_edge_width = 2,
## エッジのラベルに hospital 変数を割り当て
tl_edge_label = "hospital",
## 全員のノード属性の形状を指定 (定義済み)
shapes = shapes,
## 色パレットを指定 (定義済み)
col_pal = colours,
## 矢印の大きさを 0.5 に設定
arrow_size = 0.5,
## 凡例を 2 列に
legend_ncol = 2,
## フォントの大きさを設定
font_size = 15,
## 日付のフォーマットを設定
date_labels = c("%d %b %Y"),
## ノードの下に ID ラベルをプロットしない
label = FALSE,
## 高さを指定
height = 1000,
## 幅を指定
width = 1200,
## 各症例のノードがユニークな y 座標を持つようにする
## これをしないと異なる症例が重なってしまうので、
## タイムラインを使うときには重要
position_dodge = TRUE
)Warning in assert_timeline(timeline, x, x_axis): 5865 timeline row(s) removed
as ID not found in linelist or start/end date is NA37.5 解析
要約化
ネットワークのプロパティの概要は、summary 関数で取得できます。
## epicontacts オブジェクトを要約
summary(epic)
/// Overview //
// number of unique IDs in linelist: 5888
// number of unique IDs in contacts: 5511
// number of unique IDs in both: 4352
// number of contacts: 3800
// contacts with both cases in linelist: 56.868 %
/// Degrees of the network //
// in-degree summary:
Min. 1st Qu. Median Mean 3rd Qu. Max.
0.0000 0.0000 1.0000 0.5392 1.0000 1.0000
// out-degree summary:
Min. 1st Qu. Median Mean 3rd Qu. Max.
0.0000 0.0000 0.0000 0.5392 1.0000 6.0000
// in and out degree summary:
Min. 1st Qu. Median Mean 3rd Qu. Max.
0.000 1.000 1.000 1.078 1.000 7.000
/// Attributes //
// attributes in linelist:
generation date_infection date_onset date_hospitalisation date_outcome outcome gender age age_unit age_years age_cat age_cat5 hospital lon lat infector source wt_kg ht_cm ct_blood fever chills cough aches vomit temp time_admission bmi days_onset_hosp
// attributes in contacts:
location duration例えば、両方の症例が登録されている contact は 57%しかありません。 これは、このような感染連鎖に関わる症例のうちかなりの数の登録データがないことを意味しています。
ペアの特徴
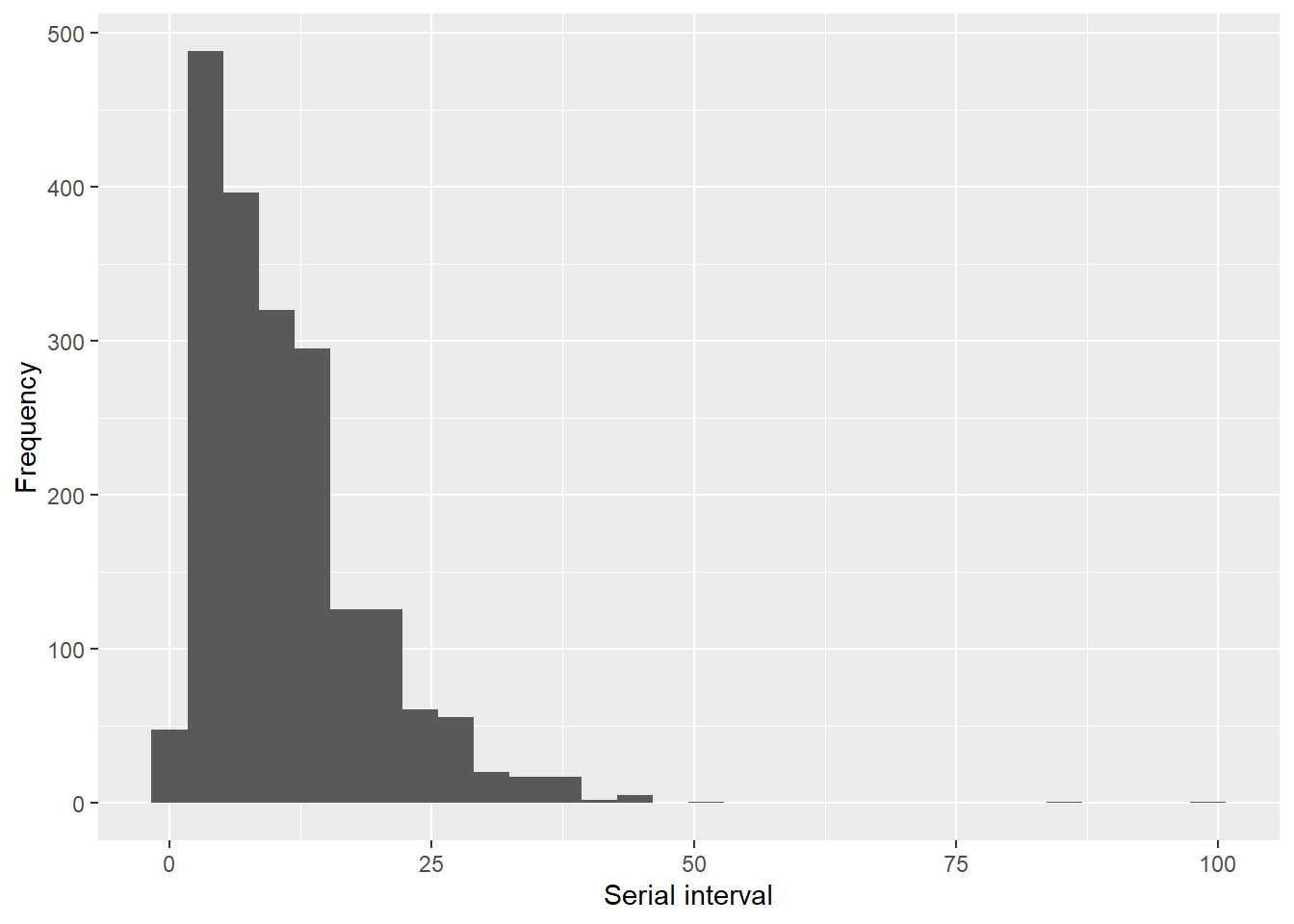
get_pairwise() 関数は、接触データセットの各ペアに応じて、linelist の変数を処理できます。 以下の例では、linelist から発症日を抽出し、各ペアの発症日の差を計算しています。 この比較から得られる値は、シリアル・インターバル (si) を表します。
si <- get_pairwise(epic, "date_onset")
summary(si) Min. 1st Qu. Median Mean 3rd Qu. Max. NA's
0.00 5.00 9.00 11.01 15.00 99.00 1820 tibble(si = si) %>%
ggplot(aes(si)) +
geom_histogram() +
labs(
x = "Serial interval",
y = "Frequency"
)`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.Warning: Removed 1820 rows containing non-finite outside the scale range
(`stat_bin()`).get_pairwise() は、比較に使われる列のデータ型を解釈し、それに応じて値の比較方法を調整します。 上記の si の例のように、数値や日付の場合、この関数は値を引きます。 文字やカテゴライズされた列に適用された場合、get_pairwise() は値をくっつけます。 この関数は任意の処理も可能なので(引数 “f” を参照)、これらの離散的な組み合わせを簡単に集計・分析できます。
head(get_pairwise(epic, "gender"), n = 10) [1] "f -> m" NA "m -> m" NA "m -> f" "f -> f" NA "f -> m"
[9] NA "m -> f"get_pairwise(epic, "gender", f = table) values.to
values.from f m
f 464 516
m 510 468fisher.test(get_pairwise(epic, "gender", f = table))
Fisher's Exact Test for Count Data
data: get_pairwise(epic, "gender", f = table)
p-value = 0.03758
alternative hypothesis: true odds ratio is not equal to 1
95 percent confidence interval:
0.6882761 0.9892811
sample estimates:
odds ratio
0.8252575 ここでは、感染リンクと性別との間に有意な関連が見られます。
クラスターを特定
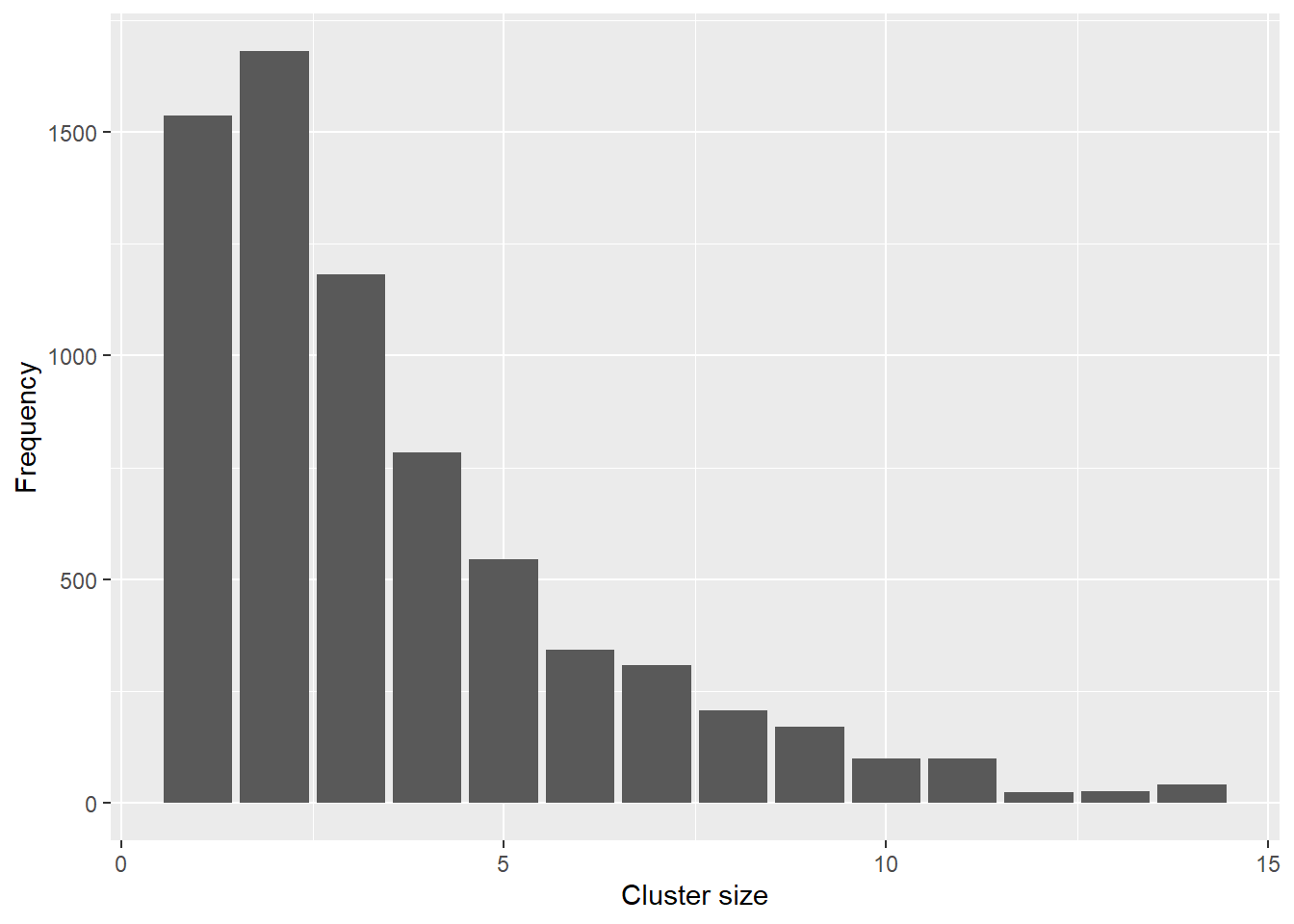
get_clusters() 関数を使って、epicontacts オブジェクト内の連結成分を特定できます。 まず、クラスター情報を含む data.frame を取得するために使います。
clust <- get_clusters(epic, output = "data.frame")
table(clust$cluster_size)
1 2 3 4 5 6 7 8 9 10 11 12 13 14
1536 1680 1182 784 545 342 308 208 171 100 99 24 26 42 ggplot(clust, aes(cluster_size)) +
geom_bar() +
labs(
x = "Cluster size",
y = "Frequency"
)最大のクラスターを見てみましょう。 このためには、epicontacts オブジェクトにクラスタ情報を追加し、それをサブセット化して、最大のクラスタだけを残します。
epic <- get_clusters(epic)
max_size <- max(epic$linelist$cluster_size)
plot(subset(epic, cs = max_size))度数を計算
ノードの度数 (degree) は、他のノードとのエッジや接続の数に相当しています。 get_degree() は epicontacts ネットワークに対してこの値を計算する簡単な方法を提供します。 この文脈において、度数が高いほど、多くの人と接触していた個人であることを示しています。 type の引数は、in-degree と out-degree の両方をカウントしたいことを示し、only_linelist の引数は、ラインリストに含まれる症例についてのみ度数を計算したいことを示します。
deg_both <- get_degree(epic, type = "both", only_linelist = TRUE)最も多く接触した人(上から順に 10 人)は誰でしょうか?
head(sort(deg_both, decreasing = TRUE), 10)916d0a 858426 6833d7 f093ea 11f8ea 3a4372 38fc71 c8c4d5 a127a7 02d8fd
7 6 6 6 5 5 5 5 5 5 平均接触回数はどのくらいですか?
mean(deg_both)[1] 1.07847337.6 参考資料
epicontacts ページ では、パッケージの機能の概要を説明しており、より詳細なドキュメント(vignette)も含まれています。
github ページ は、問題提起や機能のリクエストに利用できます。