Warning in epicontacts::make_epicontacts(linelist = linelist, contacts =
contacts, : Cycle(s) detected in the contact network: this may be unwanted37 Цепочки передачи
37.1 Обзор
Основным инструментом для обработки, анализа и визуализации цепочек передачи и данных о контактах является пакет epicontacts, разработанный специалистами компании RECON. Попробуйте воспользоваться приведенным ниже интерактивным графиком, наводя курсор на вершины для получения дополнительной информации, перетаскивая их для перемещения и нажимая на них для выделения случаев заболевания.
37.2 Подготовка
Загрузка пакетов
Сначала загрузите стандартные пакеты, необходимые для импорта и управления данными. В данном руководстве мы делаем акцент на функции p_load() из pacman, которая при необходимости устанавливает пакет и загружает его для использования. Также можно загрузить пакеты с помощью library() из базового R. Более подробную информацию о пакетах R см. на странице [Основы R].
pacman::p_load(
rio, # Импорт файлов
here, # Локатор файлов
tidyverse, # Управление данными + графика ggplot2
remotes # Установка пакетов из github
)Вам потребуется версия epicontacts, которая может быть установлена из github с помощью функции p_install_github() из pacman. Эту команду необходимо выполнить только ниже, а не при каждом использовании пакета (в дальнейшем можно использовать p_load() как обычно).
pacman::p_install_gh("reconhub/epicontacts@timeline")Импорт данных
Мы импортируем набор данных о случаях из смоделированной эпидемии лихорадки Эбола. Если вы хотите загрузить данные, чтобы проследить за ходом работы, см. инструкции на странице [Скачивание руководства и данных]. Набор данных импортируется с помощью функции import() из пакета rio. Различные способы импорта см. на странице [Импорт и экспорт].ta.
# импортировать построчный список
linelist <- import("linelist_cleaned.xlsx")Ниже показаны первые 50 строк построчного списка. Особый интерес представляют столбцы case_id, generation, infector и source.
Создание объекта epicontacts
Затем нам необходимо создать объект epicontacts, для которого требуется два типа данных:
- построчный список, документирующий случаи, где столбцы являются переменными, а строки соответствуют уникальным случаям
- список ребер, определяющих связи между случаями на основе их уникальных идентификаторов (это могут быть контакты, события передачи и т.д.)
Поскольку у нас уже есть построчный список, нам просто нужно создать список ребер между случаями, а точнее между их идентификаторами. Мы можем извлечь связи передачи из построчного списка, связав столбец infector со столбцом case_id. На этом этапе мы также можем добавить “свойства ребра”, под которыми мы понимаем любую переменную, описывающую связь между двумя случаями, а не сами случаи. Для примера добавим переменную location, описывающую местоположение события передачи, и переменную продолжительность, описывающую продолжительность контакта в днях.
В приведенном ниже коде dplyr функция transmute аналогична mutate, за исключением того, что она сохраняет только те столбцы, которые мы указали в функции. Функция drop_na отфильтрует все строки, в которых указанные столбцы имеют значение NA; в данном случае мы хотим сохранить только те строки, в которых известно заразившее лицо.
## создание контактов
contacts <- linelist %>%
transmute(
infector = infector,
case_id = case_id,
location = sample(c("Community", "Nosocomial"), n(), TRUE),
duration = sample.int(10, n(), TRUE)
) %>%
drop_na(infector)Теперь мы можем создать объект epicontacts с помощью функции make_epicontacts. Нам необходимо указать, какой столбец в построчном списке указывает на уникальный идентификатор случая, а также какие столбцы в контактах указывают на уникальные идентификаторы случаев, участвующих в каждой связи. Эти связи имеют направление в том смысле. что инфекция идет от заразившего лица к случаю, поэтому нам необходимо задать аргументы from и to соответственно. Поэтому мы также устанавливаем аргумент directed в значение TRUE, что повлияет на дальнейшие операции.
## создание объекта epicontacts
epic <- make_epicontacts(
linelist = linelist,
contacts = contacts,
id = "case_id",
from = "infector",
to = "case_id",
directed = TRUE
)Warning in make_epicontacts(linelist = linelist, contacts = contacts, id =
"case_id", : Cycle(s) detected in the contact network: this may be unwantedИзучив объекты epicontacts, мы видим, что столбец case_id в построчном списке был переименован в id, а столбцы case_id и infector в контактах были переименованы в столбцы from и to. Это обеспечивает последовательность в последующих операциях обработки, визуализации и анализа.
## просмотр объекта epicontacts
epic
/// Epidemiological Contacts //
// class: epicontacts
// 5,888 cases in linelist; 3,800 contacts; directed
// linelist
# A tibble: 5,888 × 30
id generation date_infection date_onset date_hospitalisation date_outcome
<chr> <dbl> <date> <date> <date> <date>
1 5fe599 4 2014-05-08 2014-05-13 2014-05-15 NA
2 8689b7 4 NA 2014-05-13 2014-05-14 2014-05-18
3 11f8ea 2 NA 2014-05-16 2014-05-18 2014-05-30
4 b8812a 3 2014-05-04 2014-05-18 2014-05-20 NA
5 893f25 3 2014-05-18 2014-05-21 2014-05-22 2014-05-29
6 be99c8 3 2014-05-03 2014-05-22 2014-05-23 2014-05-24
7 07e3e8 4 2014-05-22 2014-05-27 2014-05-29 2014-06-01
8 369449 4 2014-05-28 2014-06-02 2014-06-03 2014-06-07
9 f393b4 4 NA 2014-06-05 2014-06-06 2014-06-18
10 1389ca 4 NA 2014-06-05 2014-06-07 2014-06-09
# ℹ 5,878 more rows
# ℹ 24 more variables: outcome <chr>, gender <chr>, age <dbl>, age_unit <chr>,
# age_years <dbl>, age_cat <fct>, age_cat5 <fct>, hospital <chr>, lon <dbl>,
# lat <dbl>, infector <chr>, source <chr>, wt_kg <dbl>, ht_cm <dbl>,
# ct_blood <dbl>, fever <chr>, chills <chr>, cough <chr>, aches <chr>,
# vomit <chr>, temp <dbl>, time_admission <chr>, bmi <dbl>,
# days_onset_hosp <dbl>
// contacts
# A tibble: 3,800 × 4
from to location duration
<chr> <chr> <chr> <int>
1 f547d6 5fe599 Nosocomial 3
2 f90f5f b8812a Community 1
3 11f8ea 893f25 Nosocomial 9
4 aec8ec be99c8 Nosocomial 8
5 893f25 07e3e8 Nosocomial 6
6 133ee7 369449 Community 9
7 996f3a 2978ac Nosocomial 4
8 133ee7 57a565 Community 9
9 37a6f6 fc15ef Nosocomial 1
10 9f6884 2eaa9a Community 4
# ℹ 3,790 more rows37.3 Обработка
Подмножество
Метод subset() для объектов epicontacts позволяет, в частности, фильтровать сети на основе свойств построчного списка (“атрибуты узлов”) и базы данных контактов (“атрибуты ребер”). Эти значения должны быть переданы в виде списков с названиями соответствующему аргументу. Например, в приведенном ниже коде мы сохраняем в списке только те мужские случаи в построенном списке, которые имеют дату заражения в период с апреля по июль 2014 года (даты указаны в виде диапазонов), а также связи передачи, произошедшие в больнице.
sub_attributes <- subset(
epic,
node_attribute = list(
gender = "m",
date_infection = as.Date(c("2014-04-01", "2014-07-01"))
),
edge_attribute = list(location = "Nosocomial")
)
sub_attributes
/// Epidemiological Contacts //
// class: epicontacts
// 69 cases in linelist; 1,913 contacts; directed
// linelist
# A tibble: 69 × 30
id generation date_infection date_onset date_hospitalisation date_outcome
<chr> <dbl> <date> <date> <date> <date>
1 5fe599 4 2014-05-08 2014-05-13 2014-05-15 NA
2 893f25 3 2014-05-18 2014-05-21 2014-05-22 2014-05-29
3 2978ac 4 2014-05-30 2014-06-06 2014-06-08 2014-06-15
4 57a565 4 2014-05-28 2014-06-13 2014-06-15 NA
5 fc15ef 6 2014-06-14 2014-06-16 2014-06-17 2014-07-09
6 99e8fa 7 2014-06-24 2014-06-28 2014-06-29 2014-07-09
7 f327be 6 2014-06-14 2014-07-12 2014-07-13 2014-07-14
8 90e5fe 5 2014-06-18 2014-07-13 2014-07-14 2014-07-16
9 a47529 5 2014-06-13 2014-07-17 2014-07-18 2014-07-26
10 da8ecb 5 2014-06-20 2014-07-18 2014-07-20 2014-08-01
# ℹ 59 more rows
# ℹ 24 more variables: outcome <chr>, gender <chr>, age <dbl>, age_unit <chr>,
# age_years <dbl>, age_cat <fct>, age_cat5 <fct>, hospital <chr>, lon <dbl>,
# lat <dbl>, infector <chr>, source <chr>, wt_kg <dbl>, ht_cm <dbl>,
# ct_blood <dbl>, fever <chr>, chills <chr>, cough <chr>, aches <chr>,
# vomit <chr>, temp <dbl>, time_admission <chr>, bmi <dbl>,
# days_onset_hosp <dbl>
// contacts
# A tibble: 1,913 × 4
from to location duration
<chr> <chr> <chr> <int>
1 f547d6 5fe599 Nosocomial 3
2 11f8ea 893f25 Nosocomial 9
3 aec8ec be99c8 Nosocomial 8
4 893f25 07e3e8 Nosocomial 6
5 996f3a 2978ac Nosocomial 4
6 37a6f6 fc15ef Nosocomial 1
7 4802b1 bbfa93 Nosocomial 3
8 ea3740 90e5fe Nosocomial 3
9 beb26e 959170 Nosocomial 7
10 567136 8ebf6e Nosocomial 8
# ℹ 1,903 more rowsМы можем использовать функцию thin либо для фильтрации построчного списка, чтобы включить в него случаи которые встречаются в контактах, задав аргумент what = " linelist", либо отфильтровать контакты для включения случаев, найденных в построчном списке, задав аргумент what = "contacts". В приведенном ниже коде мы дополнительно фильтруем объект epicontacts, чтобы сохранить только связи передачи, связанные со случаями заражения мужчин в период с апреля по июль, которые мы отфильтровали выше. Мы видим, что только две известные связи соответствуют этому требованию.
sub_attributes <- thin(sub_attributes, what = "contacts")
nrow(sub_attributes$contacts)[1] 5В дополнение к подмножеству по атрибутам узлов и ребер, сети могут быть обрезаны таким образом, чтобы включать только те компоненты, которые связаны с определенными узлами. Аргумент cluster_id принимает вектор идентификаторов случаев и возвращает построчный список лиц, которые прямо или косвенно связаны с этими идентификаторами. В приведенном ниже коде мы видим, что в кластерах, содержащих 2ae019 и 2ae019, задействовано в общей сложности 13 построчных списков.
sub_id <- subset(epic, cluster_id = c("2ae019","71577a"))
nrow(sub_id$linelist)[1] 13Метод subset() для объектов epicontacts также позволяет осуществлять фильтрацию по размеру кластера используя аргументы cs, cs_min и cs_max. В приведенном ниже коде мы сохраняем только случаи, связанные с кластерами, состоящими из 10 случаев и более, и видим, что 271 случай из построчного списка участвует в таких кластерах.
sub_cs <- subset(epic, cs_min = 10)
nrow(sub_cs$linelist)[1] 271Доступ к идентификаторам
Функция get_id() извлекает информацию об идентификаторах случаев в наборе данных и может быть определена следующим образом:
- linelist: Идентификаторы в данных построчного списка
- contacts: Идентификаторы в наборе данных о контактах (объединение “от” и ” к”)
- from: идентификаторы в столбце ““от”” набора данных о контактах
- to: идентификаторы в столбце “к” набора данных о контактах
- all: Идентификаторы, встречающиеся в любом месте любого из наборов данных
- common: Идентификаторы, которые встречаются как в наборе данных контактов, так и в построчном списке
Например, каковы первые десять идентификаторов в наборе данных контактов?
contacts_ids <- get_id(epic, "contacts")
head(contacts_ids, n = 10) [1] "f547d6" "f90f5f" "11f8ea" "aec8ec" "893f25" "133ee7" "996f3a" "37a6f6"
[9] "9f6884" "4802b1"Сколько идентификаторов содержится в построчном списке и в контактах?
length(get_id(epic, "common"))[1] 435237.4 Визуализация
Базовое построение графиков
Все визуализации объектов epicontacts обрабатываются функцией plot. Сначала отфильтруем объект epicontacts, чтобы включить в него только случаи с датой начала заболевания в июне 2014 года, используя функцию subset, и включим только контакты, связанные с этими случаями, с помощью функции thin.
## объект подмножества epicontacts
sub <- epic %>%
subset(
node_attribute = list(date_onset = c(as.Date(c("2014-06-30", "2014-06-01"))))
) %>%
thin("contacts")После этого мы можем построить базовый интерактивный график следующим образом:
## построение объекта epicontacts
plot(
sub,
width = 700,
height = 700
)Узлы можно перемещать, перетаскивая их, наводить на них курсор, чтобы получить дополнительную информацию, и щелкать по ним мышью, выделяя связанные случаи.
Существует большое количество аргументов для дальнейшей модификации этого графика. Мы рассмотрим основные из них, но ознакомьтесь с документацией по функции ?vis_epicontacts (функция, вызываемая при использовании plot на объекте epicontacts), чтобы получить полное описание аргументов функции.
Визуализация атрибутов узлов
Цвет узла, форма узла и его размер могут быть сопоставлены с заданным столбцом построчного списка используя аргументы node_color, node_shape и node_size. Это аналогично синтаксису aes, который вы можете узнать из ggplot2.
Конкретные цвета, формы и размеры узлов могут быть заданы следующим образом:
Цвета через аргумент
col_pal, либо предоставив список названий для указания каждого цвета вручную, как это сделано ниже, либо задав функцию палитры цветов,
например,colorRampPalette(c("black", "red", "orange")), которая обеспечит градиент между указанными цветами.Формы, передавая список с названием в аргументе
shapes, указывая одну форму для каждого уникального элемента построчного списка, указанного аргументомnode_shape. Доступные формы см. в разделеcodeawesome.Размер путем передачи диапазона размеров узлов в аргумент
size_range.
Приведем пример, где цвет обозначает результат, форма - пол, а размер возраст:
plot(
sub,
node_color = "outcome",
node_shape = "gender",
node_size = "age",
col_pal = c(Death = "firebrick", Recover = "green"),
shapes = c(f = "female", m = "male"),
size_range = c(40, 60),
height = 700,
width = 700
)Визуализация атрибутов ребер
Цвет, ширина и тип линии ребра могут быть привязаны к заданному столбцу в датафрейме контактов с помощью edge_color, edge_width и edge_linetype. Конкретные цвета и ширина ребер могут быть заданы следующим образом:
Цвета через аргумент
edge_col_pal, аналогично тому, как это делается дляcol_pal.Ширины, передавая диапазон размеров узлов в аргументе
width_range.
Приведем пример:
plot(
sub,
node_color = "outcome",
node_shape = "gender",
node_size = 'age',
col_pal = c(Death = "firebrick", Recover = "green"),
shapes = c(f = "female", m = "male"),
size_range = c(40, 60),
edge_color = 'location',
edge_linetype = 'location',
edge_width = 'duration',
edge_col_pal = c(Community = "orange", Nosocomial = "purple"),
width_range = c(1, 3),
height = 700,
width = 700
)Временная ось
Мы также можем визуализировать сеть вдоль временной оси, связав аргумент x_axis со столбцом построчного списка. В приведенном ниже примере ось x обозначает дату начала симптомов. Мы также указали аргумент arrow_size чтобы стрелки не были слишком большими, и задали label = FALSE, чтобы сделать рисунок менее загроможденным.
plot(
sub,
x_axis = "date_onset",
node_color = "outcome",
col_pal = c(Death = "firebrick", Recover = "green"),
arrow_size = 0.5,
node_size = 13,
label = FALSE,
height = 700,
width = 700
)Существует большое количество дополнительных аргументов, позволяющих более точно определить, как эта сеть визуализируется по временной оси, что можно проверить с помощью функции ?vis_temporal_interactive (функция, вызываемая при использовании plot на объекте epicontacts с указанной x_axis). Ниже мы рассмотрим некоторые примеры.
Задание формы дерева передачи
Существует две основные формы, которые может принимать дерево передачи, задаваемые с помощью аргумента network_shape. Первая - это разветвленная форма branching, как показано выше, где прямое ребро соединяет любые два узла. Это наиболее интуитивно понятное представление, однако в густонаселенной сети оно может привести к наложению ребер друг на друга Вторая форма - прямоугольник rectangle - представляет собой дерево, напоминающее филогенез Например:
plot(
sub,
x_axis = "date_onset",
network_shape = "rectangle",
node_color = "outcome",
col_pal = c(Death = "firebrick", Recover = "green"),
arrow_size = 0.5,
node_size = 13,
label = FALSE,
height = 700,
width = 700
)Каждому узлу случая может быть присвоено уникальное вертикальное положение путем переключения аргумента position_dodge. Положение несвязанных случаев (т.е. не имеющих контактов) задается с помощью аргумента unlinked_pos.
plot(
sub,
x_axis = "date_onset",
network_shape = "rectangle",
node_color = "outcome",
col_pal = c(Death = "firebrick", Recover = "green"),
position_dodge = TRUE,
unlinked_pos = "bottom",
arrow_size = 0.5,
node_size = 13,
label = FALSE,
height = 700,
width = 700
)Положение родительского узла относительно дочерних узлов может быть задано с помощью аргумента parent_pos. По умолчанию родительский узел располагается в середине, однако его можно расположить снизу (parent_pos = 'bottom') или сверху (parent_pos = 'top').
plot(
sub,
x_axis = "date_onset",
network_shape = "rectangle",
node_color = "outcome",
col_pal = c(Death = "firebrick", Recover = "green"),
parent_pos = "top",
arrow_size = 0.5,
node_size = 13,
label = FALSE,
height = 700,
width = 700
)Сохранение графиков и рисунков
Сохранить график в виде интерактивного, самодостаточного html-файла можно с помощью функции visSave из пакета VisNetwork:
plot(
sub,
x_axis = "date_onset",
network_shape = "rectangle",
node_color = "outcome",
col_pal = c(Death = "firebrick", Recover = "green"),
parent_pos = "top",
arrow_size = 0.5,
node_size = 13,
label = FALSE,
height = 700,
width = 700
) %>%
visNetwork::visSave("network.html")Сохранение этих исходов в виде изображения, к сожалению, не так просто и требует сохранить файл в формате html, а затем сделать скриншот этого файла с помощью пакета webshot. В приведенном ниже коде мы преобразуем сохраненный html-файл в PNG:
webshot(url = "network.html", file = "network.png")Временные рамки
В сети также можно рассматривать временные рамки, которые представлены на оси x каждого случая. Это можно сделать, например, для визуализации местоположения случая или времени до исхода. Чтобы сгенерировать временную шкалу, необходимо создать датафрейм, состоящий как минимум из трех столбцов с указанием идентификатора случая, даты начала “события” и даты окончания “события”. Можно также добавить любое количество других столбцов, которые затем можно сопоставить со свойствами узлов и ребер временной шкалы. В приведенном ниже коде мы генерируем временную шкалу, начиная с даты начала симптомов и заканчивая датой исхода, и сохраняем переменные исхода и больницы, которые мы используем для определения формы и цвета узлов. Обратите внимание, что в каждом случае может быть более одной строки/события временной шкалы на один случай, например, если случай переводится между несколькими больницами.
## создание временных рамок
timeline <- linelist %>%
transmute(
id = case_id,
start = date_onset,
end = date_outcome,
outcome = outcome,
hospital = hospital
)Затем мы передаем элемент временной шкалы в аргумент timeline. Мы можем сопоставить атрибуты временной шкалы цветам, формам и размерам узлов временной шкалы таким же образом, какой был определен в предыдущих разделах. за исключением того, что у нас есть два узла: начальный и конечный узел каждой временной шкалы, которые имеют отдельные аргументы. Например, tl_start_node_color определяет, какой столбец временной шкалы сопоставлен с цветом начального узла, а tl_end_node_shape определяет, какой столбец временной шкалы будет сопоставлен с формой конечного узла. Мы также можем сопоставить цвет, ширину, тип линий и метки с аргументами временной шкалы tl_edge_*.
Подробную документацию по аргументам см. в разделе ?vis_temporal_interactive (функция, вызываемая при построении графика объекта epicontacts). для получения подробной документации по аргументам. Каждый аргумент аннотирован в приведенном ниже коде:
## определение форм
shapes <- c(
f = "female",
m = "male",
Death = "user-times",
Recover = "heartbeat",
"NA" = "question-circle"
)
## определение цветов
colours <- c(
Death = "firebrick",
Recover = "green",
"NA" = "grey"
)
## создание графика
plot(
sub,
## максимальная координата x к дате начала заболевания
x_axis = "date_onset",
## использовать прямоугольную форму сети
network_shape = "rectangle",
## сопоставить формы узлов случая со столбоцом пола
node_shape = "gender",
## мы не хотим сопоставлять цвет узла с каким-либо столбцом - это важно, поскольку
## значение по умолчанию сопоставляется с id узла, что приводит к нарушению цветовой схемы
node_color = NULL,
## установить размер узла случая равным 30 (поскольку это не символ, node_size не
## отображается на столбец, но вместо этого интерпретируется как фактический размер узла)
node_size = 30,
## установить ширину связи передачи равной 4 (поскольку это не символ, ширина edge_width
## не отображается на столбец, а интерпретируется как фактическая ширина ребра)
edge_width = 4,
## предоставить объект временной шкалы
timeline = timeline,
## сопоставить форму конечного узла со столбцом исходов в объекте временной шкалы
tl_end_node_shape = "outcome",
## установить размер конечного узла равным 15 (поскольку это не символ,
## аргумент не сопоставляется со столбцом, а интерпретируется как фактический
## размер узла)
tl_end_node_size = 15,
## сопоставить цвет ребра временной шкалы со столбцом больницы
tl_edge_color = "hospital",
## установить ширину ребра временной шкалы равной 2 (поскольку это не символ,
## аргумент не сопоставляется со столбцом, а интерпретируется как фактическая
## ширина ребра)
tl_edge_width = 2,
## сопоставить метки ребер с переменной больницы
tl_edge_label = "hospital",
## задать форму для каждого атрибута узла (см. выше)
shapes = shapes,
## задать цветовую палитру (см. выше)
col_pal = colours,
## установить размер стрелки равным 0,5
arrow_size = 0.5,
## использовать два столбца в легенде
legend_ncol = 2,
## установить размер шрифта
font_size = 15,
## определение форматирования для дат
date_labels = c("%d %b %Y"),
## не отображать метки идентификаторов ниже узлов
label = FALSE,
## задать высоту
height = 1000,
## задать ширину
width = 1200,
## убедитесь, что каждый узел случая имеет уникальную координату y - это очень важно
## при использовании временных шкал, иначе у вас будут перекрываться временные шкалы от
## различных случаев
position_dodge = TRUE
)Warning in assert_timeline(timeline, x, x_axis): 5865 timeline row(s) removed
as ID not found in linelist or start/end date is NA37.5 Анализ
Обобщение
Мы можем получить общее представление о некоторых свойствах сети с помощью функции summary.
## обобщить объект epicontacts
summary(epic)
/// Overview //
// number of unique IDs in linelist: 5888
// number of unique IDs in contacts: 5511
// number of unique IDs in both: 4352
// number of contacts: 3800
// contacts with both cases in linelist: 56.868 %
/// Degrees of the network //
// in-degree summary:
Min. 1st Qu. Median Mean 3rd Qu. Max.
0.0000 0.0000 1.0000 0.5392 1.0000 1.0000
// out-degree summary:
Min. 1st Qu. Median Mean 3rd Qu. Max.
0.0000 0.0000 0.0000 0.5392 1.0000 6.0000
// in and out degree summary:
Min. 1st Qu. Median Mean 3rd Qu. Max.
0.000 1.000 1.000 1.078 1.000 7.000
/// Attributes //
// attributes in linelist:
generation date_infection date_onset date_hospitalisation date_outcome outcome gender age age_unit age_years age_cat age_cat5 hospital lon lat infector source wt_kg ht_cm ct_blood fever chills cough aches vomit temp time_admission bmi days_onset_hosp
// attributes in contacts:
location durationНапример, мы видим, что только 57% контактов имеют оба случая в построчном списке Это означает, что мы не располагаем данными построчного списка по значительному числу случаев, задействованных в этих цепочках передачи.
Парные характеристики
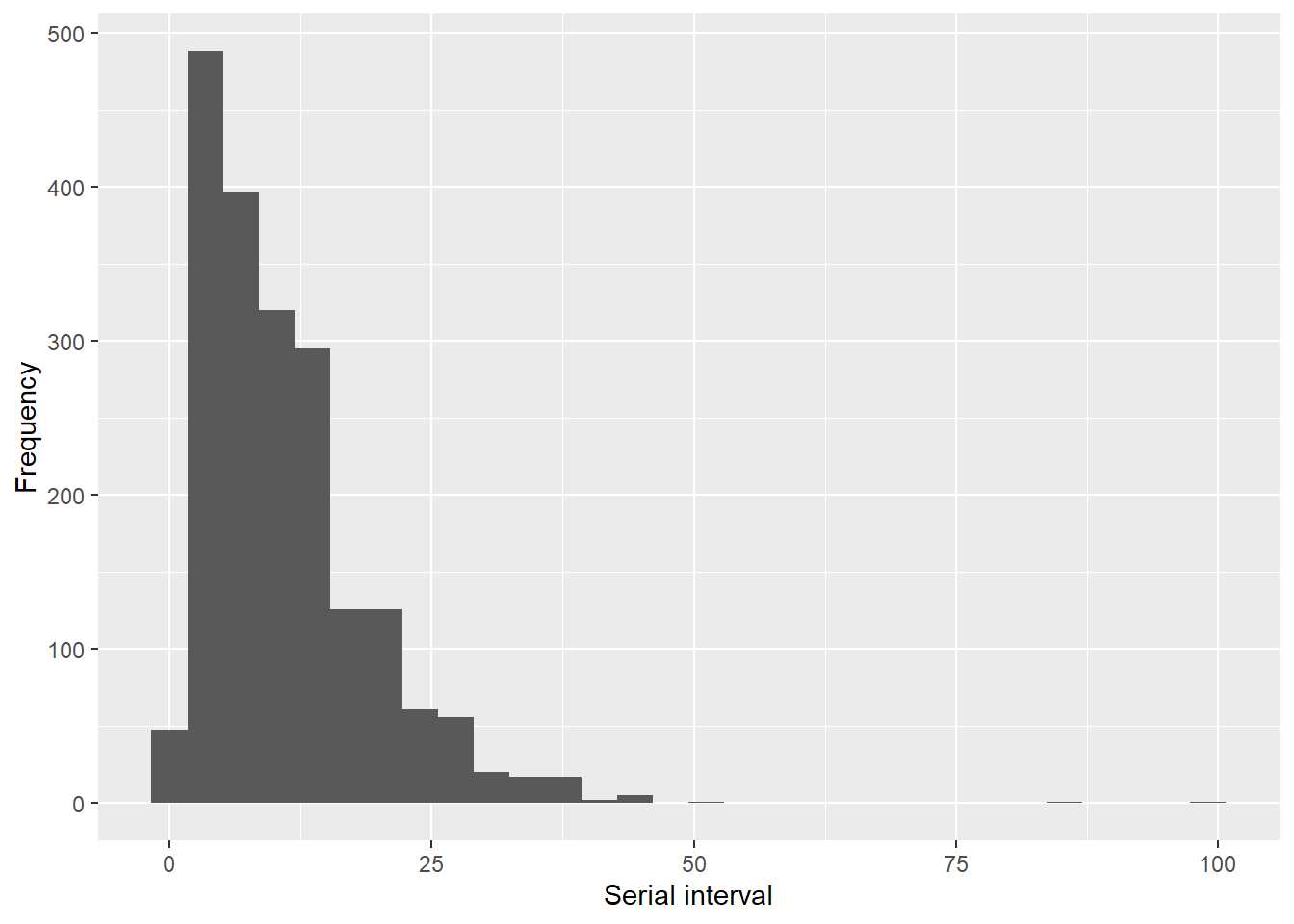
Функция get_pairwise() позволяет обрабатывать переменную(ые) в списке строк в соответствии с каждой парой в наборе данных контактов. В следующем примере из списка строк извлекается дата начала заболевания, чтобы вычислить разницу между датами начала заболевания для каждой пары. Полученное в результате этого сравнения, представляет собой серийный интервал (si).
si <- get_pairwise(epic, "date_onset")
summary(si) Min. 1st Qu. Median Mean 3rd Qu. Max. NA's
0.00 5.00 9.00 11.01 15.00 99.00 1820 tibble(si = si) %>%
ggplot(aes(si)) +
geom_histogram() +
labs(
x = "Serial interval",
y = "Frequency"
)`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.Warning: Removed 1820 rows containing non-finite outside the scale range
(`stat_bin()`).Функция get_pairwise() будет интерпретировать класс столбца, используемого для сравнения и соответствующим образом корректирует свой метод сравнения значений. Для чисел и дат (как в примере si выше), функция будет вычитать значения. При применении к столбцам, имеющим символы или категориальный характер, get_pairwise() будет соединять значения. Поскольку функция также допускает произвольную обработку (см. аргумент “f”), эти дискретные комбинации могут быть легко табулированы и проанализированы.
head(get_pairwise(epic, "gender"), n = 10) [1] "f -> m" NA "m -> m" NA "m -> f" "f -> f" NA "f -> m"
[9] NA "m -> f"get_pairwise(epic, "gender", f = table) values.to
values.from f m
f 464 516
m 510 468fisher.test(get_pairwise(epic, "gender", f = table))
Fisher's Exact Test for Count Data
data: get_pairwise(epic, "gender", f = table)
p-value = 0.03758
alternative hypothesis: true odds ratio is not equal to 1
95 percent confidence interval:
0.6882761 0.9892811
sample estimates:
odds ratio
0.8252575 Здесь мы видим значительную связь между цепочками передачи и полом.
Определение кластеров
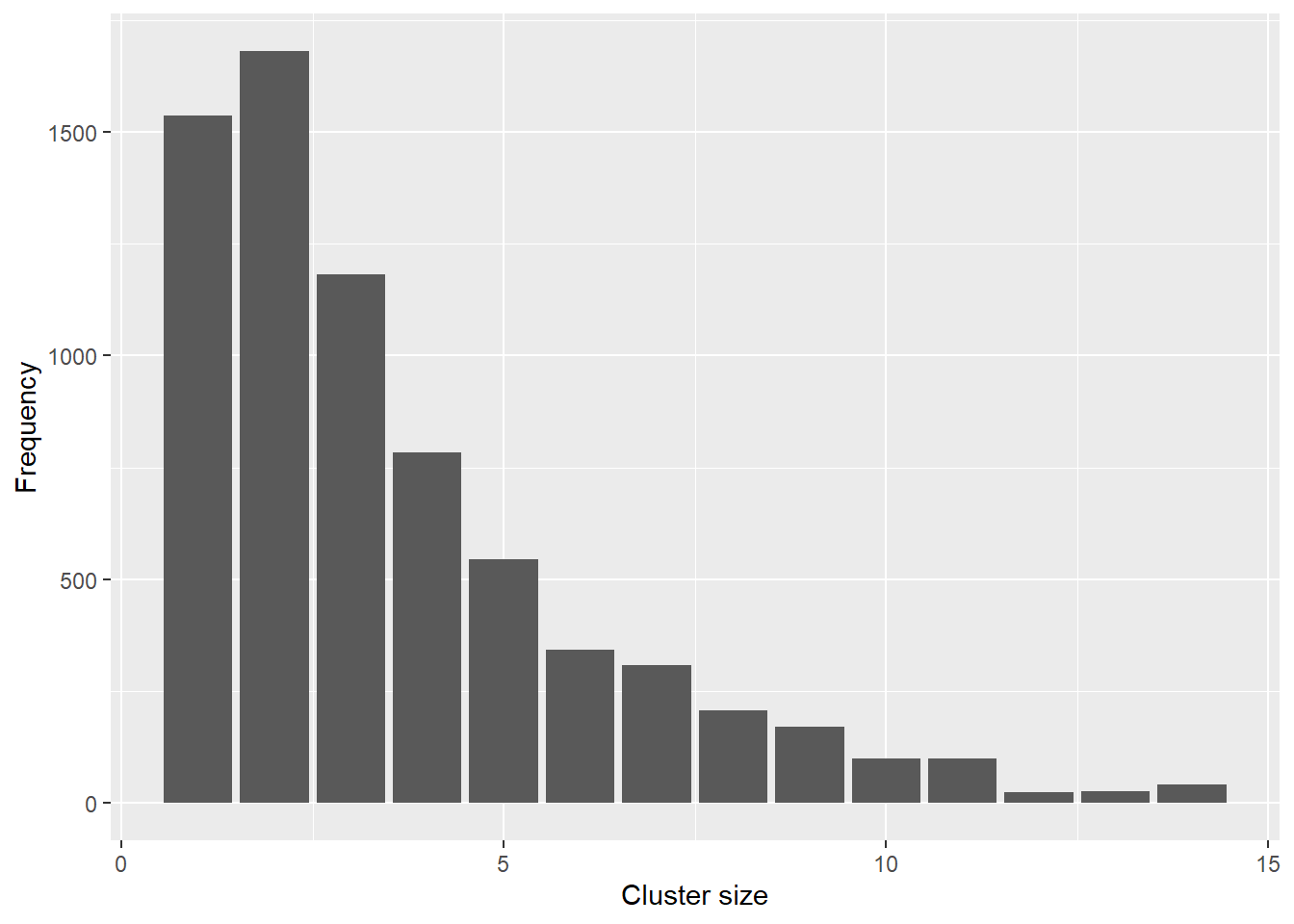
Функция get_clusters() может быть использована для определения связанных компонентов в объекте epicontacts. Сначала с ее помощью мы получаем датафрейм data.frame. содержащий информацию о кластерах:
clust <- get_clusters(epic, output = "data.frame")
table(clust$cluster_size)
1 2 3 4 5 6 7 8 9 10 11 12 13 14
1536 1680 1182 784 545 342 308 208 171 100 99 24 26 42 ggplot(clust, aes(cluster_size)) +
geom_bar() +
labs(
x = "Cluster size",
y = "Frequency"
)Рассмотрим наиболее крупные кластеры. Для этого мы добавляем информацию о кластере в объект epicontacts, а затем выделим в нем только самые крупные кластеры:
epic <- get_clusters(epic)
max_size <- max(epic$linelist$cluster_size)
plot(subset(epic, cs = max_size))Вычисление степени
Степень узла соответствует количеству его ребер или связей с другими узлами. get_degree() предоставляет простой метод вычисления этого значения для сети epicontacts. Высокая степень в данном контексте указывает на человека, который контактировал со многими другими людьми. Аргумент type указывает на то, что мы хотим считать как входящие, так и исходящие степени, аргумент only_linelist указывает на то, что мы хотим вычислить только степень для случаев в построчном списке.
deg_both <- get_degree(epic, type = "both", only_linelist = TRUE)Какие люди имеют десять наибольших контактов?
head(sort(deg_both, decreasing = TRUE), 10)916d0a 858426 6833d7 f093ea 11f8ea 3a4372 38fc71 c8c4d5 a127a7 02d8fd
7 6 6 6 5 5 5 5 5 5 Каково среднее количество контактов?
mean(deg_both)[1] 1.07847337.6 Ресурсы
Страница epicontacts содержит обзор функций пакета и несколько более подробных виньеток.
Страница github может быть использована для поднятия проблем и запросов на добавление новых возможностей.