32 Эпидемические кривые
Эпидемическая кривая (также известная как “эпидкривая”) - это основной эпидемиологический график, обычно используемый для визуализации временной структуры возникновения заболевания среди кластера или эпидемии случаев.
Анализ эпидкривой позволяет выявить временные тенденции, выпадающие значения, масштабы вспышки, наиболее вероятный период заражения, временные интервалы между поколениями заболевших и даже определить способ передачи неустановленного заболевания (например, точечный источник, непрерывный общий источник, распространение от человека к человеку). Один из онлайн-уроков по интерпретации эпидкривых можно найти на сайте CDC США.
На этой странице мы демонстрируем два подхода к построению эпидкривых в R:
- Пакет incidence2, который позволяет получить кривую эпидкривую с помощью простых команд
- Пакет ggplot2, который позволяет расширить возможности настройки с помощью более сложных команд
Также рассматриваются конкретные примеры использования, такие как:
- Построение графиков агрегированных данных о подсчетах.
- Построение фасетов или создание малых множеств
- Применение скользящих средних
- Отображение данных, которые являются “предварительными” или подвергаются задержкам в отчетности
- Наложение кумулятивной заболеваемости с помощью второй оси
32.1 Подготовка
Пакеты
В этом фрагменте кода показана загрузка пакетов, необходимых для проведения анализа. В данном руководстве мы делаем акцент на функции p_load() из pacman, которая при необходимости устанавливает пакет и загружает его для использования. Установленные пакеты можно также загрузить с помощью library() из базового R. Более подробную информацию о пакетах R см. на странице [Основы R].
pacman::p_load(
rio, # импорт/экспорт файлов
here, # относительные пути файлов
lubridate, # работа с датами/эпинеделями
aweek, # альтернативный пакет для работы с датами/эпидемиями
incidence2, # эпидкривые данных построчного списка
i2extras, # дополнение к заболеваемости2
stringr, # поиск и работа с последовательностью символов
forcats, # работа с факторами
RColorBrewer, # Цветовые палитры с сайта colorbrewer2.org
tidyverse # управление данными + графика ggplot2
) Импорт данных
В данном разделе используются два примера наборов данных:
- Построчный список отдельных случаев из смоделированной эпидемии
- Агрегированные подсчеты по больницам из той же смоделированной эпидемии.
Наборы данных импортируются с помощью функции import() из пакета rio. Различные способы импорта данных см. на странице [Импорт и экспорт].
Построчный список случаев
Мы импортируем набор данных о случаях заболевания из смоделированной эпидемии лихорадки Эбола. Если вы хотите загрузить данные, чтобы проследить за ходом работы, смотрите инструкцию на странице [Скачивание руководства и данных]. Мы предполагаем, что файл находится в рабочей директории, поэтому в пути к файлу не указываются вложенные папки.
linelist <- import("linelist_cleaned.xlsx")Ниже отображаются первые 50 строк.
Подсчеты, агрегированные по больницам
В данном руководстве набор данных еженедельных агрегированных подсчетов случаев по больницам создается из построчного списка с помощью следующего кода.
# Импорт данных о подсчетах R
count_data <- linelist %>%
group_by(hospital, date_hospitalisation) %>%
summarize(n_cases = dplyr::n()) %>%
filter(date_hospitalisation > as.Date("2013-06-01")) %>%
ungroup()Ниже отображаются первые 50 строк:
Задать параметры
При формировании отчета может потребоваться задать редактируемые параметры, например, дату, для которой данные являются актуальными (“дата данных”). Затем на объект data_date можно ссылаться в коде при применении фильтров или в динамических подписях.
## установить дату отчета для отчета
## Примечание: может быть установлено значение Sys.Date() для текущей даты
data_date <- as.Date("2015-05-15")Проверить даты
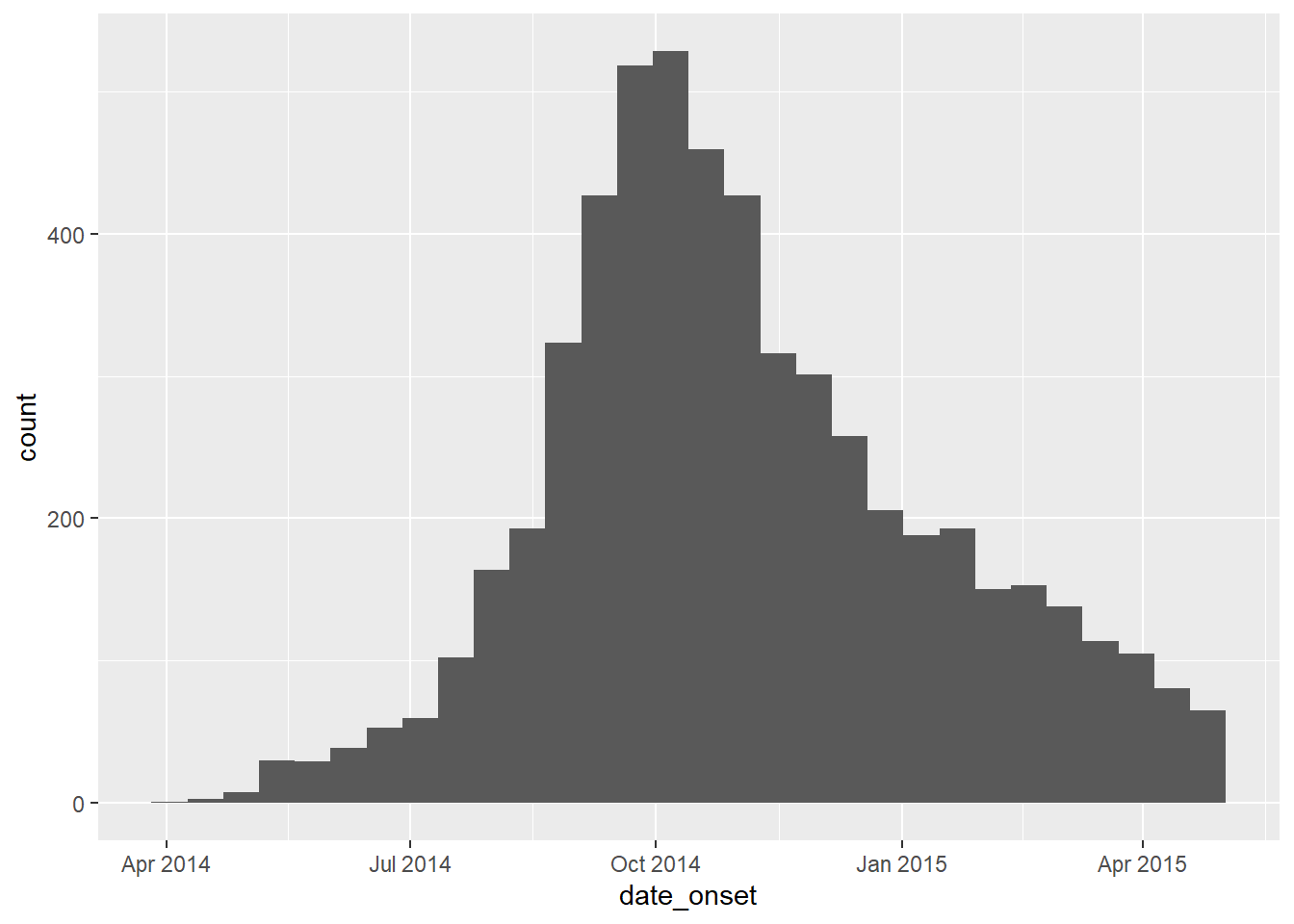
Убедитесь, что каждый соответствующий столбец даты относится к классу Дата и имеет соответствующий диапазон значений. Для этого можно просто использовать hist() для гистограмм, или range() с na.rm=TRUE, или ggplot(), как показано ниже.
32.2 эпидкривые с помощью ggplot2
Использование ggplot() для построения эпидкривой позволяет добиться большей гибкости и настраиваемости, но требует больших усилий и понимания принципов работы ggplot().
В отличие от использования пакета incidence2, здесь необходимо вручную управлять агрегированием случаев по времени (по неделям, месяцам и т.д.) и интервалами меток на оси дат. Это требует тщательного контроля.
В этих примерах используется подмножество построчного списка данных - только случаи из Центральной больницы.
central_data <- linelist %>%
filter(hospital == "Central Hospital")Для получения эпидкривой с помощью ggplot() необходимо три основных элемента:
- Гистограмма, в которой случаи из построчного списка объединены в “корзины”, отличающиеся определенными точками ” излома”.
- Шкалы для осей и их метки
- Темы оформления графика, включая заголовки, метки, подписи и т.д.
Укажите корзины для случаев
Ниже мы покажем, как указать, каким образом случаи будут агрегироваться в корзины гистограммы (“столбики”). Важно понимать, что объединение случаев в корзины гистограммы не обязательно совпадает с интервалами дат, которые будут отображаться на оси x.
Далее приведен, пожалуй, наиболее простой код для получения ежедневных и еженедельных эпидкривых.
Во всеохватывающей команде ggplot() набор данных передается в data =. На эту основу с помощью знака + добавляется геометрия гистограммы. В команде geom_histogram() мы задаем эстетику таким образом, что столбец date_onset отображается на ось x. Также внутри geom_histogram(), но не внутри aes(), мы устанавливаем binwidth = корзины гистограммы, в днях. Если этот синтаксис ggplot2 вызывает недоумение, просмотрите страницу [Основы ggplot].
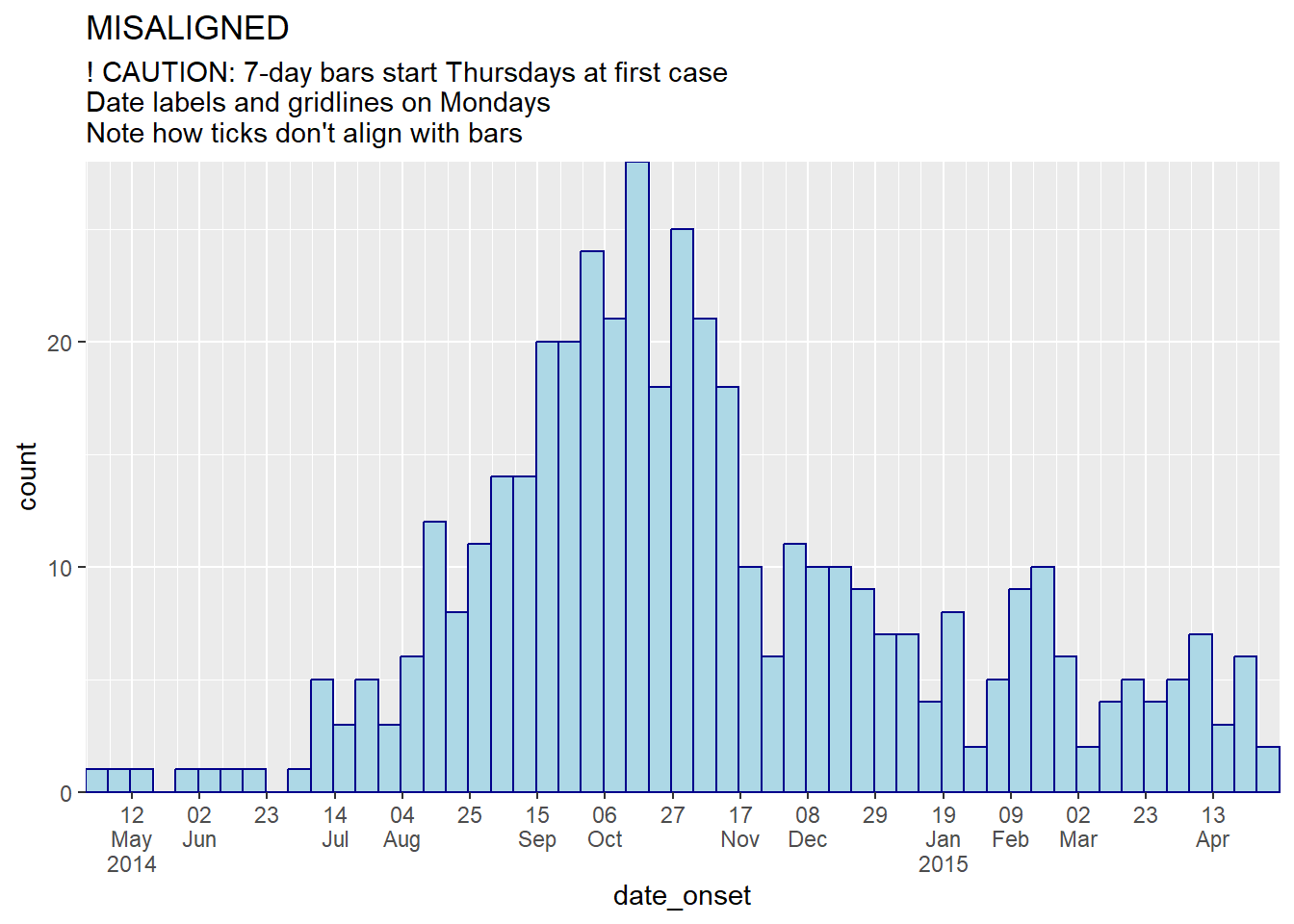
ВНИМАНИЕ: Построение недельных случаев с использованием binwidth = 7 начинает первую 7-дневную корзину с первого случая, который может быть любым днем недели! Для создания конкретных недель см. раздел ниже .
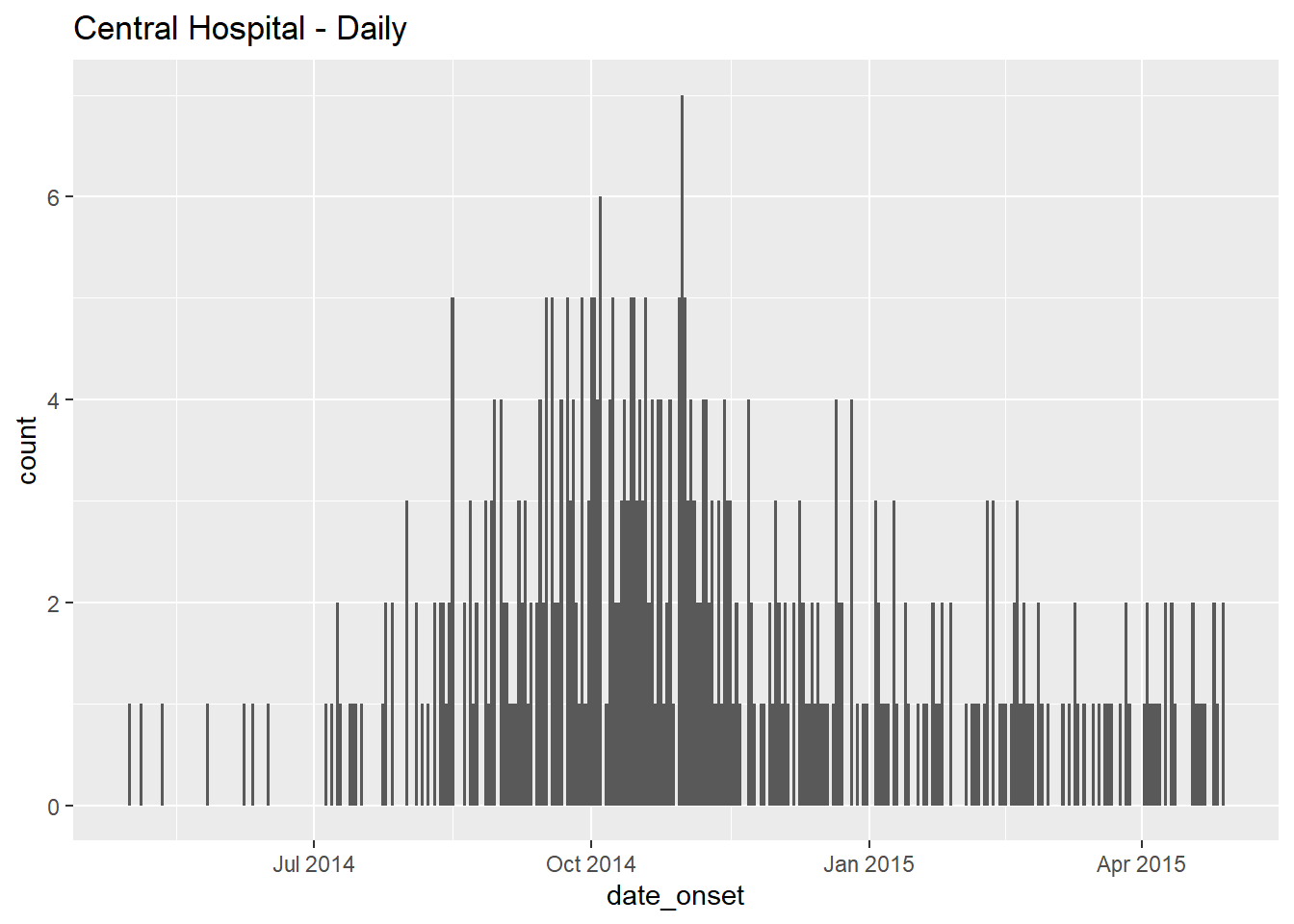
# ежедневно
ggplot(data = central_data) + # задать данные
geom_histogram( # добавить гистограмму
mapping = aes(x = date_onset), # сопоставить столбец даты с осью x
binwidth = 1)+ # случаи, разделенные на корзины по 1 дню
labs(title = "Central Hospital - Daily") # заголовок
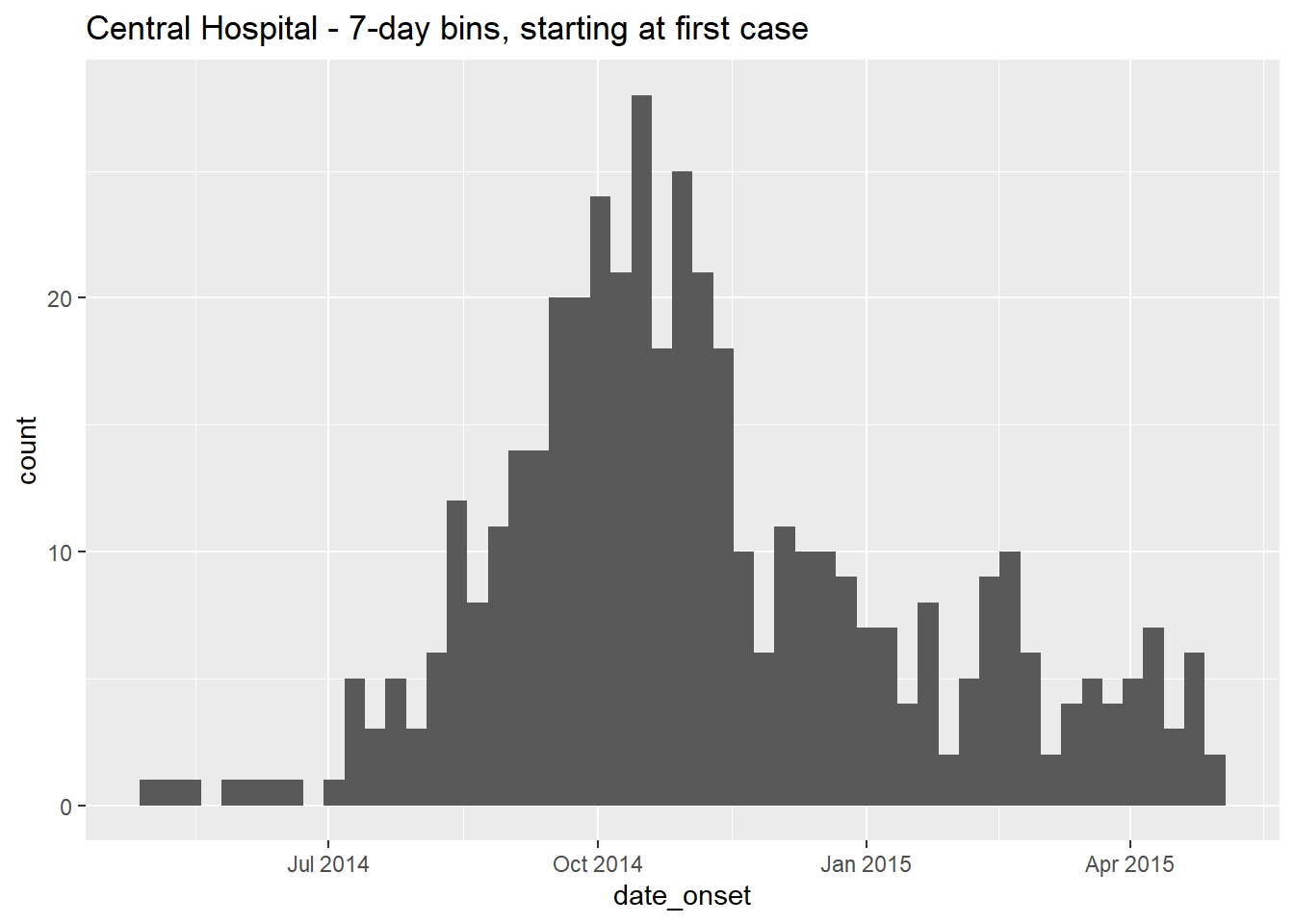
# еженедельно
ggplot(data = central_data) + # задать данные
geom_histogram( # добавить гистограмму
mapping = aes(x = date_onset), # сопоставить столбец даты с осью x
binwidth = 7)+ # случаи, разделенные на корзины каждые 7 дней, начиная с первого случая (!)
labs(title = "Central Hospital - 7-day bins, starting at first case") # titleОтметим, что первый случай в наборе данных Central Hospital имел дату возникновения симптомов:
format(min(central_data$date_onset, na.rm=T), "%A %d %b, %Y")[1] "Thursday 01 May, 2014"Чтобы вручную указать корзины гистограммы, не используйте аргумент binwidth =, а вместо этого передайте вектор дат в breaks =..
Создайте вектор дат с помощью базовой функции R seq.Date(). Эта функция принимает аргументы to =, from = и by =. Например, приведенная ниже команда возвращает даты за месяц, начиная с 15 января и заканчивая 28 июня.
monthly_breaks <- seq.Date(from = as.Date("2014-02-01"),
to = as.Date("2015-07-15"),
by = "months")
monthly_breaks # печать [1] "2014-02-01" "2014-03-01" "2014-04-01" "2014-05-01" "2014-06-01"
[6] "2014-07-01" "2014-08-01" "2014-09-01" "2014-10-01" "2014-11-01"
[11] "2014-12-01" "2015-01-01" "2015-02-01" "2015-03-01" "2015-04-01"
[16] "2015-05-01" "2015-06-01" "2015-07-01"Этот вектор может быть передан в geom_histogram() в виде breaks =:
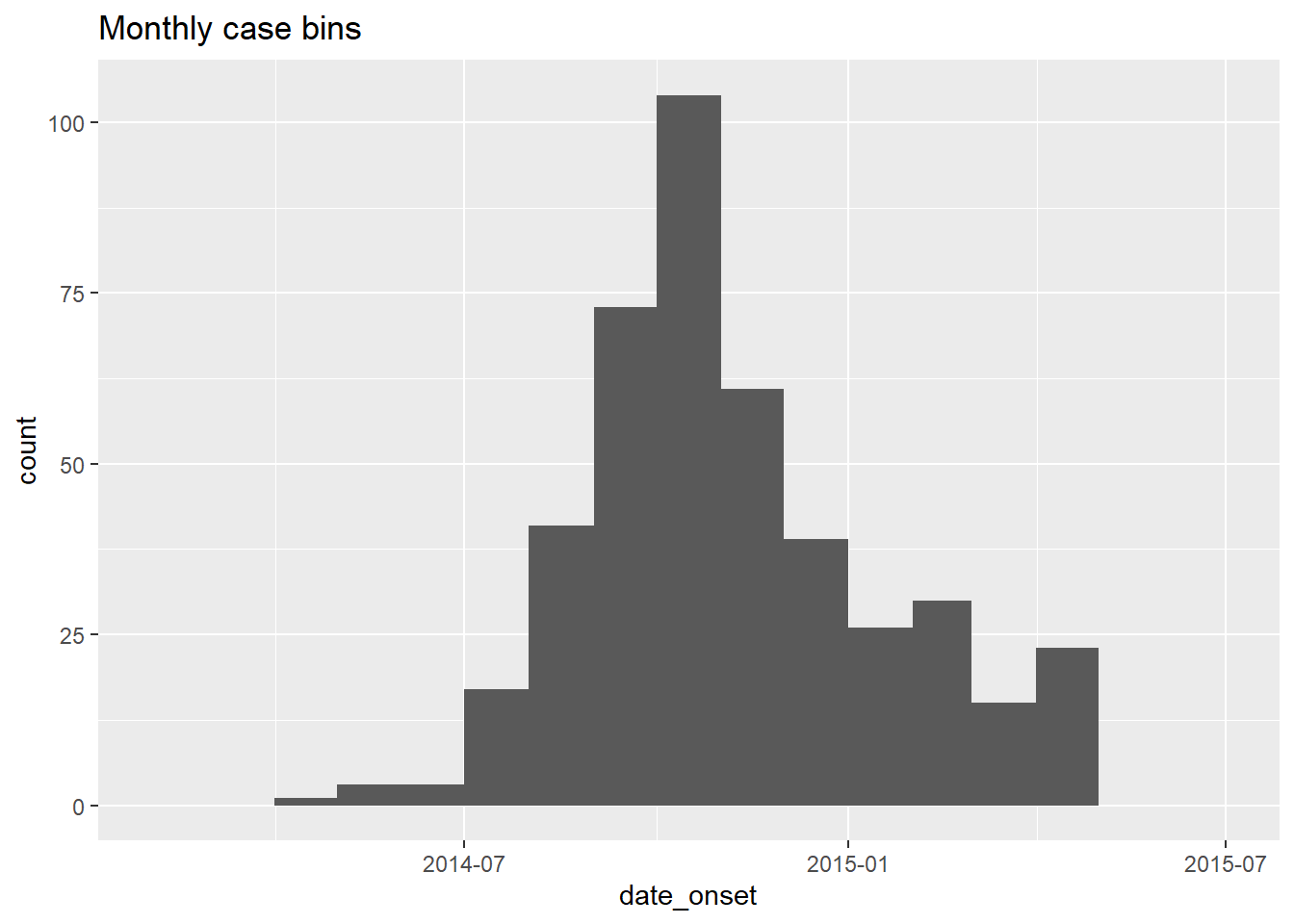
# ежемесячно
ggplot(data = central_data) +
geom_histogram(
mapping = aes(x = date_onset),
breaks = monthly_breaks)+ # обеспечить заданный вектор разрывов
labs(title = "Monthly case bins") # заголовокПростую недельную последовательность дат можно вернуть, задав by = "week". Например:
weekly_breaks <- seq.Date(from = as.Date("2014-02-01"),
to = as.Date("2015-07-15"),
by = "week")Альтернативой заданию конкретных дат начала и конца является написание динамического кода, в котором недельные интервалы начинаются с понедельника, предшествующего первому случаю. Мы будем использовать эти векторы дат во всех приведенных ниже примерах.
# Последовательность еженедельных дат понедельника для ЦЕНТРАЛЬНОЙ БОЛЬНИЦЫ
weekly_breaks_central <- seq.Date(
from = floor_date(min(central_data$date_onset, na.rm=T), "week", week_start = 1), # понедельник, предшествующий первому случаю
to = ceiling_date(max(central_data$date_onset, na.rm=T), "week", week_start = 1), # понедельник после последнего случая
by = "week")Давайте разберем довольно сложный код, приведенный выше:
- Значение ” с” (самая ранняя дата последовательности) создается следующим образом: минимальное значение даты (
min()сna.rm=TRUE) в столбцеdate_onsetподается наfloor_date()из пакета lubridate.floor_date(), установленная в значение ” неделя”, возвращает дату начала “недели” этого случая, учитывая, что день начала каждой недели - понедельник (week_start = 1).
- Аналогично, значение ” до” (дата окончания последовательности) создается с помощью обратной функции
ceiling_date()для возврата понедельника, следующего за последним случаем.
- Аргумент ” по” функции
seq.Date()может быть установлен на любое количество дней, недель или месяцев.
- Используйте
week_start = 7для недели, гачигающейся с воскресенья.
Поскольку мы будем использовать эти векторы дат на протяжении всей страницы, мы также определим один из них для всей вспышки (выше приведен только для Центральной больницы).
# Последовательность для всей вспышки
weekly_breaks_all <- seq.Date(
from = floor_date(min(linelist$date_onset, na.rm=T), "week", week_start = 1), # понедельник, предшествующий первому случаю
to = ceiling_date(max(linelist$date_onset, na.rm=T), "week", week_start = 1), # понедельник после последнего случая
by = "week")Такие результаты seq.Date() могут быть использованы для создания разрывов между корзинами гистограммы, а также разрывов для меток даты, которые могут быть независимыми от корзин. Подробнее о метках дат читайте в последующих разделах.
TIP: Для более простой команды ggplot() заранее сохраните разрывы между корзинами и разрывы меток даты в виде именованных векторов и просто укажите их названия в breaks =.
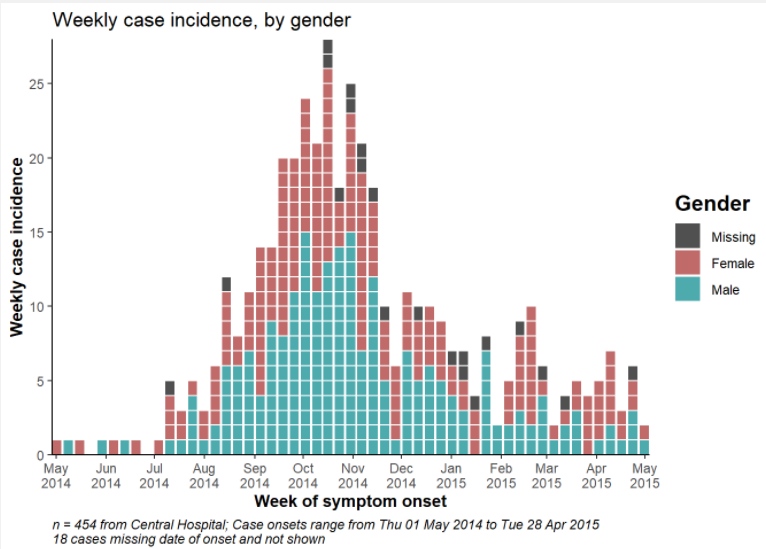
Пример недельной эпидкривой
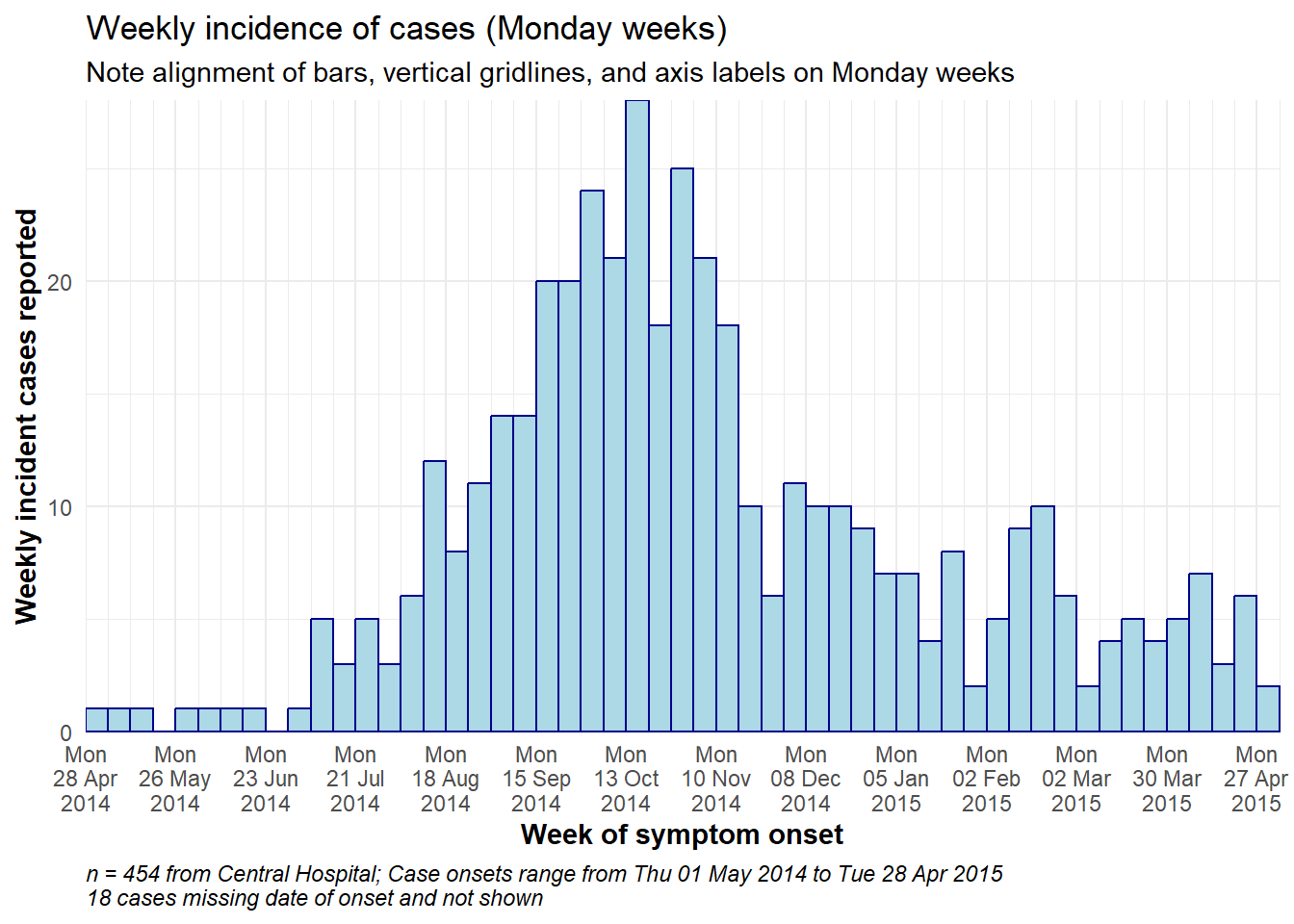
Ниже приведен подробный пример кода для создания еженедельных эпидкривых для недели, начинающейся в понедельник, с выровненными столбцами, метками даты и вертикальными линиями сетки. Этот раздел предназначен для пользователя, которому код нужен быстро. Для более глубокого понимания каждого аспекта (темы, метки даты и т.д.) перейдите к последующим разделам. Примечание:
- Разрывы между корзинами гистограммы* задаются с помощью
seq.Date(), как описано выше, чтобы начинаться в понедельник перед самым ранним случаем и заканчиваться в понедельник после последнего случая.
- Интервал между метками даты задается с помощью
date_breaks =вscale_x_date().
- Интервал мелких вертикальных линий сетки между метками даты задается с помощью
date_minor_breaks =.
- Мы используем
closed = "left"вgeom_histogram(), чтобы обеспечить подсчет дат в правильных корзинах
-
expand = c(0,0)в шкалах x и y удаляет лишнее пространство по обе стороны от осей, что также обеспечивает начало отсчета меток дат с первого столбика.
# ОБЩЕЕ ВЫРАВНИВАНИЕ НЕДЕЛИ С ПОНЕДЕЛЬНИКА
#############################
# Определить последовательность еженедельных разрывов
weekly_breaks_central <- seq.Date(
from = floor_date(min(central_data$date_onset, na.rm=T), "week", week_start = 1), # Понедельник перед первым случаем
to = ceiling_date(max(central_data$date_onset, na.rm=T), "week", week_start = 1), # Понедельник после последнего случая
by = "week") # bins are 7-days
ggplot(data = central_data) +
# построить гистограмму: указать точки разрыва между корзинами: начало - понедельник перед первым случаем, конец - понедельник после последнего случая
geom_histogram(
# сопоставление эстетики
mapping = aes(x = date_onset), # столбец даты сопоставлен с осью x
# разрывы между корзинами гистограммы
breaks = weekly_breaks_central, # разрывы между корзинами гистограммы, заданные ранее
closed = "left", # подсчет случаев от начала точки прерывания
# столбики
color = "darkblue", # цвет линий вокруг столбиков
fill = "lightblue" # цвет заливки внутри столбиков
)+
# метки оси x
scale_x_date(
expand = c(0,0), # удалить лишнее пространство по оси x до и после столбиков случая
date_breaks = "4 weeks", # метки даты и основные вертикальные линии сетки появляются каждые 3 недели понедельника
date_minor_breaks = "week", # Незначительные вертикальные линии появляются каждую неделю понедельника
date_labels = "%a\n%d %b\n%Y")+ # формат меток даты
# ось y
scale_y_continuous(
expand = c(0,0))+ # удалить лишнее пространство по оси y ниже 0 (выровнять гистограмму вровень с осью x)
# темы эстетики
theme_minimal()+ # упростить фон графика
theme(
plot.caption = element_text(hjust = 0, # надпись с левой стороны
face = "italic"), # надпись курсивом
axis.title = element_text(face = "bold"))+ # заголовки осей выделены жирным шрифтом
# метки, включая динамические надписи
labs(
title = "Weekly incidence of cases (Monday weeks)",
subtitle = "Note alignment of bars, vertical gridlines, and axis labels on Monday weeks",
x = "Week of symptom onset",
y = "Weekly incident cases reported",
caption = stringr::str_glue("n = {nrow(central_data)} from Central Hospital; Case onsets range from {format(min(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')} to {format(max(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')}\n{nrow(central_data %>% filter(is.na(date_onset)))} cases missing date of onset and not shown"))Недели с воскресенья
Для получения приведенного выше графика для недель с воскресенья необходимо внести некоторые изменения, поскольку date_breaks = "weeks" работает только для недель с понедельника.
- Точки разрыва корзин гистограммы должны быть установлены на воскресенье (
week_start = 7)
- В функции
scale_x_date()следует задать аналогичные разрывы датbreaks =иminor_breaks =, чтобы метки дат и вертикальные линии сетки выравнивались по воскресеньям.
Например, команда scale_x_date() для недель с воскресенья может выглядеть следующим образом:
scale_x_date(
expand = c(0,0),
# задать интервал между метками даты и основными вертикальными линиями сетки
breaks = seq.Date(
from = floor_date(min(central_data$date_onset, na.rm=T), "week", week_start = 7), # Воскресенье, предшествующее первому случаю
to = ceiling_date(max(central_data$date_onset, na.rm=T), "week", week_start = 7), # Воскресенье после последнего случая
by = "4 weeks"),
# задать интервал малой вертикальной линии сетки
minor_breaks = seq.Date(
from = floor_date(min(central_data$date_onset, na.rm=T), "week", week_start = 7), # Воскресенье, предшествующее первому случаю
to = ceiling_date(max(central_data$date_onset, na.rm=T), "week", week_start = 7), # Воскресенье после последнего случая
by = "week"),
# формат метки даты
#date_labels = "%a\n%d %b\n%Y")+ # день, над аббревиатурой месяца, над двузначным годом
label = scales::label_date_short())+ # автоматическое форматирование метокГруппировка/окрашивание по значению
Столбцы гистограммы могут быть окрашены по группам и “сложены”. Чтобы назначить столбец группировки, выполните следующие изменения. Подробности см. на странице [Основы ggplot].
- Внутри эстетического отображения гистограммы
aes()сопоставьте название столбца с аргументамиgroup =иfill =.
- Удалите любой аргумент
fill =, находящийся внеaes(), так как он будет замещать тот, который находится внутри.
- Аргументы внутри
aes()будут применяться по группе, в то время как аргументы снаружи будут применяться ко всем столбикам (например, вы все еще можете захотеть использоватьcolor =снаружи, чтобы каждый столбик имел одинаковую границу)
Вот как будет выглядеть команда aes() для группировки и окрашивания столбиков по полу:
aes(x = date_onset, group = gender, fill = gender)В данном случае она применяется:
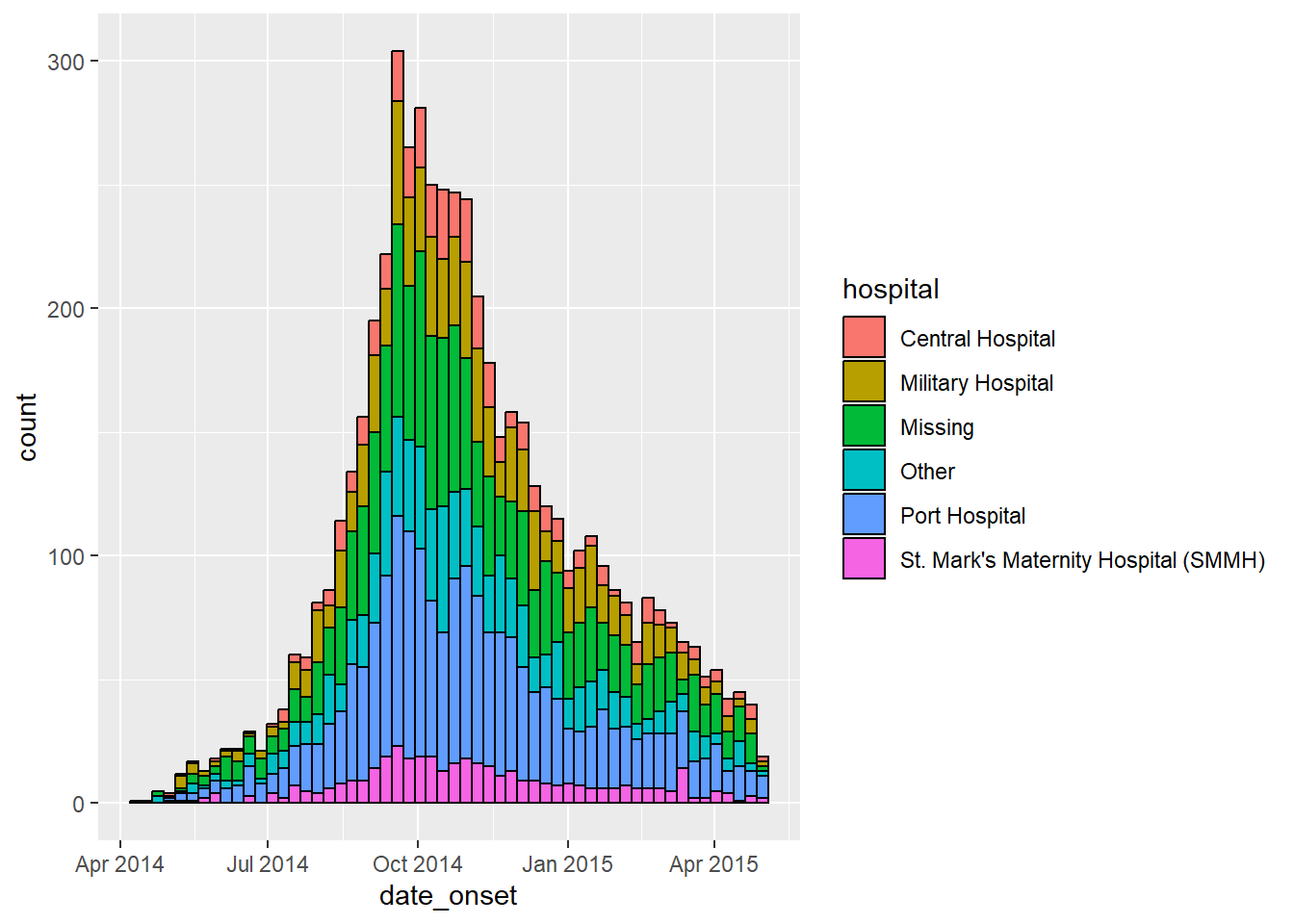
ggplot(data = linelist) + # начать с построчного списка (много больниц)
# построить гистограмму: указать точки разрыва корзин: начало - понедельник перед первым случаем, конец - понедельник после последнего случая
geom_histogram(
mapping = aes(
x = date_onset,
group = hospital, # задать группировку данных по больницам
fill = hospital), # заливка столбиков (внутренний цвет) по больницам
# Разрывы между корзинами - недели с понедельника
breaks = weekly_breaks_all, # последовательность разрывов между корзинами неделей с понедельника для всей вспышки, определенная в предыдущем коде
closed = "left", # подсчет случаев от начала точки прерывания
# Цвет вокруг столбиков
color = "black")Настройка цвета
- Для ручной настройки заливки для каждой группы используйте
scale_fill_manual()(обратите внимание:scale_color_manual()отличается!).- Используйте аргумент
values =для применения вектора цветов.
- Используйте аргумент
na.value =, чтобы задать цвет для значенийNA.
- Используйте аргумент
labels =для изменения текста элементов легенды. Для надежности задайте вектор с названием типаc("old" = "new", "old" = "new")или скорректируйте значения в самих данных.
- Используйте аргумент
name =, чтобы дать легенде соответствующее название
- Используйте аргумент
- Дополнительные сведения о цветовых шкалах и палитрах см. на странице [Основы ggplot].
ggplot(data = linelist)+ # начать с построчного списка (много больниц)
# построить гистограмму
geom_histogram(
mapping = aes(x = date_onset,
group = hospital, # случаи, сгруппированные по больницам
fill = hospital), # заливка столбиков по больницам
# разрывы кмежду корзин
breaks = weekly_breaks_all, # последовательность разрывов между корзинами неделей с понедельника для всей вспышки, определенная в предыдущем коде
closed = "left", # подсчет случаев от начала точки прерывания
# Цвет вокруг столбиков
color = "black")+ # цвет границы каждого столбика
# ручная спецификация цвета
scale_fill_manual(
values = c("black", "orange", "grey", "beige", "blue", "brown"),
labels = c("St. Mark's Maternity Hospital (SMMH)" = "St. Mark's"),
name = "Hospital") # укажите цвета заливки ("значения") - внимание на порядок!Настройка порядка уровней
Порядок укладки сгруппированных столбцов лучше всего настроить, отнеся столбец группировки к классу Фактор. Затем можно задать порядок уровней факторов (и их отображаемые метки). Подробности см. на странице [Факторы] или [Советы по использованию ggplot].
Перед построением графика воспользуйтесь функцией fct_relevel() из пакета forcats, чтобы преобразовать столбец группировки в класс Фактор и вручную настроить порядок уровней, как описано на странице [Факторы].
# загрузить пакет forcats для работы с факторами
pacman::p_load(forcats)
# Определить новый набор данных с больницей в качестве фактора
plot_data <- linelist %>%
mutate(hospital = fct_relevel(hospital, c("Missing", "Other"))) # Преобразовать в фактор и установить "Отсутствует" и "Другое" в качестве верхних уровней для отображения на эпидкривой сверху
levels(plot_data$hospital) # печать уровней по порядку[1] "Missing"
[2] "Other"
[3] "Central Hospital"
[4] "Military Hospital"
[5] "Port Hospital"
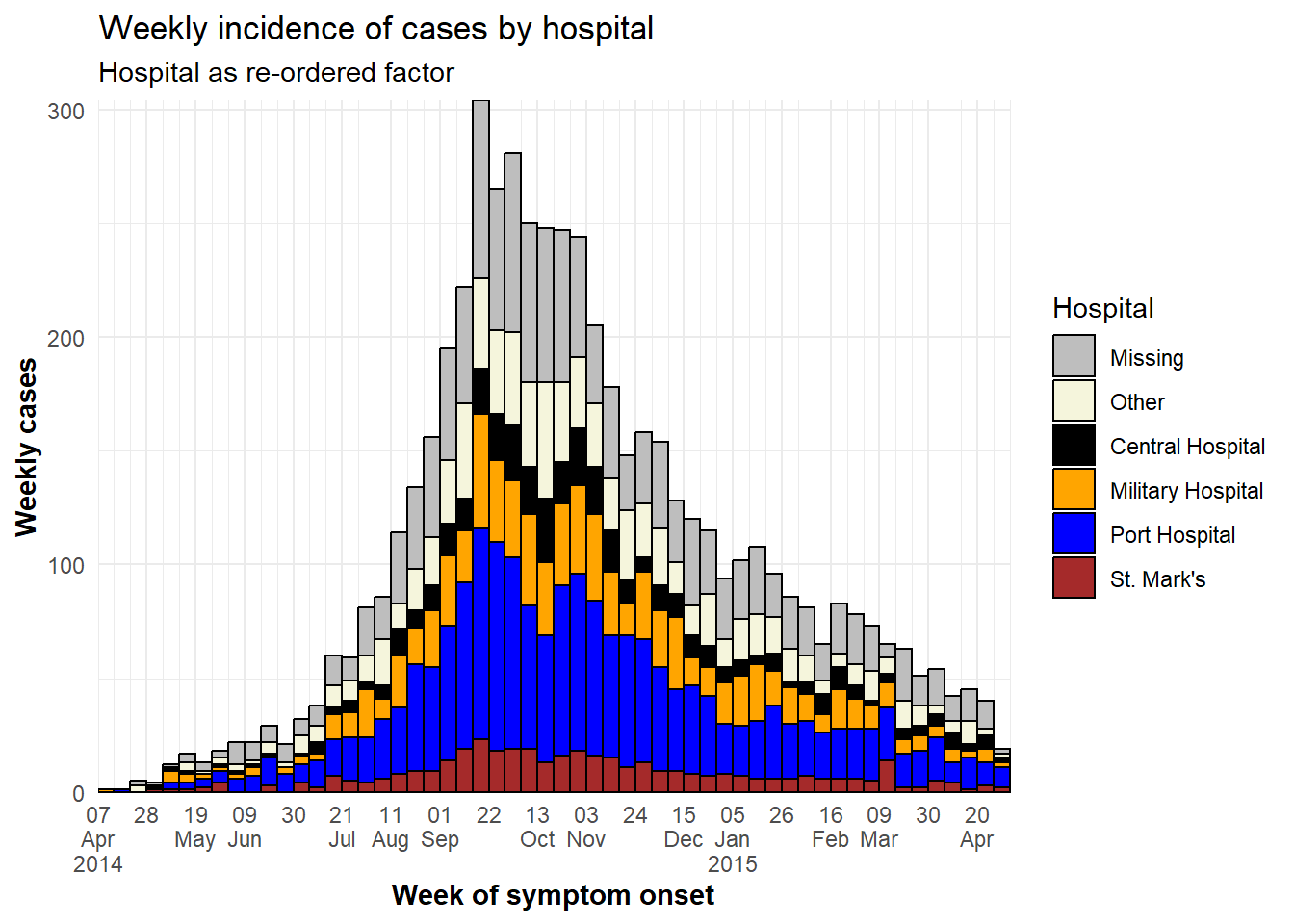
[6] "St. Mark's Maternity Hospital (SMMH)"На приведенном ниже графике единственным отличием от предыдущего является то, что столбец hospital был объединен, как указано выше, и мы используем guides() для изменения порядка легенды, так что ““Отсутствует”” находится в нижней части легенды.
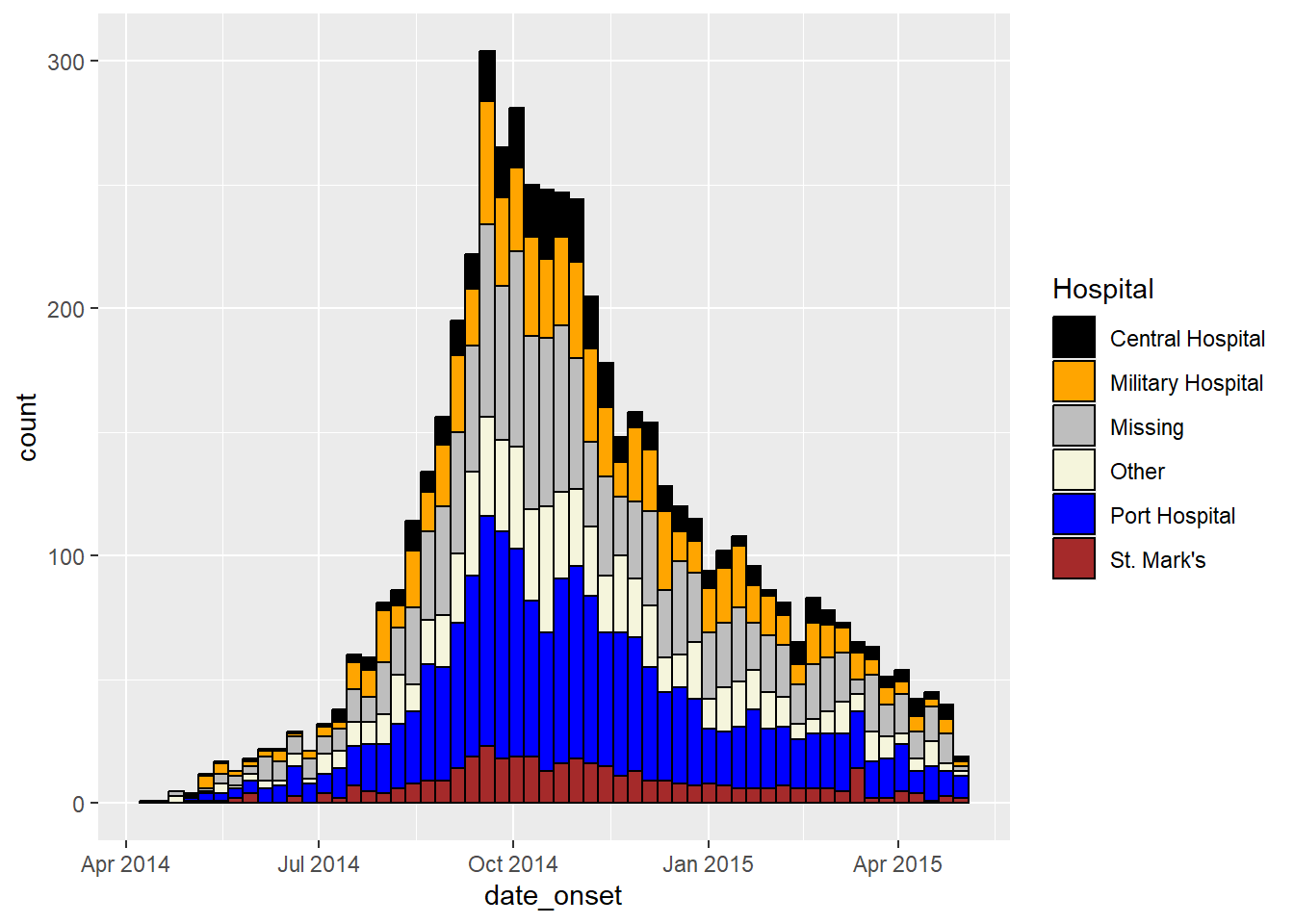
ggplot(plot_data) + # Использовать НОВЫЙ набор данных с больницей в качестве переупорядоченного фактора
# построить гистограмму
geom_histogram(
mapping = aes(x = date_onset,
group = hospital, # случаи, сгруппированные по больницам
fill = hospital), # Заливка столбиков (цвет) по больницам
breaks = weekly_breaks_all, # последовательность разрывов между корзинами неделей с понедельника для всей вспышки, определенная в верхней части раздела ggplot
closed = "left", # подсчет случаев от начала точки прерывания
color = "black")+ # цвет границы вокруг каждого столбика
# метки оси x
scale_x_date(
expand = c(0,0), # удалить лишнее пространство по оси x до и после столбиков случая
date_breaks = "3 weeks", # метки появляются каждые 3 недели с понедельника
date_minor_breaks = "week", # вертикальные линии появляются каждую неделю с понедельника
label = scales::label_date_short()) + # удобное форматирование меток
# ось y
scale_y_continuous(
expand = c(0,0))+ # удалить лишнее пространство по оси y ниже 0
# ручная спецификация цветов, внимание к порядку!
scale_fill_manual(
values = c("grey", "beige", "black", "orange", "blue", "brown"),
labels = c("St. Mark's Maternity Hospital (SMMH)" = "St. Mark's"),
name = "Hospital")+
# темы эстетики
theme_minimal()+ # упростить фон графика
theme(
plot.caption = element_text(face = "italic", # надпись слева курсивом
hjust = 0),
axis.title = element_text(face = "bold"))+ # заголовки осей выделены жирным шрифтом
# метки
labs(
title = "Weekly incidence of cases by hospital",
subtitle = "Hospital as re-ordered factor",
x = "Week of symptom onset",
y = "Weekly cases")СОВЕТ: Чтобы изменить порядок только легенды, добавьте следующую команду ggplot2: guides(fill = guide_legend(reverse = TRUE)).
Настройка легенды
Подробнее о легендах и шкалах можно прочитать на странице [Советы по использованию ggplot]. Вот несколько основных моментов:
- Редактирование заголовка легенды либо в функции шкалы, либо с помощью
labs(fill = "Заголовок легенды")(если вы используетеcolor =aesthetic, то используйтеlabs(color = ""))
-
theme(legend.title = element_blank())для того, чтобы не иметь заголовка легенды
-
theme(legend.position = "top")(” низ”, “лево”, “право” или “нет” для удаления легенды) -
theme(legend.direction = "horizontal")горизонтальная легенда - `
guides(fill = guide_legend(reverse = TRUE))для обратного порядка легенды
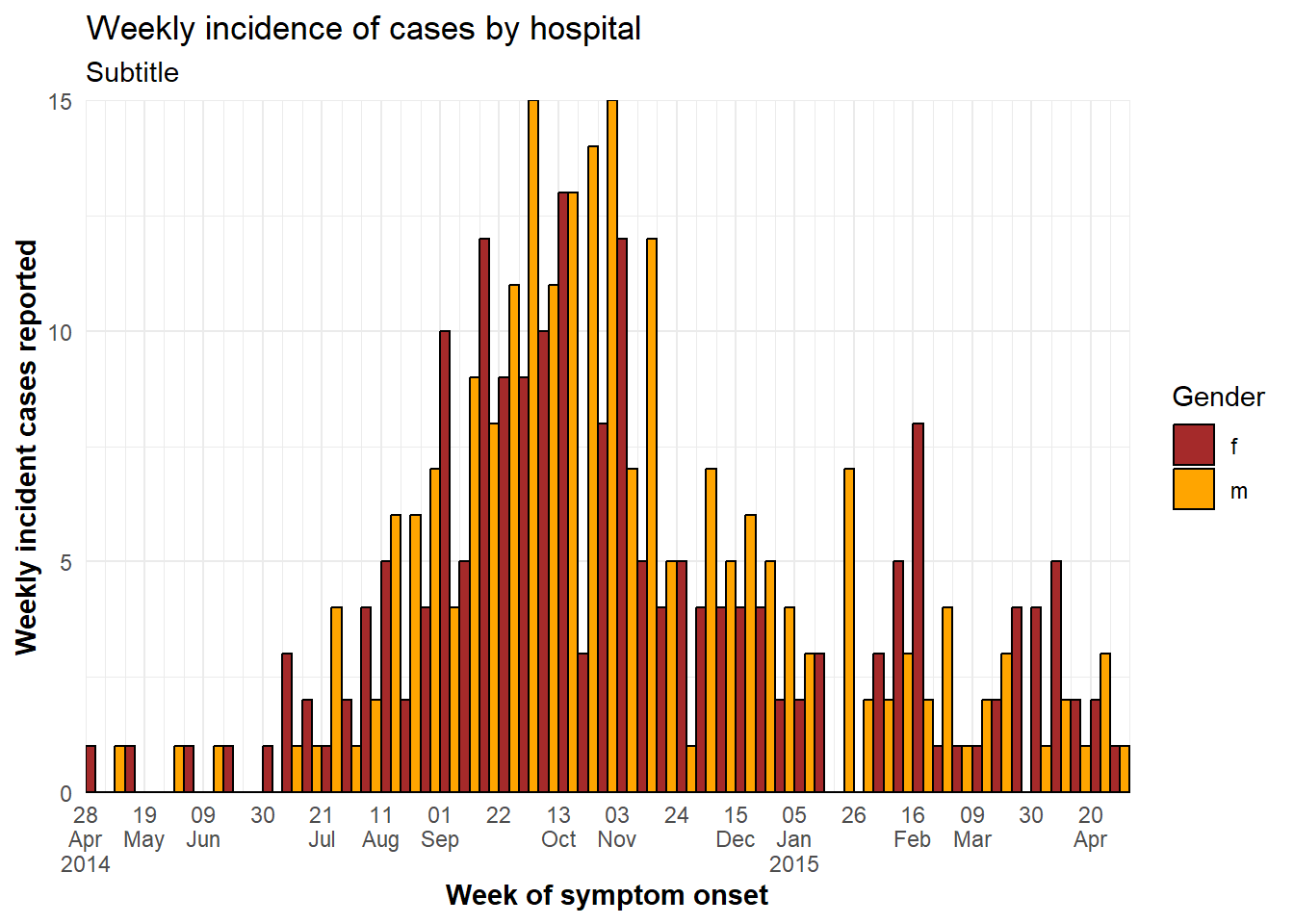
Столбики рядом
Отображение столбиков групп “бок о бок” (в отличие от сложенных) задается в geom_histogram() с position = "dodge", размещенной вне aes().
При наличии более двух групп значений они могут стать трудночитаемыми. Вместо этого можно использовать фасетный график (“малые множества”). Для улучшения читаемости в данном примере удалены недостающие значения пола.
ggplot(central_data %>% drop_na(gender))+ # начать со случаев в Центральной больнице, в которых отсутствует пол
geom_histogram(
mapping = aes(
x = date_onset,
group = gender, # случаи, сгруппированные по полу
fill = gender), # столбики, заполненные по полу
# разрывы между корзин гистограмм
breaks = weekly_breaks_central, # последовательность недельных дат для вспышки в Центральной больнице - определена в верхней части раздела ggplot
closed = "left", # подсчет случаев от начала точки прерывания
color = "black", # цвет кромки столбика
position = "dodge")+ # столбики бок о бок
# метки на шкале x
scale_x_date(expand = c(0,0), # удалить лишнее пространство по оси x ниже и после столбиков случаев
date_breaks = "3 weeks", # метки появляются каждые 3 недели с понедельника
date_minor_breaks = "week", # вертикальные линии появляются каждую неделю с понедельника
label = scales::label_date_short())+ # удобные метки даты
# ось y
scale_y_continuous(expand = c(0,0))+ # удаляет лишнее пространство по оси y между нижней частью столбиков и метками
#шкала цветов и меток легенды
scale_fill_manual(values = c("brown", "orange"), # укажите цвета заливки ("значения") - внимание на порядок!
na.value = "grey" )+
# темы эстетики
theme_minimal()+ # набор тем для упрощения графика
theme(plot.caption = element_text(face = "italic", hjust = 0), # надпись слева курсивом
axis.title = element_text(face = "bold"))+ # заголовки осей выделены жирным шрифтом
# метки
labs(title = "Weekly incidence of cases by hospital",
subtitle = "Subtitle",
fill = "Gender", # указать новый заголовок легенды
x = "Week of symptom onset",
y = "Weekly incident cases reported")Границы осей
Существует два способа ограничения размаха значений оси.
Как правило, предпочтительным является использование команды coord_cartesian(), которая принимает значения xlim = c(min, max) и ylim = c(min, max) (где вы указываете минимальное и максимальное значения). Это действует как ” увеличение масштаба” без фактического удаления данных, что важно для статистики и суммарных показателей.
В качестве альтернативы можно задать максимальное и минимальное значения даты, используя limits = c() внутри scale_x_date(). Например:
scale_x_date(limits = c(as.Date("2014-04-01"), NA)) # задает минимальную дату, а максимальную оставляет открытой. Аналогично, если необходимо, чтобы ось x простиралась до определенной даты (например, текущей), даже если новые случаи не были зарегистрированы, можно воспользоваться функцией:
scale_x_date(limits = c(NA, Sys.Date()) # обеспечивает продление оси дат до текущей даты ВНИМАНИЕ: Будьте внимательны, задавая перерывы или границы шкалы оси y (например, от 0 до 30 на 5: seq(0, 30, 5)). Такие статические числа могут слишком резко оборвать ваш график, если данные изменятся и выйдут за предел!
Метки/сеточные линии по оси дат
СОВЕТ: Помните, что метки на оси дат не зависят от объединения данных в столбцы, но визуально может быть важно выровнять корзины, метки дат и вертикальные линии сетки.
Чтобы изменить метки даты и линии сетки, используйте scale_x_date() одним из этих способов:
-
Если корзины гистограммы - это дни, недели с понедельника, месяцы или годы:
- Используйте
date_breaks =для задания интервала между метками и основными линиями сетки (например, “день”, “неделя”, “3 недели”, “месяц” или “год”) - Используйте
date_minor_breaks =для указания интервала между вертикальными линиями сетки (между метками даты).
- Добавьте
expand = c(0,0), чтобы начать метки с первого столбца.
- Используйте
date_labels =для указания формата меток даты - см. советы на странице Даты (используйте\nдля новой строки)
- Используйте
-
Если корзины гистограммы представляют собой недели с воскресенья:
- Используйте
breaks =иminor_breaks =, предоставляя последовательность переносов даты для каждого из них. - Вы все еще можете использовать
date_labels =иexpand =для форматирования, как описано выше.
- Используйте
Некоторые примечания:
- Инструкции по созданию последовательности дат с помощью
seq.Date()см. в разделе “Открытие ggplot”.
- Советы по созданию меток дат см. здесь эта страница или на странице [Работа с датами].
Демонстрация
Ниже показаны графики, на которых корзины и метки графика/линии сетки выровнены или не выровнены:
# корзины 7 дней и метки понедельника
#############################
ggplot(central_data) +
geom_histogram(
mapping = aes(x = date_onset),
binwidth = 7, # корзины 7 дней, которые начинаются с первым случаем
color = "darkblue",
fill = "lightblue") +
scale_x_date(
expand = c(0,0), # удалить лишнее пространство по оси x ниже и после столбиков случаев
date_breaks = "3 weeks", # Понедельник каждые 3 недели
date_minor_breaks = "week", # недели с понедельника
label = scales::label_date_short())+ # автоматическое форматирование меток
scale_y_continuous(
expand = c(0,0))+ # удалить лишнее пространство под осью x, сделать все ровным
labs(
title = "MISALIGNED",
subtitle = "! CAUTION: 7-day bars start Thursdays at first case\nDate labels and gridlines on Mondays\nNote how ticks don't align with bars")
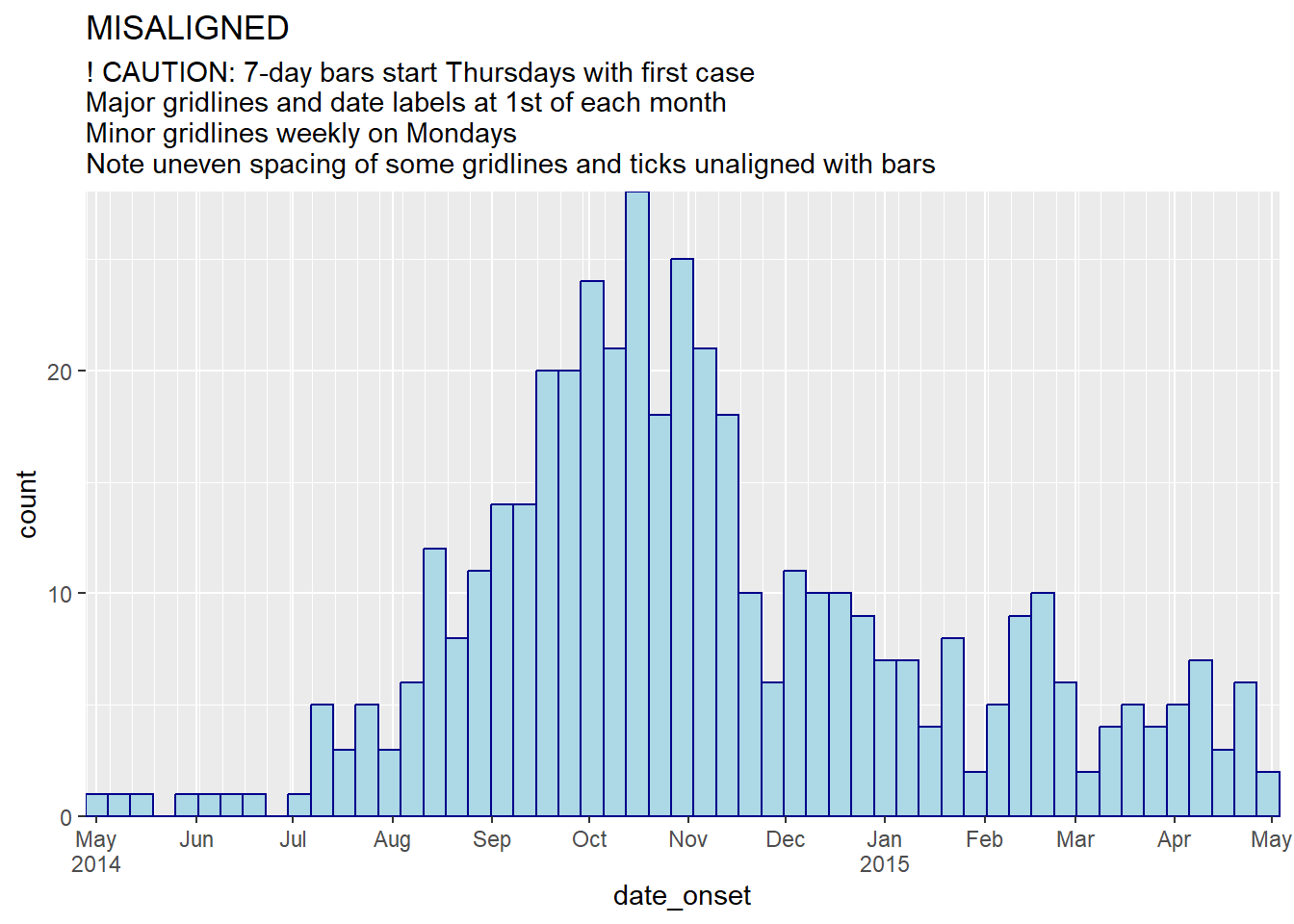
# корзины 7 дней + месяцы
#####################
ggplot(central_data) +
geom_histogram(
mapping = aes(x = date_onset),
binwidth = 7,
color = "darkblue",
fill = "lightblue") +
scale_x_date(
expand = c(0,0), # удалить лишнее пространство по оси x ниже и после столбиков случаев
date_breaks = "months", # 1-ый месяц
date_minor_breaks = "week", # недели с понедельника
label = scales::label_date_short())+ # автоматическое форматирование меток
scale_y_continuous(
expand = c(0,0))+ # удалить лишнее пространство под осью x, сделать все ровным
labs(
title = "MISALIGNED",
subtitle = "! CAUTION: 7-day bars start Thursdays with first case\nMajor gridlines and date labels at 1st of each month\nMinor gridlines weekly on Mondays\nNote uneven spacing of some gridlines and ticks unaligned with bars")
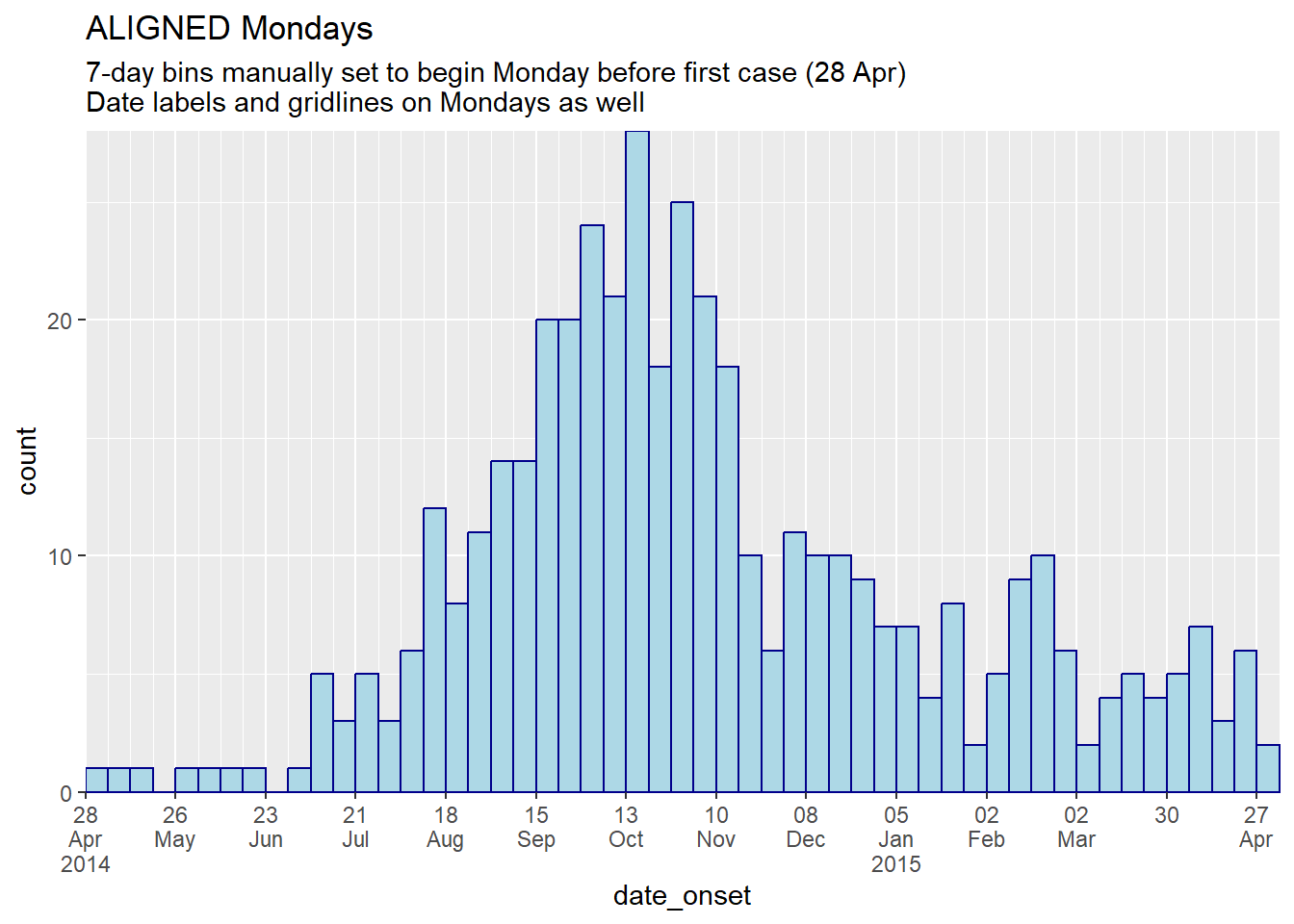
# ПОЛНОЕ ВЫРАВНИВАНИЕ ПО ПОНЕДЕЛЬНИКАМ: укажите вручную, что разрывы между корзинами должны приходиться на понедельник
#################################################################
ggplot(central_data) +
geom_histogram(
mapping = aes(x = date_onset),
# Разрыв гистограммы установлен на 7 дней, начиная с понедельника перед первым случаем
breaks = weekly_breaks_central, # определены ранее на этой странице
closed = "left", # подсчет случаев от начала точки прерывания
color = "darkblue",
fill = "lightblue") +
scale_x_date(
expand = c(0,0), # удалить лишнее пространство по оси x ниже и после столбиков случаев
date_breaks = "4 weeks", # понедельник каждые 4 недели
date_minor_breaks = "week", # недели с понедельника
label = scales::label_date_short())+ # форматирование меток
scale_y_continuous(
expand = c(0,0))+ # удалить лишнее пространство под осью x, сделать все ровным
labs(
title = "ALIGNED Mondays",
subtitle = "7-day bins manually set to begin Monday before first case (28 Apr)\nDate labels and gridlines on Mondays as well")
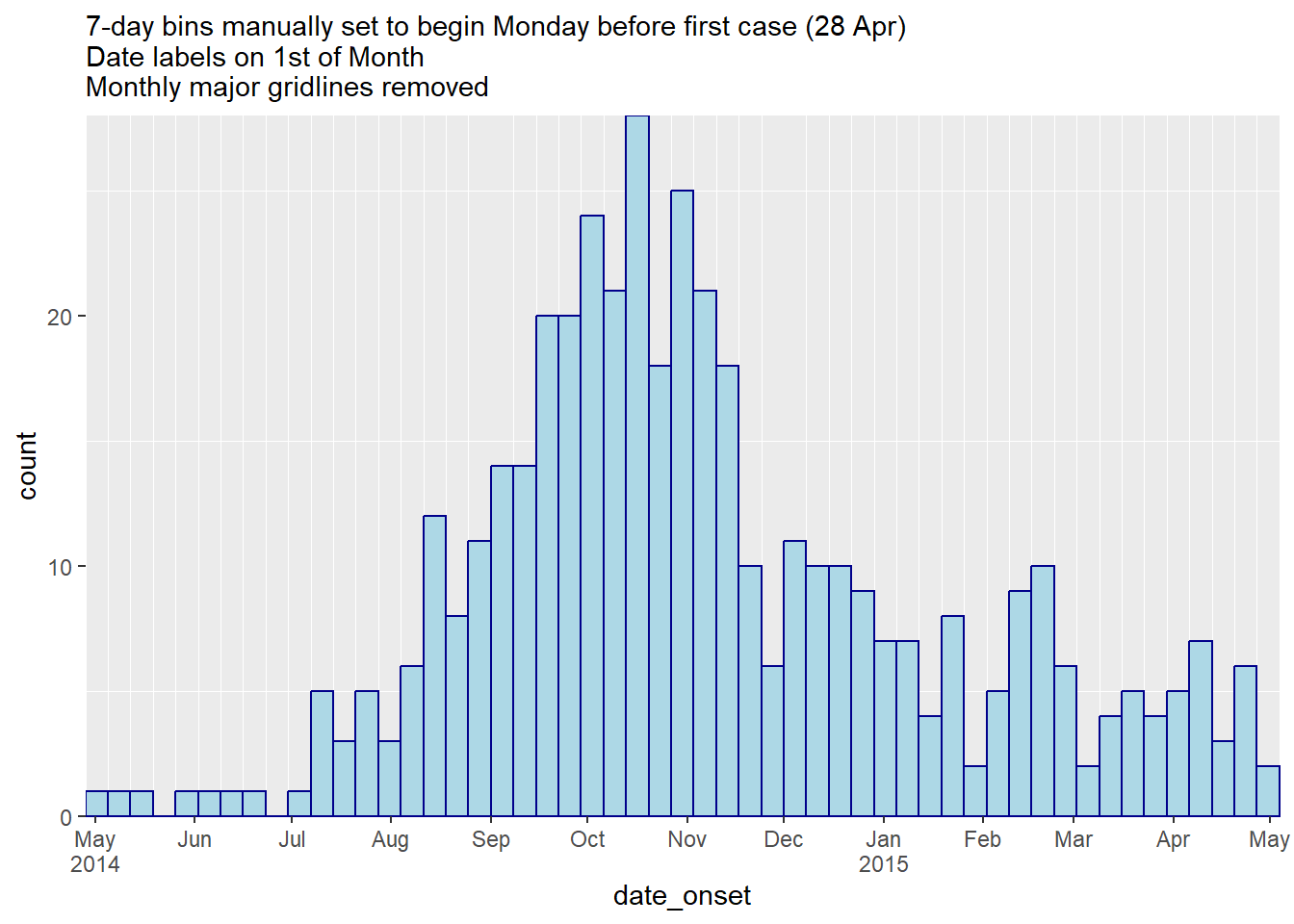
# ПОЛНОЕ ВЫРАВНИВАНИЕ ПО ПОНЕДЕЛЬНИКАМ С МЕТКАМИ МЕСЯЦЕВ:
############################################
ggplot(central_data) +
geom_histogram(
mapping = aes(x = date_onset),
# Разбиение гистограммы на 7 дней, начиная с понедельника перед первым случаем
breaks = weekly_breaks_central, # определены ранее на этой странице
closed = "left", # подсчет случаев от начала точки прерывания
color = "darkblue",
fill = "lightblue") +
scale_x_date(
expand = c(0,0), # удалить лишнее пространство по оси x ниже и после столбиков случаев
date_breaks = "months", # понедельник каждые 4 недели
date_minor_breaks = "week", # недели с понедельника
label = scales::label_date_short())+ # форматирование меток
scale_y_continuous(
expand = c(0,0))+ # удалить лишнее пространство под осью x, сделать все ровным
theme(panel.grid.major = element_blank())+ # Удаление основных линий сетки (приходится на 1-е число месяца)
labs(
itle = "ALIGNED Mondays with MONTHLY labels",
subtitle = "7-day bins manually set to begin Monday before first case (28 Apr)\nDate labels on 1st of Month\nMonthly major gridlines removed")
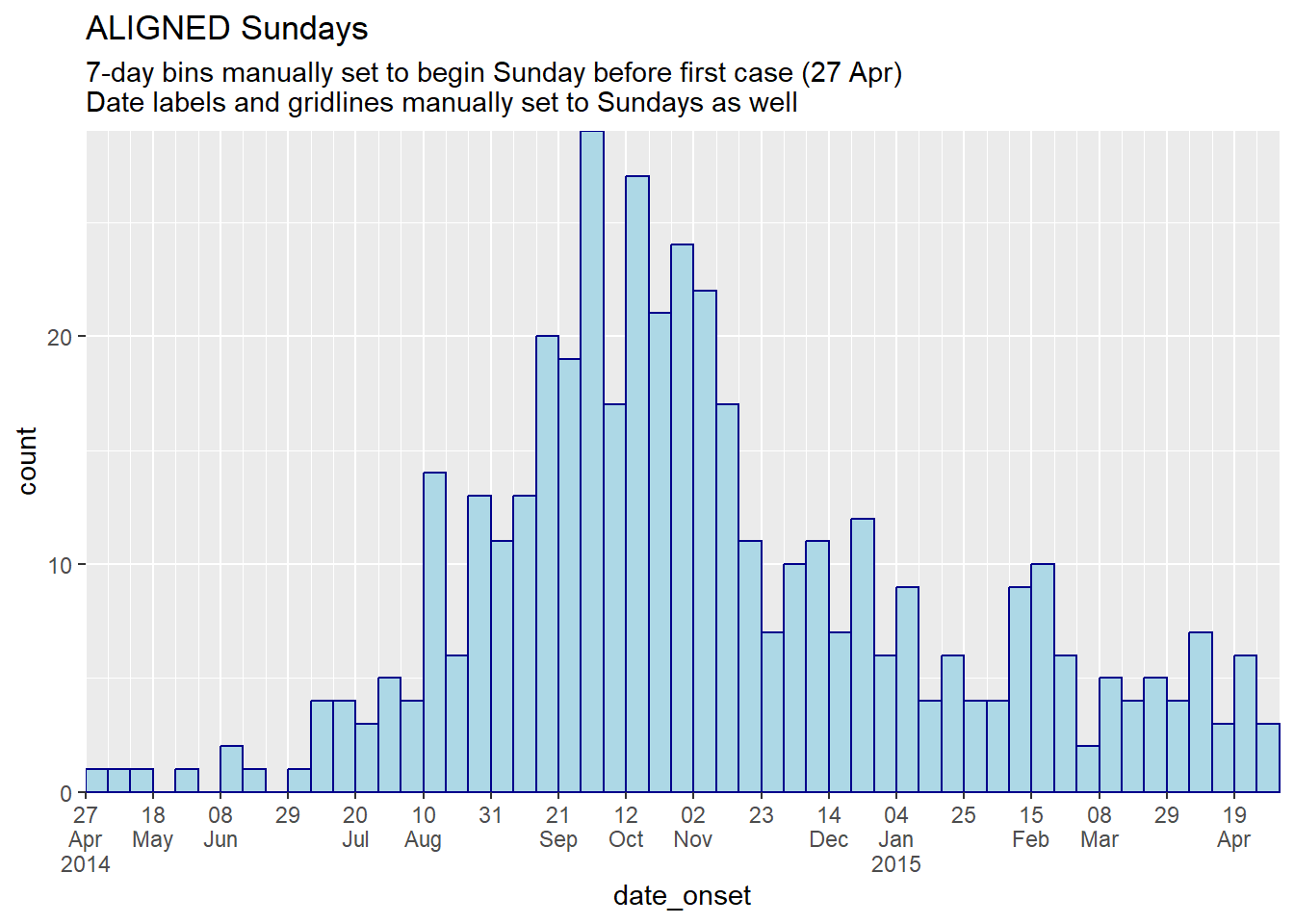
# ПОЛНОЕ ВЫРАВНИВАНИЕ ПО ВОСКРЕСЕНЬЯМ: укажите вручную разрывы корзин и метки, которые должны быть по воскресеньям
############################################################################
ggplot(central_data) +
geom_histogram(
mapping = aes(x = date_onset),
# Разрывы гистограммы устанавливаются на 7 дней, начиная с воскресенья перед первым случаем
breaks = seq.Date(from = floor_date(min(central_data$date_onset, na.rm=T), "week", week_start = 7),
to = ceiling_date(max(central_data$date_onset, na.rm=T), "week", week_start = 7),
by = "7 days"),
closed = "left", # подсчет случаев от начала точки прерывания
color = "darkblue",
fill = "lightblue") +
scale_x_date(
expand = c(0,0),
# разрывы меток даты и основные линии сетки устанавливаются на каждые 3 недели, начиная с воскресенья перед первым случаем
breaks = seq.Date(from = floor_date(min(central_data$date_onset, na.rm=T), "week", week_start = 7),
to = ceiling_date(max(central_data$date_onset, na.rm=T), "week", week_start = 7),
by = "3 weeks"),
# Малые линии сетки устанавливаются на еженедельное начало с воскресенья перед первым случаем
minor_breaks = seq.Date(from = floor_date(min(central_data$date_onset, na.rm=T), "week", week_start = 7),
to = ceiling_date(max(central_data$date_onset, na.rm=T), "week", week_start = 7),
by = "7 days"),
label = scales::label_date_short())+ # форматирование меток
scale_y_continuous(
expand = c(0,0))+ # удалить лишнее пространство под осью x, сделать все ровным
labs(title = "ALIGNED Sundays",
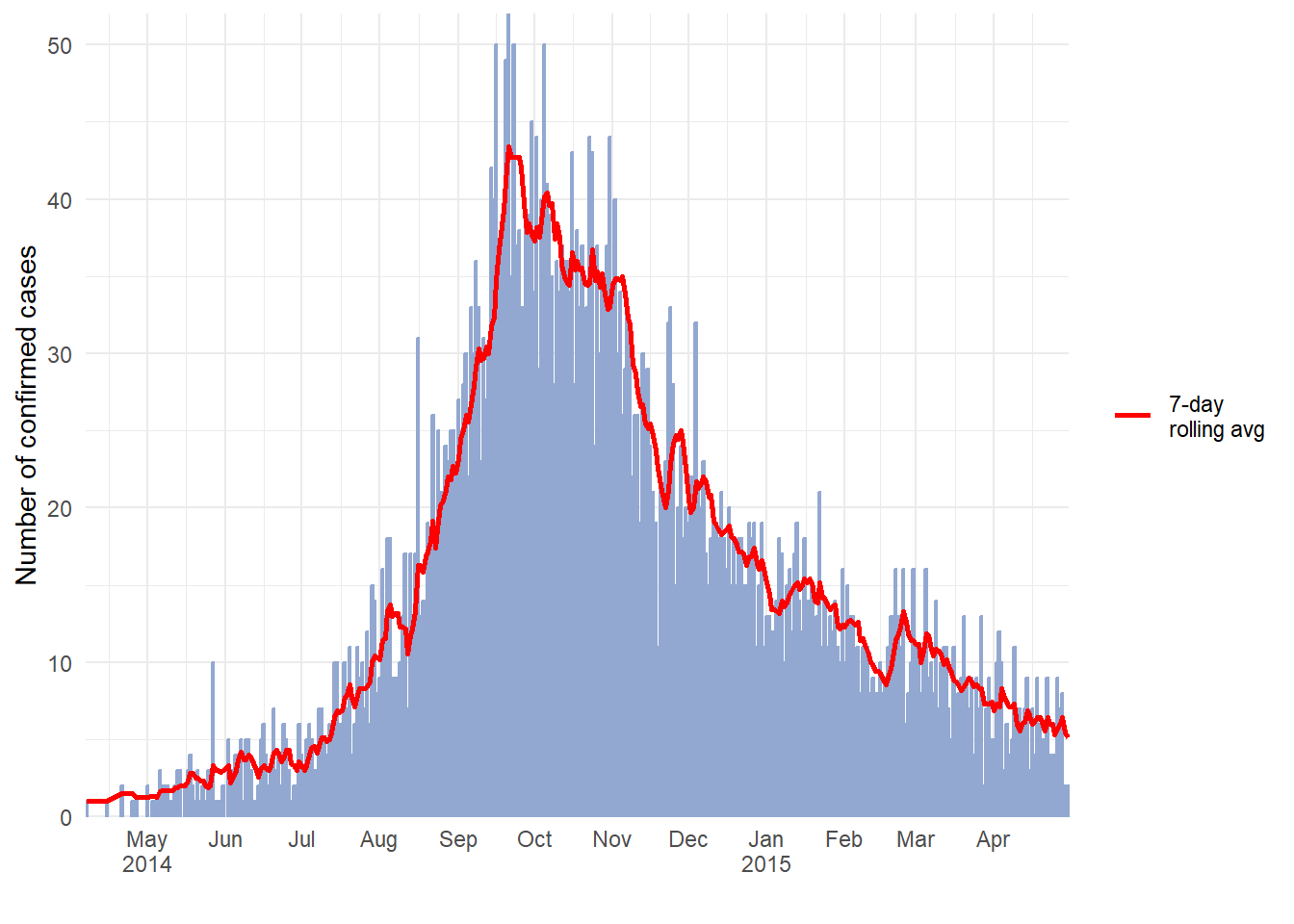
subtitle = "7-day bins manually set to begin Sunday before first case (27 Apr)\nDate labels and gridlines manually set to Sundays as well")Агрегированные данные
Зачастую вместо построчного списка используются агрегированные подсчеты по учреждениям, районам и т.д. эпидкривую можно построить с помощью ggplot(), но код будет несколько иным. В этом разделе будет использован набор данных count_data, который был импортирован ранее, в разделе подготовки данных. Этот набор данных представляет собой linelist, агрегированный до подсчетов день-больница. Ниже показаны первые 50 строк.
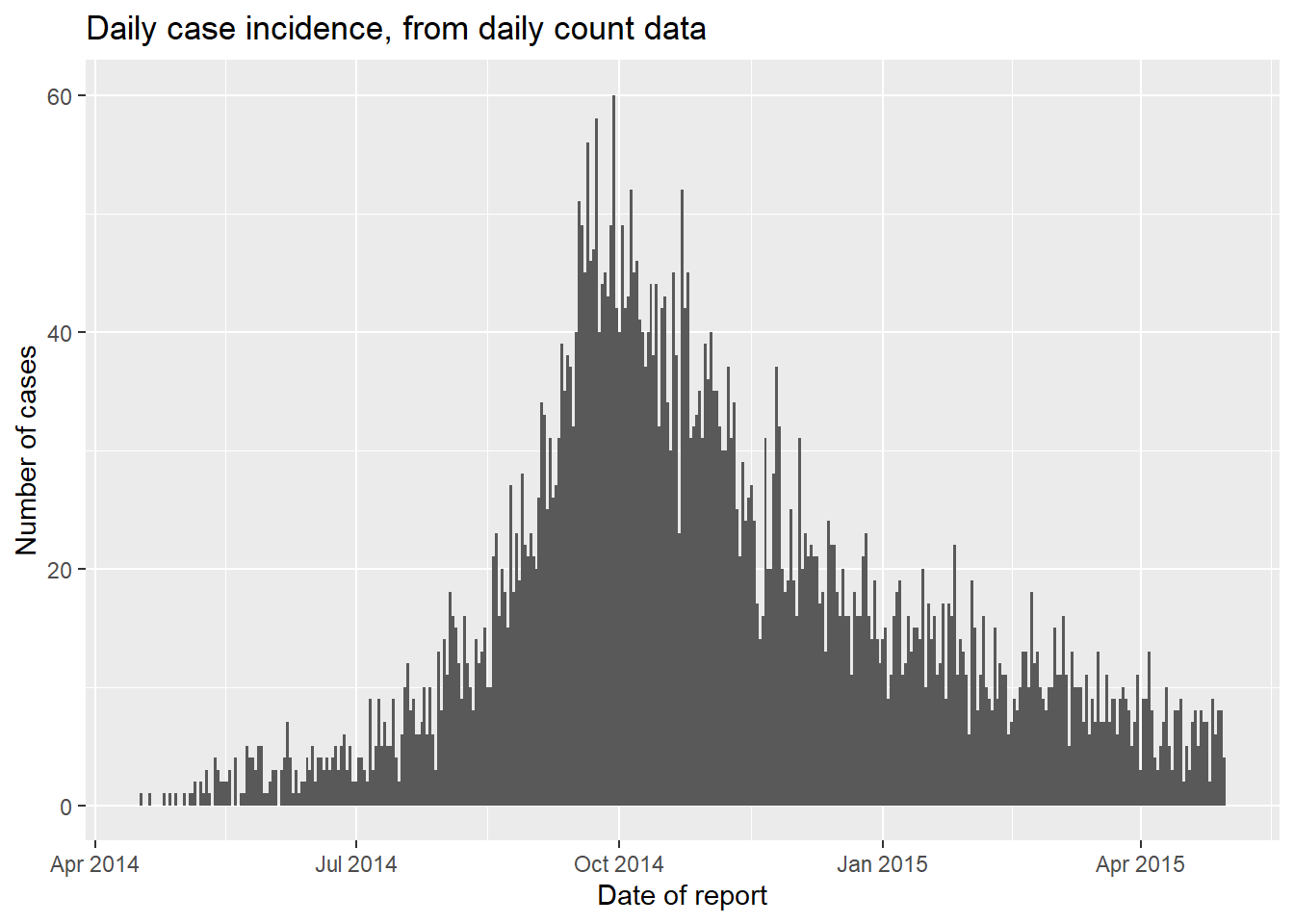
Построение графика ежедневных подсчетов
По этим дневным подсчетам мы можем построить дневную эпидкривую. Вот отличия в коде:
- В эстетическом отображении
aes()укажитеy =в качестве столбца подсчетов (в данном случае название столбца -n_cases) - Добавить аргумент
stat = "identity"вgeom_histogram(), который определяет, что высота столбца должна быть равна значениюy =, а не количеству строк, как по умолчанию. - Добавьте аргумент
width =, чтобы избежать вертикальных белых линий между столбиками. Для ежедневных данных установите значение 1. Для недельных данных - 7. Для месячных данных белые линии являются проблемой (в каждом месяце разное количество дней) - рассмотрите возможность преобразования оси x в категориальный упорядоченный фактор (месяцы) и использованияgeom_col().
ggplot(data = count_data)+
geom_histogram(
mapping = aes(x = date_hospitalisation, y = n_cases),
stat = "identity",
width = 1)+ # Для ежедневных подсчетов задайте ширину = 1, чтобы избежать пробелов между столбиками
labs(
x = "Date of report",
y = "Number of cases",
title = "Daily case incidence, from daily count data")Построение графика недельных подсчетов
Если ваши данные уже представляют собой подсчеты случаев по неделям, то они могут выглядеть как этот набор данных (под названием count_data_weekly):
Ниже показаны первые 50 строк count_data_weekly. Видно, что подсчеты были агрегированы по неделям. Каждая неделя отображается по первому дню недели (по умолчанию это понедельник).
Теперь постройте график так, чтобы x = столбец эпинедели. Не забудьте добавить y = столбец подсчетов в эстетическое отображение и добавить stat = "identity", как объяснялось выше.
ggplot(data = count_data_weekly)+
geom_histogram(
mapping = aes(
x = epiweek, # Ось x - эпинеделя (как дата класса)
y = n_cases_weekly, # высота оси y в еженедельных подсчетах случаев
group = hospital, # мы группируем столбики и раскрашиваем их по больницам
fill = hospital),
stat = "identity")+ # Это также необходимо при построении графиков по данным подсчета
# метки для оси x
scale_x_date(
date_breaks = "2 months", # метки каждые 2 месяца
date_minor_breaks = "1 month", # линии сетки каждый месяц
label = scales::label_date_short())+ # форматирование меток
# Выбор цветовой палитры (используется пакет RColorBrewer)
scale_fill_brewer(palette = "Pastel2")+
theme_minimal()+
labs(
x = "Week of onset",
y = "Weekly case incidence",
fill = "Hospital",
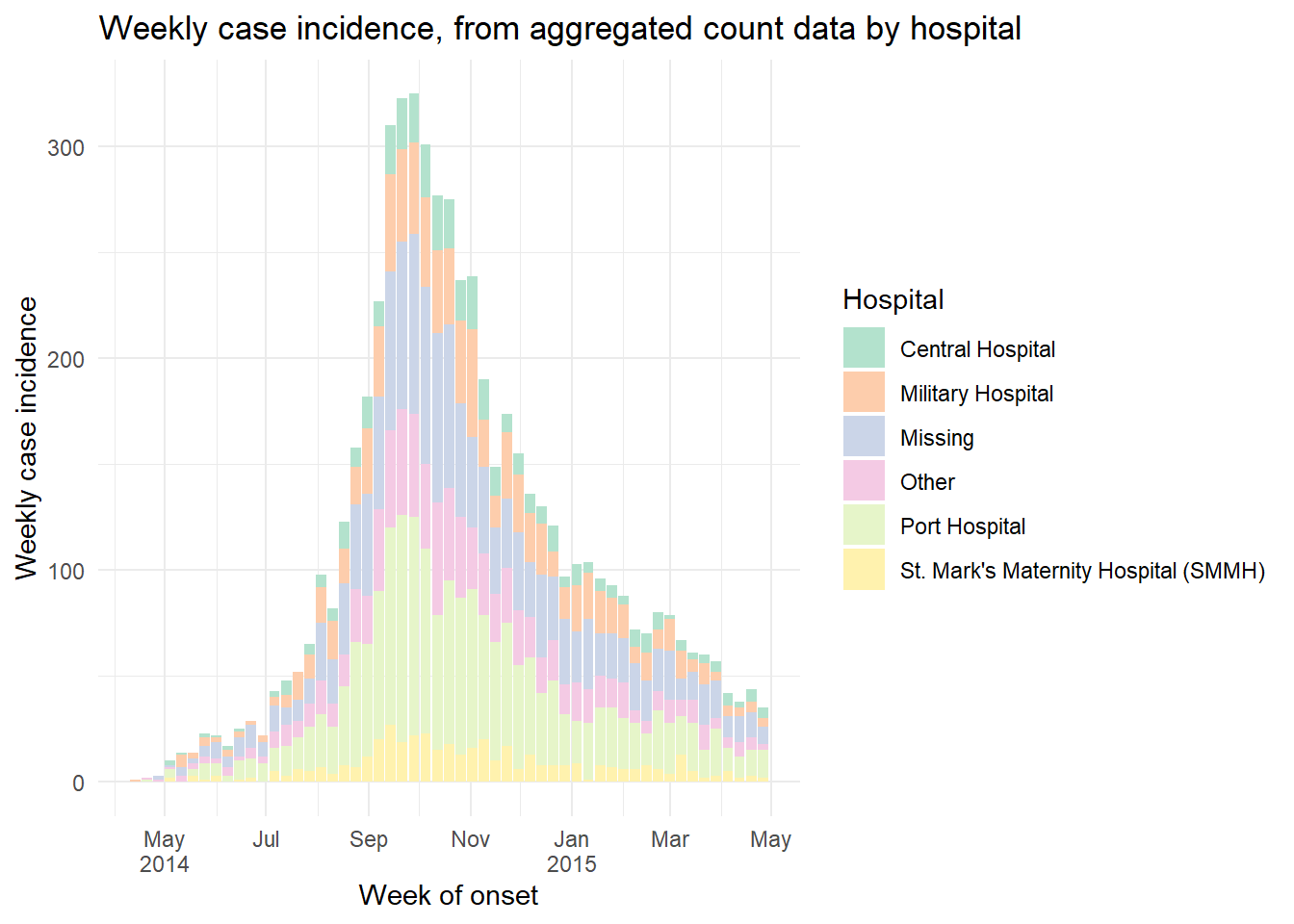
title = "Weekly case incidence, from aggregated count data by hospital")Скользящие средние значения
Подробное описание и несколько вариантов приведены на странице [Скользящие средние]. Ниже приведен один из вариантов расчета скользящих средних с помощью пакета slider. При таком подходе скользящее среднее вычисляется в наборе данных перед построением графика:
- Агрегируем данные в подсчеты по мере необходимости (“дневные”, “недельные” и т.д.) (см. страницу [Группировка данных])
- Создать новый столбец для скользящего среднего, созданный с помощью функции
slide_index()из пакета slider.
- Построить скользящее среднее в виде
geom_line()поверх (после) гистограммы эпидкривой.
Смотрите полезный онлайн ресурс виньетка для пакета slider.
# загрузка пакета
pacman::p_load(slider) # slider, используемый для расчета скользящих средних
# составить набор данных из дневных подсчетов и 7-дневного скользящего среднего
#######################################################
ll_counts_7day <- linelist %>% # начать с построчного списка
## подсчет случаев по дате
count(date_onset, name = "new_cases") %>% # назвать новый столбец с подсчетами как "new_cases"
drop_na(date_onset) %>% # Удаление случаев с отсутствующей датой date_onset
## рассчитать среднее количество случаев в 7-дневном окне
mutate(
avg_7day = slider::slide_index( # создать новый столбец
new_cases, # вычисление на основе значения в столбце new_cases
.i = date_onset, # индекс date_onset col, поэтому в окно включаются даты, не относящиеся к настоящему времени
.f = ~mean(.x, na.rm = TRUE), # функция mean() с удаленными отсутствующими значениями
.before = 6, # окно - день и 6 дней до
.complete = FALSE), # для работы функции unlist() на следующем шаге должно быть FALSE (неверно)
avg_7day = unlist(avg_7day)) # преобразование списка классов в числовые значения классов
# график
######
ggplot(data = ll_counts_7day) + # начать с нового набора данных, определенного выше
geom_histogram( # построение гистограммы эпидкривой
mapping = aes(
x = date_onset, # столбец даты в качестве оси x
y = new_cases), # высота - количество ежедневных новых случаев
stat = "identity", # высота - значение y
fill="#92a8d1", # холодный цвет для столбиков
colour = "#92a8d1", # тот же цвет для границы столбика
)+
geom_line( # строка для скользящего среднего значения
mapping = aes(
x = date_onset, # столбец даты для оси x
y = avg_7day, # значение y задается в столбце скользящего среднего
lty = "7-day \nrolling avg"), # название строки в легенде
color="red", # цвет линии
size = 1) + # ширина линии
scale_x_date( # шкала даты
date_breaks = "1 month",
label = scales::label_date_short(), # форматирование меток
expand = c(0,0)) +
scale_y_continuous( # масштаб оси y
expand = c(0,0),
limits = c(0, NA)) +
labs(
x="",
y ="Number of confirmed cases",
fill = "Legend")+
theme_minimal()+
theme(legend.title = element_blank()) # удаляет заголовок легендыФасетирование/малые множества
Как и в случае с другими ggplots, можно создавать фасетные графики (“малые множества”). Как объясняется на странице [Советы по использованию ggplot] данного руководства, для этого можно использовать либо facet_wrap(), либо facet_grid(). Здесь мы демонстрируем использование facet_wrap(). Для эпидкривых обычно проще использовать facet_wrap(), так как, скорее всего, фасетировать нужно только один столбец.
Общий синтаксис - facet_wrap(rows ~ cols), где слева от тильды (~) - название столбца, который будет распределен по “строкам” фасетного графика, а справа от тильды - название столбца, который будет распределен по “столбцам” фасетного графика. Проще всего использовать одно название столбца справа от тильды: facet_wrap(~age_cat).
Свободные оси
Вам необходимо решить, будут ли шкалы осей для каждого фасета “фиксированы” на одних и тех же размерах (по умолчанию) или “свободны” (то есть будут меняться в зависимости от данных в фасете). Это можно сделать с помощью аргумента scales = в функции facet_wrap(), указав “free_x” или “free_y”, или “free”.
Количество столбцов и строк фасетов
Это может быть задано с помощью ncol = и nrow = внутри facet_wrap()..
Порядок панелей
Чтобы изменить порядок вывода фасетов, измените базовый порядок уровней факторного столбца, используемого для создания фасетов.
Эстетика
Размер и формат шрифта, цвет полосы и т.д. могут быть изменены с помощью функции theme() с аргументами типа:
-
strip.text = element_text()( размер, цвет, шрифт, угол…) -
strip.background = element_rect()(например, element_rect(fill=“grey”))
-
strip.position =(положение полосы “внизу”, “вверху”, “слева” или “справа”)
Метки полоски
Метки фасетных графиков могут быть изменены через “метки” столбца как фактора или с помощью ” маркера меток”.
Создайте такой маркер меток, используя функцию as_labeller() из ggplot2. Затем укажите эту метку в аргументе labeller = функции facet_wrap(), как показано ниже.
my_labels <- as_labeller(c(
"0-4" = "Ages 0-4",
"5-9" = "Ages 5-9",
"10-14" = "Ages 10-14",
"15-19" = "Ages 15-19",
"20-29" = "Ages 20-29",
"30-49" = "Ages 30-49",
"50-69" = "Ages 50-69",
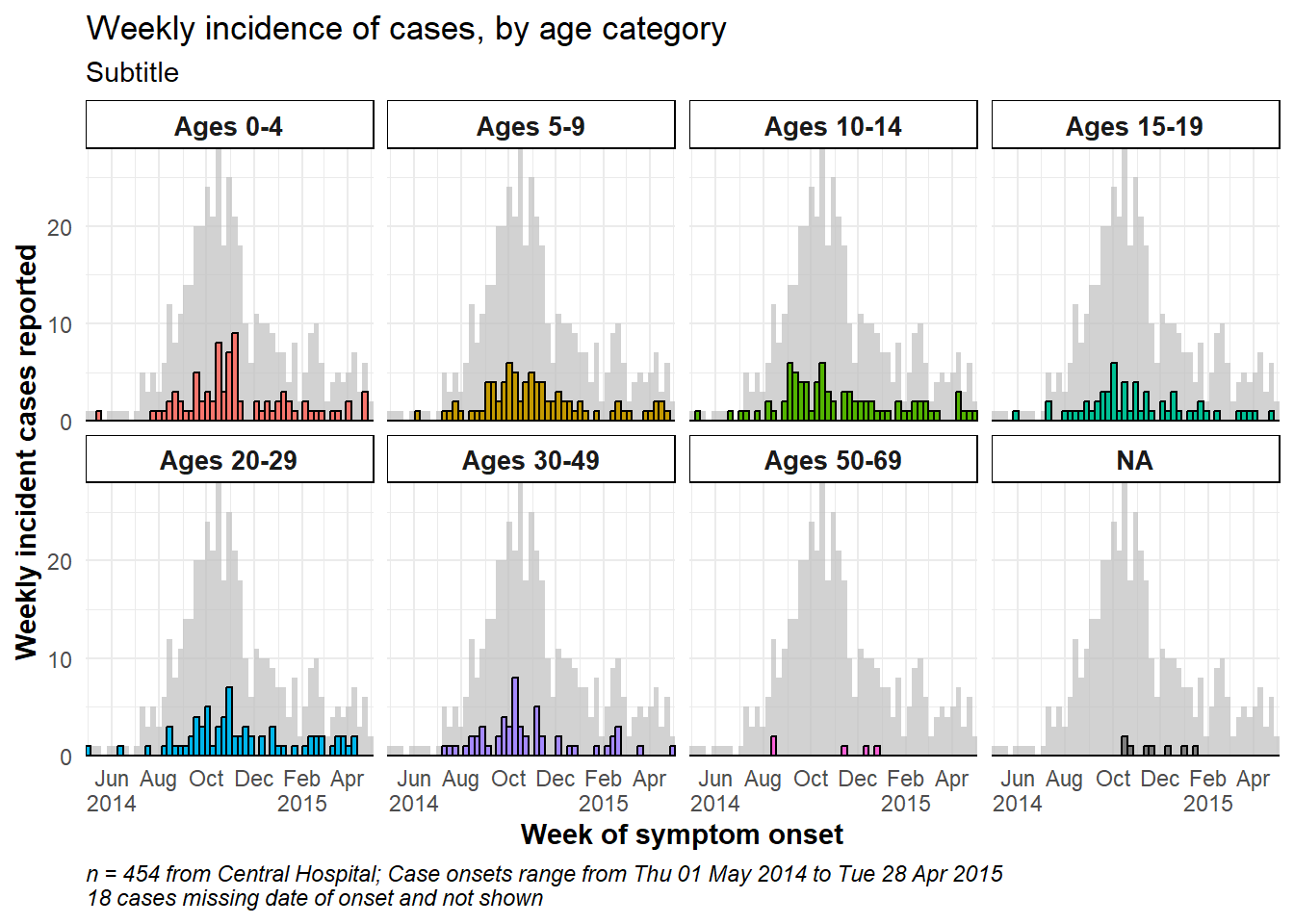
"70+" = "Over age 70"))Пример фасетного графика - фасет по столбцу age_cat.
# построить график
###########
ggplot(central_data) +
geom_histogram(
mapping = aes(
x = date_onset,
group = age_cat,
fill = age_cat), # аргументы внутри aes() применяются по группам
color = "black", # Аргументы вне aes() применяются ко всем данным
# разрывы гистограммы
breaks = weekly_breaks_central, # предварительно заданный вектор дат (см. ранее на этой странице)
closed = "слева" # подсчет случаев от начала точки прерывания
)+
# метки на оси х
scale_x_date(
expand = c(0,0), # удалить лишнее пространство по оси x под и после столбиков случаев
date_breaks = "2 months", # метки появляются каждые 2 месяца
date_minor_breaks = "1 month", # вертикальные линии появляются каждый 1 месяц
label = scales::label_date_short())+ # форматирование меток
# ось y
scale_y_continuous(expand = c(0,0))+ # удаляет лишнее пространство по оси y между нижней частью столбиков и метками
# темы эстетики
theme_minimal()+ # набор тем для упрощения графика
theme(
plot.caption = element_text(face = "italic", hjust = 0), # надпись слева курсивом
axis.title = element_text(face = "bold"),
legend.position = "bottom",
strip.text = element_text(face = "bold", size = 10),
strip.background = element_rect(fill = "grey"))+ # заголовки осей выделены жирным шрифтом
# создать фасеты
facet_wrap(
~age_cat,
ncol = 4,
strip.position = "top",
labeller = my_labels)+
# метки
labs(
title = "Weekly incidence of cases, by age category",
subtitle = "Subtitle",
fill = "Age category", # задать новый заголовок для легенды
x = "Week of symptom onset",
y = "Weekly incident cases reported",
caption = stringr::str_glue("n = {nrow(central_data)} from Central Hospital; Case onsets range from {format(min(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')} to {format(max(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')}\n{nrow(central_data %>% filter(is.na(date_onset)))} cases missing date of onset and not shown"))Более подробную информацию о маркере меток здесь ссылка.
Вся эпидемия в фасетном фоне
Чтобы показать общую эпидемию на фоне каждого фасета, добавьте к ggplot функцию gghighlight() с пустыми скобками. Это сделано с помощью пакета gghighlight. Обратите внимание на то, что теперь максимум по оси y во всех фасетах основывается на пике всей эпидемии. Другие примеры использования этого пакета приведены на странице [Советы по использованию ggplot].
ggplot(central_data) +
# эпидкривые по группам
geom_histogram(
mapping = aes(
x = date_onset,
group = age_cat,
fill = age_cat), # аргументы внутри aes() применяются по группам
color = "black", # Аргументы вне aes() применяются ко всем данным
# разрывы гистограммы
breaks = weekly_breaks_central, # предварительно заданный вектор дат (см. ранее на этой странице)
closed = "left", # подсчет случаев от начала точки прерывания
)+ # предварительно заданный вектор дат (см. начало раздела ggplot)
# добавить серую эпидемию к фону каждого фасета
gghighlight::gghighlight()+
# метки на оси х
scale_x_date(
expand = c(0,0), # удалить лишнее пространство по оси x ниже и после столбиков случаев
date_breaks = "2 months", # метки появляются каждые 2 месяца
date_minor_breaks = "1 month", # вертикальные линии появляются каждый 1 месяц
label = scales::label_date_short())+ # форматирование меток
# ось y
scale_y_continuous(expand = c(0,0))+ # удаляет лишнее пространство по оси y ниже 0
# темы эстетики
theme_minimal()+ # набор тем для упрощения графика
theme(
plot.caption = element_text(face = "italic", hjust = 0), # надпись слева курсивом
axis.title = element_text(face = "bold"),
legend.position = "bottom",
strip.text = element_text(face = "bold", size = 10),
strip.background = element_rect(fill = "white"))+ # заголовки осей выделены жирным шрифтом
# создать фасеты create facets
facet_wrap(
~age_cat, # каждый график - это одно значение age_cat
ncol = 4, # количество столбцов
strip.position = "top", # положение заголовка/полосы фасета
labeller = my_labels)+ # маркер меток определяется выше
# метки
labs(
title = "Weekly incidence of cases, by age category",
subtitle = "Subtitle",
fill = "Age category", # задать новый заголовок для легенды
x = "Week of symptom onset",
y = "Weekly incident cases reported",
caption = stringr::str_glue("n = {nrow(central_data)} from Central Hospital; Case onsets range from {format(min(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')} to {format(max(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')}\n{nrow(central_data %>% filter(is.na(date_onset)))} cases missing date of onset and not shown"))Один фасет с данными
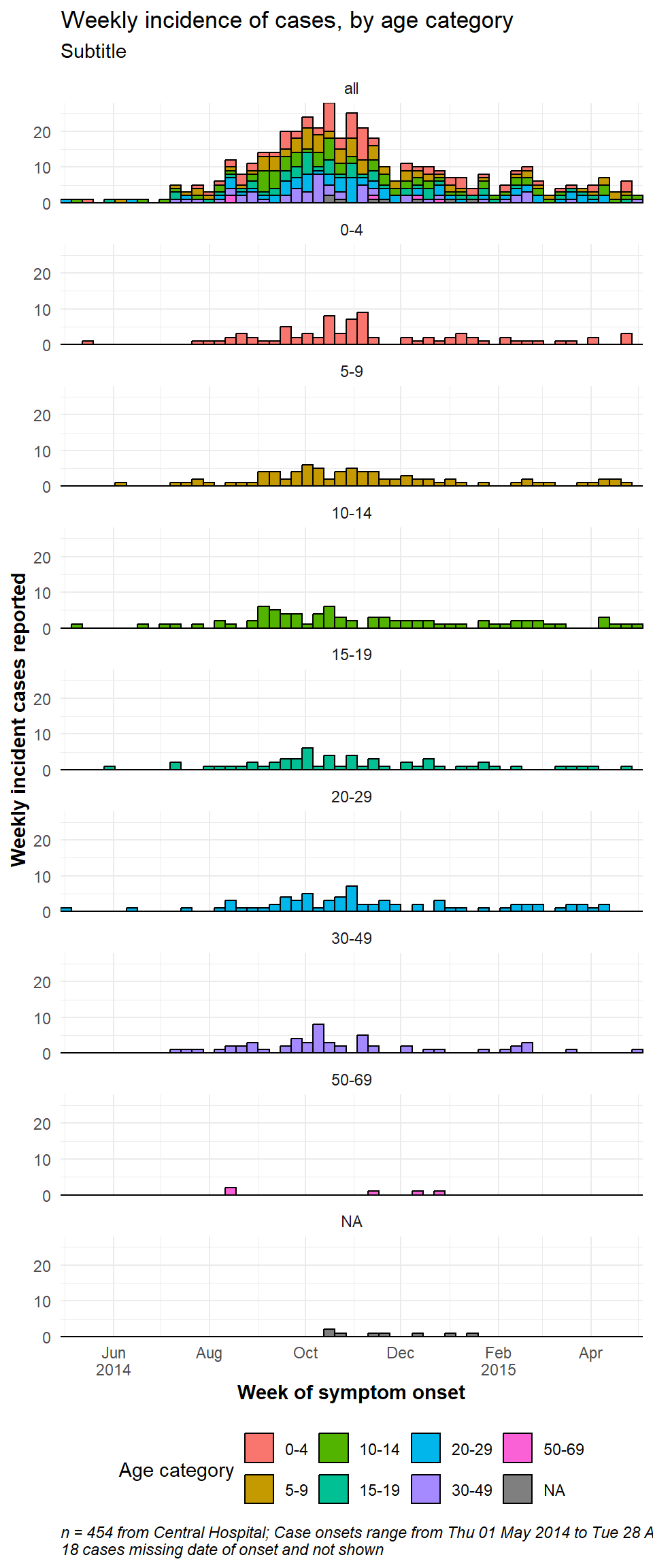
Если вы хотите иметь один фасет, содержащий все данные, продублируйте весь набор данных и рассматривайте дубликаты как одно фасетное значение. В этом может помочь “вспомогательная” функция CreateAllFacet(), представленная ниже (см.этот пост в блоге). При ее выполнении количество строк удваивается, и появляется новый столбец facet, в котором дублированные строки будут иметь значение ” все”, а исходные строки - исходное значение столбца фасета. Теперь остается выполнить фасет на столбце facet.
Вот вспомогательная функция. Запустите ее, чтобы она была вам доступна.
# Определение вспомогательной функции
CreateAllFacet <- function(df, col){
df$facet <- df[[col]]
temp <- df
temp$facet <- "all"
merged <-rbind(temp, df)
# убедиться, что значение фасета является фактором
merged[[col]] <- as.factor(merged[[col]])
return(merged)
}Теперь применим вспомогательную функцию к набору данных, к столбцу age_cat:
# Создать дублированный набор данных с новым столбцом " фасет" для отображения "всех" возрастных категорий в качестве другого уровня фасета
central_data2 <- CreateAllFacet(central_data, col = "age_cat") %>%
# задать уровни факторов
mutate(facet = fct_relevel(facet, "all", "0-4", "5-9",
"10-14", "15-19", "20-29",
"30-49", "50-69", "70+"))Warning: There was 1 warning in `mutate()`.
ℹ In argument: `facet = fct_relevel(...)`.
Caused by warning:
! 1 unknown level in `f`: 70+# проверка уровней
table(central_data2$facet, useNA = "always")
all 0-4 5-9 10-14 15-19 20-29 30-49 50-69 <NA>
454 84 84 82 58 73 57 7 9 Заметными изменениями в команде ggplot() являются:
- Теперь используются данные central_data2 (в два раза больше строк, с новым столбцом ” фасет”)
- Необходимо обновить метку, если она используется.
- Дополнительно: для получения вертикально сложенных фасетов: столбец фасет переносится в строку уравнения и справа заменяется на “.” (
facet_wrap(facet~.)), аncol = 1. Также может потребоваться настройка ширины и высоты сохраняемого изображения графика в формате png (см.ggsave()в [Советы по использованию ggplot]).
ggplot(central_data2) +
# фактические эпидкривые по группам
geom_histogram(
mapping = aes(
x = date_onset,
group = age_cat,
fill = age_cat), # аргументы внутри aes() применяются по группам
color = "black", # Аргументы вне aes() применяются ко всем данным
# разрывы гистограммы
breaks = weekly_breaks_central, # предварительно заданный вектор дат (см. ранее на этой странице)
closed = "left", # подсчет случаев от начала точки прерывания
)+ # предварительно заданный вектор дат (см. начало раздела ggplot)
# Метки на оси x
scale_x_date(
expand = c(0,0), # удалить лишнее пространство по оси x под и после гистограмм
date_breaks = "2 months", # метки появляются каждые 2 месяца
date_minor_breaks = "1 month", # вертикальные линии появляются каждые 1 месяц
label = scales::label_date_short())+ # автоматическое форматирование меток
# ось y
scale_y_continuous(expand = c(0,0))+ # удаляет лишнее пространство по оси y между нижней частью столбиков и метками
# эстетические темы
theme_minimal()+ # набор тем для упрощения графика
theme(
plot.caption = element_text(face = "italic", hjust = 0), # надпись слева курсивом
axis.title = element_text(face = "bold"),
legend.position = "bottom")+
# создать фасеты
facet_wrap(facet~. , # каждый график представляет собой одно значение фасета
ncol = 1)+
# метки
labs(title = "Weekly incidence of cases, by age category",
subtitle = "Subtitle",
fill = "Age category", # задать новый заголовок для легенды
x = "Week of symptom onset",
y = "Weekly incident cases reported",
caption = stringr::str_glue("n = {nrow(central_data)} from Central Hospital; Case onsets range from {format(min(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')} to {format(max(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')}\n{nrow(central_data %>% filter(is.na(date_onset)))} cases missing date of onset and not shown"))32.3 Предварительные данные
Самые недавние данные, представленные на эпидкривых, часто должны быть помечены как предварительные или с учетом задержек в представлении информации. Это можно сделать, добавив вертикальную линию и/или прямоугольник за определенное количество дней. Ниже приведены два варианта:
- Используйте функцию
annotate():- Для линии используйте
annotate(geom = "segment"). Укажитеx,xend,yиyend. Настройте размер, тип линии (lty) и цвет.
- Для прямоугольника используйте
annotate(geom = "rect"). Укажите xmin/xmax/ymin/ymax. Настройте цвет и альфу.
- Для линии используйте
- Сгруппируйте данные по предварительному статусу и раскрасьте эти столбики по-разному
ВНИМАНИЕ: Для построения прямоугольника можно попробовать geom_rect(), но регулировка прозрачности не подходит в контексте построчного списка. Данная функция накладывает один прямоугольник на каждое наблюдение/строку! Используйте либо очень низкую альфу (например, 0.01), либо другой подход.
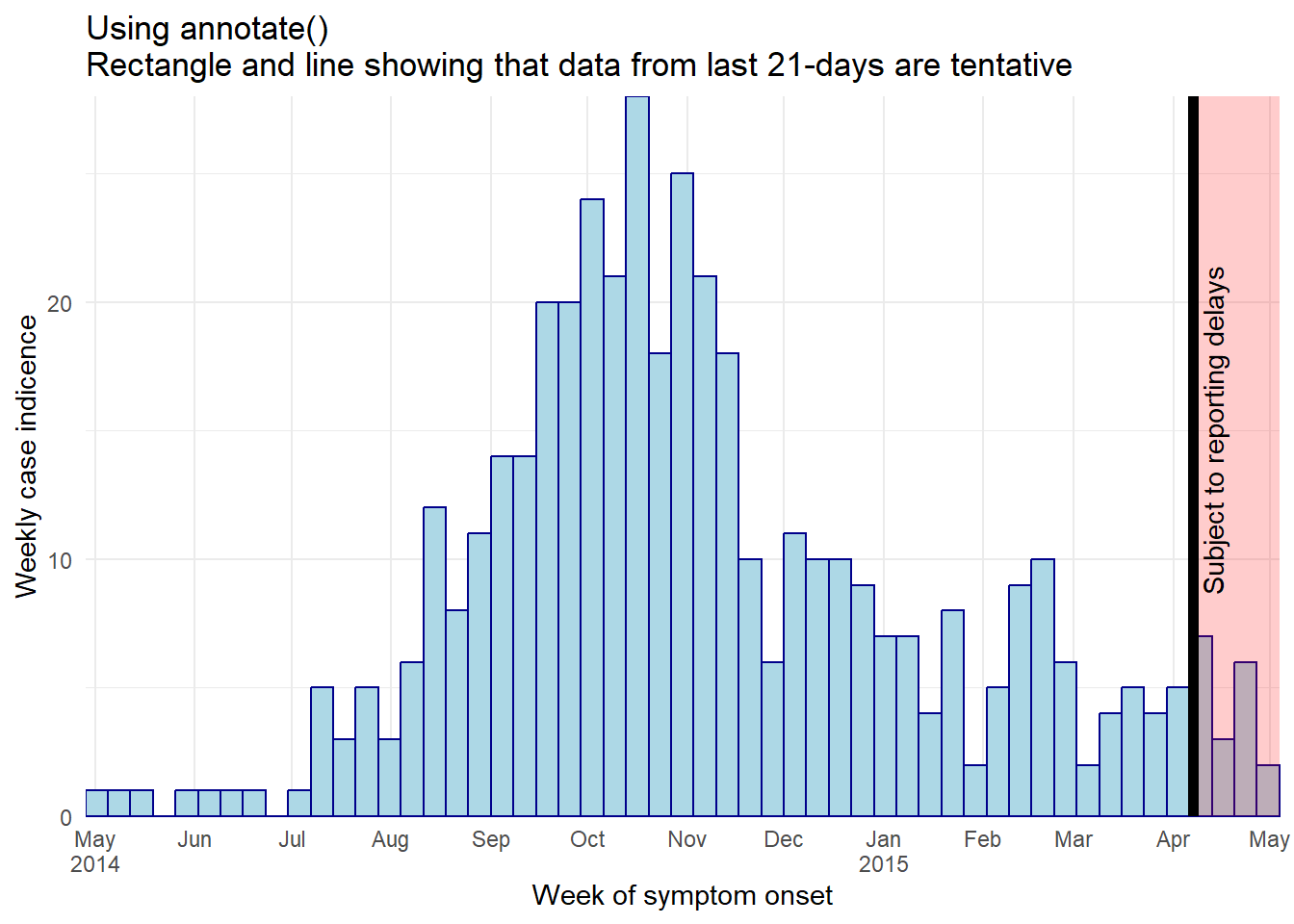
Использование annotate()
- Внутри
annotate(geom = "rect")аргументыxminиxmaxдолжны иметь значения класса Дата.
- Обратите внимание на то, что поскольку данные агрегируются в недельные столбики, а последний столбик простирается до понедельника после последней точки данных, то может показаться, что заштрихованная область охватывает 4 недели
- Вот
annotate()онлайн пример
ggplot(central_data) +
# гистограмма
geom_histogram(
mapping = aes(x = date_onset),
breaks = weekly_breaks_central, # предопределенный вектор дат - см. верхнюю часть раздела ggplot
closed = "left", # подсчет случаев от начала точки прерывания
color = "darkblue",
fill = "lightblue") +
# шкалы
scale_y_continuous(expand = c(0,0))+
scale_x_date(
expand = c(0,0), # удалить лишнее пространство по оси x под и после столбиков случая
date_breaks = "1 month", # 1-е число месяца
date_minor_breaks = "1 month", # 1-е число месяца
label = scales::label_date_short())+ # автоматическое форматирование меток
# метки и тема
labs(
title = "Using annotate()\nRectangle and line showing that data from last 21-days are tentative",
x = "Week of symptom onset",
y = "Weekly case indicence")+
theme_minimal()+
# добавить полупрозрачный красный прямоугольник к предварительным данным
annotate(
"rect",
xmin = as.Date(max(central_data$date_onset, na.rm = T) - 21), # Примечание должно быть обернуто в as.Date()
xmax = as.Date(Inf), # Примечание должно быть обернуто в as.Date()
ymin = 0,
ymax = Inf,
alpha = 0.2, # альфа легко и интуитивно настраивается с помощью функции annotate()
fill = "red")+
# добавьте черную вертикальную линию поверх других слоев
annotate(
"segment",
x = max(central_data$date_onset, na.rm = T) - 21, # 21 день до получения последних данных
xend = max(central_data$date_onset, na.rm = T) - 21,
y = 0, # линия начинается в точке y = 0
yend = Inf, # линия к вершине графика
size = 2, # размер линии
color = "black",
lty = "solid")+ # Тип линии, например, "сплошная", "пунктирная"
# добавить текст в прямоугольник
annotate(
"text",
x = max(central_data$date_onset, na.rm = T) - 15,
y = 15,
label = "Subject to reporting delays",
angle = 90)Такую же черную вертикальную линию можно получить с помощью приведенного ниже кода, но при использовании geom_vline() теряется возможность управления высотой:
geom_vline(xintercept = max(central_data$date_onset, na.rm = T) - 21,
size = 2,
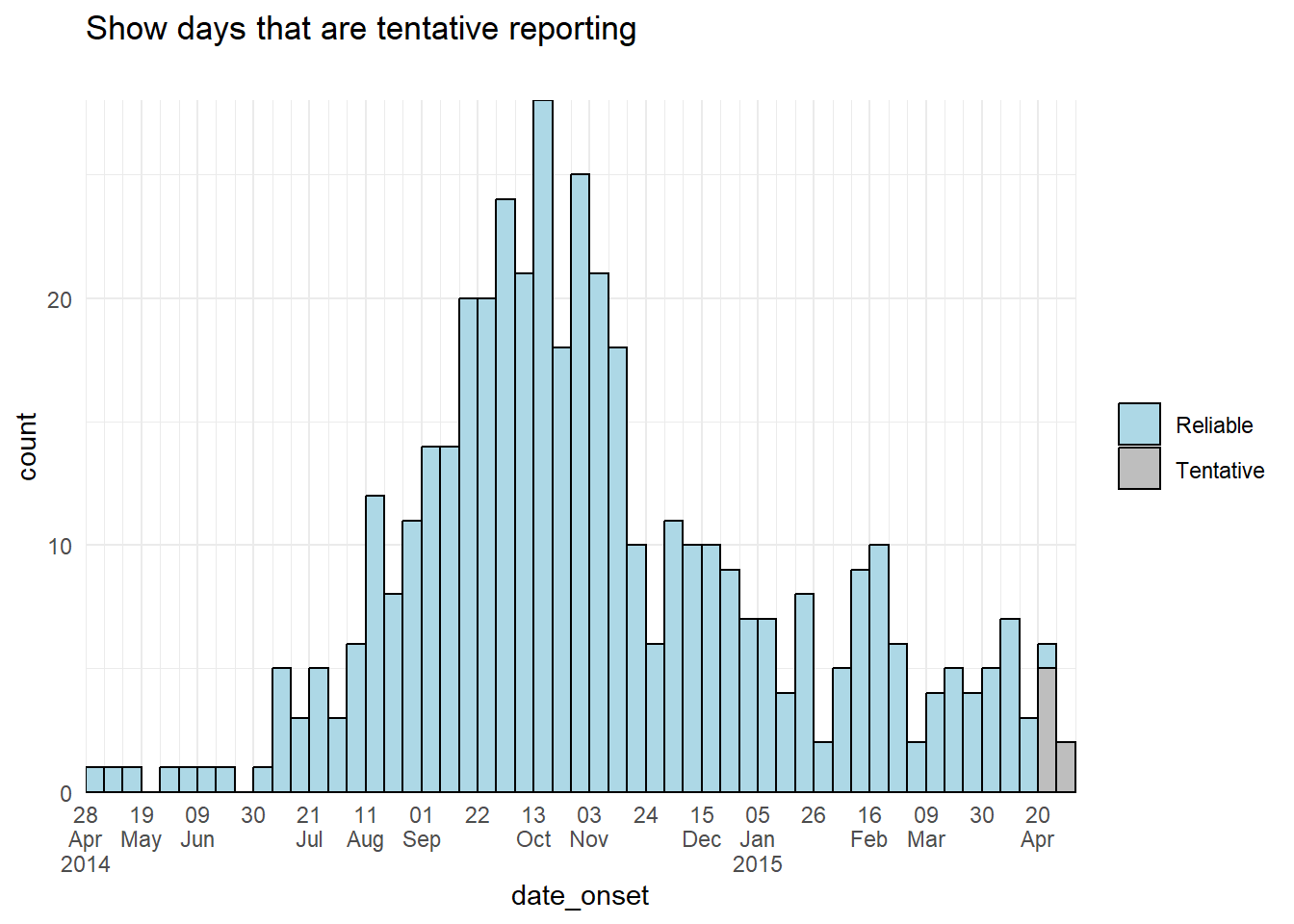
color = "black")Цвет столбиков
Альтернативным подходом может быть настройка цвета или отображения самих столбиков предварительных данных. Можно создать новый столбец на этапе подготовки данных и использовать его для группировки данных таким образом, чтобы aes(fill = ) предварительных данных мог быть другого цвета или альфа, чем другие столбцы.
# добавить столбец
############
plot_data <- central_data %>%
mutate(tentative = case_when(
date_onset >= max(date_onset, na.rm=T) - 7 ~ "Tentative", # предварительно, если за последние 7 дней
TRUE ~ "Reliable")) # все остальные данные надежные
# график
######
ggplot(plot_data, aes(x = date_onset, fill = tentative)) +
# гистограмма
geom_histogram(
breaks = weekly_breaks_central, # предопределенный вектор данных, см. верхнюю часть страницы ggplot
closed = "left", # подсчет случаев от начала точки прерывания
color = "black") +
# гкалы
scale_y_continuous(expand = c(0,0))+
scale_fill_manual(values = c("lightblue", "grey"))+
scale_x_date(
expand = c(0,0), # удалить лишнее пространство по оси x под и после столбиков случая
date_breaks = "3 weeks", # Понедельник каждые 3 недели
date_minor_breaks = "week", # Недели с понедельника
label = scales::label_date_short())+ # автоматическое форматирование меток
# метки и тема
labs(title = "Show days that are tentative reporting",
subtitle = "")+
theme_minimal()+
theme(legend.title = element_blank()) # удалить заголовок легенды32.4 Многоуровневые метки даты
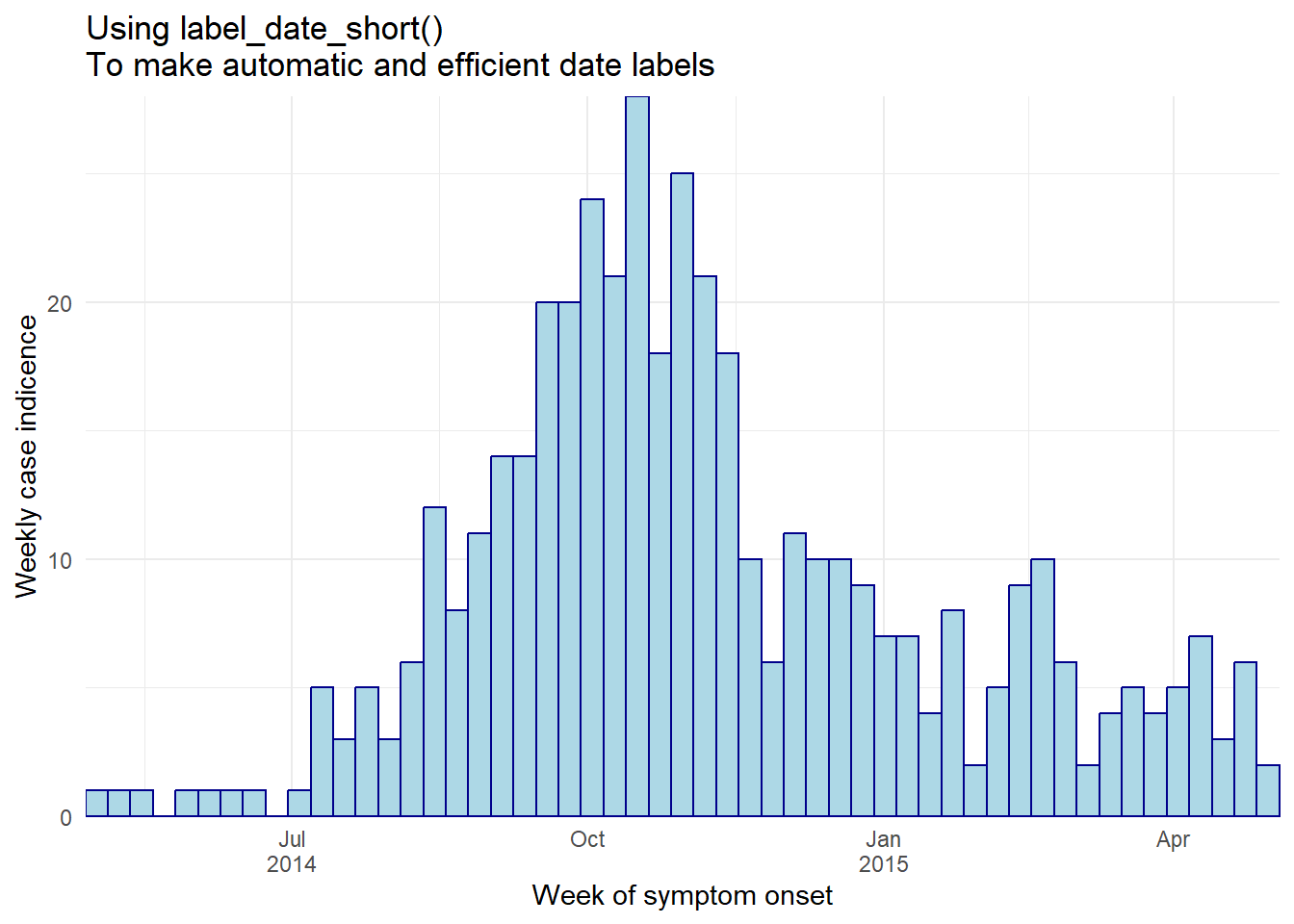
Если вам нужны многоуровневые метки даты (например, месяц и год) без дублирования нижних уровней меток, рассмотрите один из описанных ниже подходов:
Помните - вы можете использовать такие инструменты, как \n внутри аргументов date_labels или labels, чтобы поместить части каждой метки на новую строку ниже. Однако приведенные ниже коды помогут вам вынести годы или месяцы (например) на нижнюю строку и только один раз.
Самый простой способ - присвоить аргумент labels = в scale_x_date() функции label_date_short() из пакета scales (внимание: не забудьте включить пустые круглые скобки (), как показано ниже). Эта функция автоматически строит эффективные метки даты (подробнее здесь). Дополнительным преимуществом этой функции является то, что метки будут автоматически подстраиваться по мере расширения данных во времени: от дней, недель, месяцев и лет.
ggplot(central_data) +
# гистограмма
geom_histogram(
mapping = aes(x = date_onset),
breaks = weekly_breaks_central, # предопределенный вектор дат - см. верхнюю часть раздела ggplot
closed = "left", # подсчет случаев от начала точки прерывания
color = "darkblue",
fill = "lightblue") +
# Масштаб оси y, как и раньше
scale_y_continuous(expand = c(0,0))+
# Масштаб оси x задает удобные метки даты
scale_x_date(
expand = c(0,0), # удалить лишнее пространство по оси x под и после стобиков случая
labels = scales::label_date_short())+ # автоматические удобные метки с датой
# метки и темы
labs(
title = "Using label_date_short()\nTo make automatic and efficient date labels",
x = "Week of symptom onset",
y = "Weekly case indicence")+
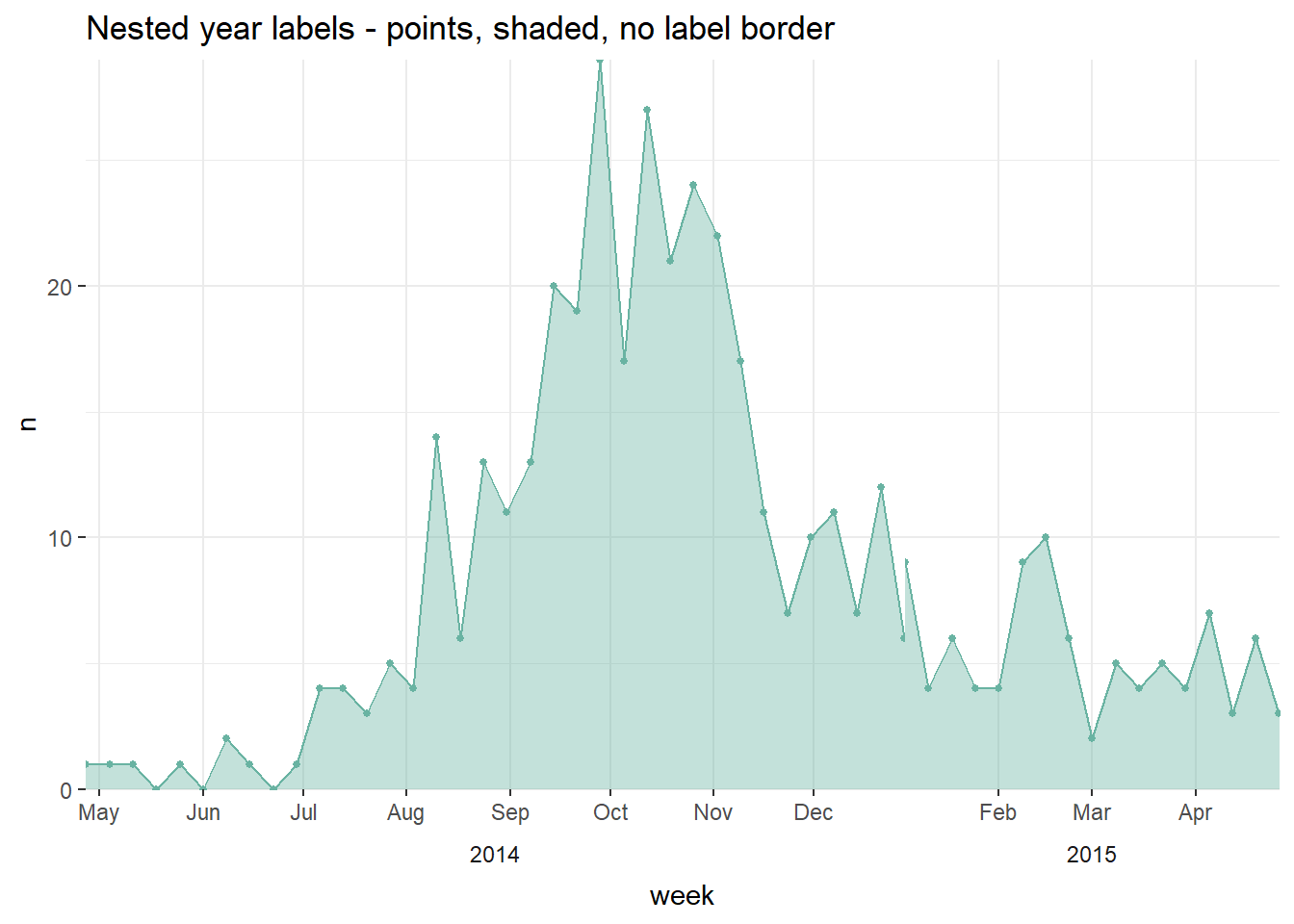
theme_minimal()Второй вариант - использование фасетирования. Ниже:
- Количество случаев агрегировано по неделям из эстетических соображений. Подробности см. на странице эпидкривые (вкладка Агрегированные данные).
- Вместо гистограммы используется линия
geom_area(), так как подход фасетирования, описанный ниже, не очень хорошо подходит для работы с гистограммами.
Агрегирование до еженедельных подсчетов
# Создание набора данных о количестве случаев по неделям
#######################################
central_weekly <- linelist %>%
filter(hospital == "Central Hospital") %>% # фильтр построчного списка
mutate(week = lubridate::floor_date(date_onset, unit = "weeks")) %>%
count(week) %>% # обобщение еженедельных данных о количестве случаев
drop_na(week) %>% # удалить случаи с отсутствующей датой начала заболевания
complete( # заполнить все недели, в которые не было зарегистрировано ни одного случая
week = seq.Date(
from = min(week),
to = max(week),
by = "week"),
fill = list(n = 0)) # преобразование новых значений NA в 0 подсчетовСоздание графиков
# график без границы фасетного поля
#################################
ggplot(central_weekly,
aes(x = week, y = n)) + # установить x и y для всего графика
geom_line(stat = "identity", # создать линию, высота линии - число подсчета
color = "#69b3a2") + # цвет линии
geom_point(size=1, color="#69b3a2") + # нанести точки на еженедельные точки данных
geom_area(fill = "#69b3a2", # заливка области под линией
alpha = 0.4)+ # прозрачность заливки
scale_x_date(date_labels="%b", # формат метки даты показывает месяц
date_breaks="month", # метки с датой 1-го числа каждого месяца
expand=c(0,0)) + # удаление лишнего пространства
scale_y_continuous(
expand = c(0,0))+ # удалить лишнее пространство под осью x
facet_grid(~lubridate::year(week), # фасет по году (из столбца класса Дата)
space="free_x",
scales="free_x", # Оси x адаптируются к диапазону данных (не "фиксируются")
switch="x") + # фасетные метки (год) в нижней части
theme_bw() +
theme(strip.placement = "outside", # размещение фасетных меток
strip.background = element_blank(), # фон фасетной таблицы отсутствует
panel.grid.minor.x = element_blank(),
panel.border = element_blank(), # Границы у панели фасетов отсутствуют
panel.spacing=unit(0,"cm"))+ # Пространство между фасетными панелями отсутствует
labs(title = "Nested year labels - points, shaded, no label border")Приведенная выше техника фасетирования была адаптирована из сообщений здесь и здесь на сайте stackoverflow.com.
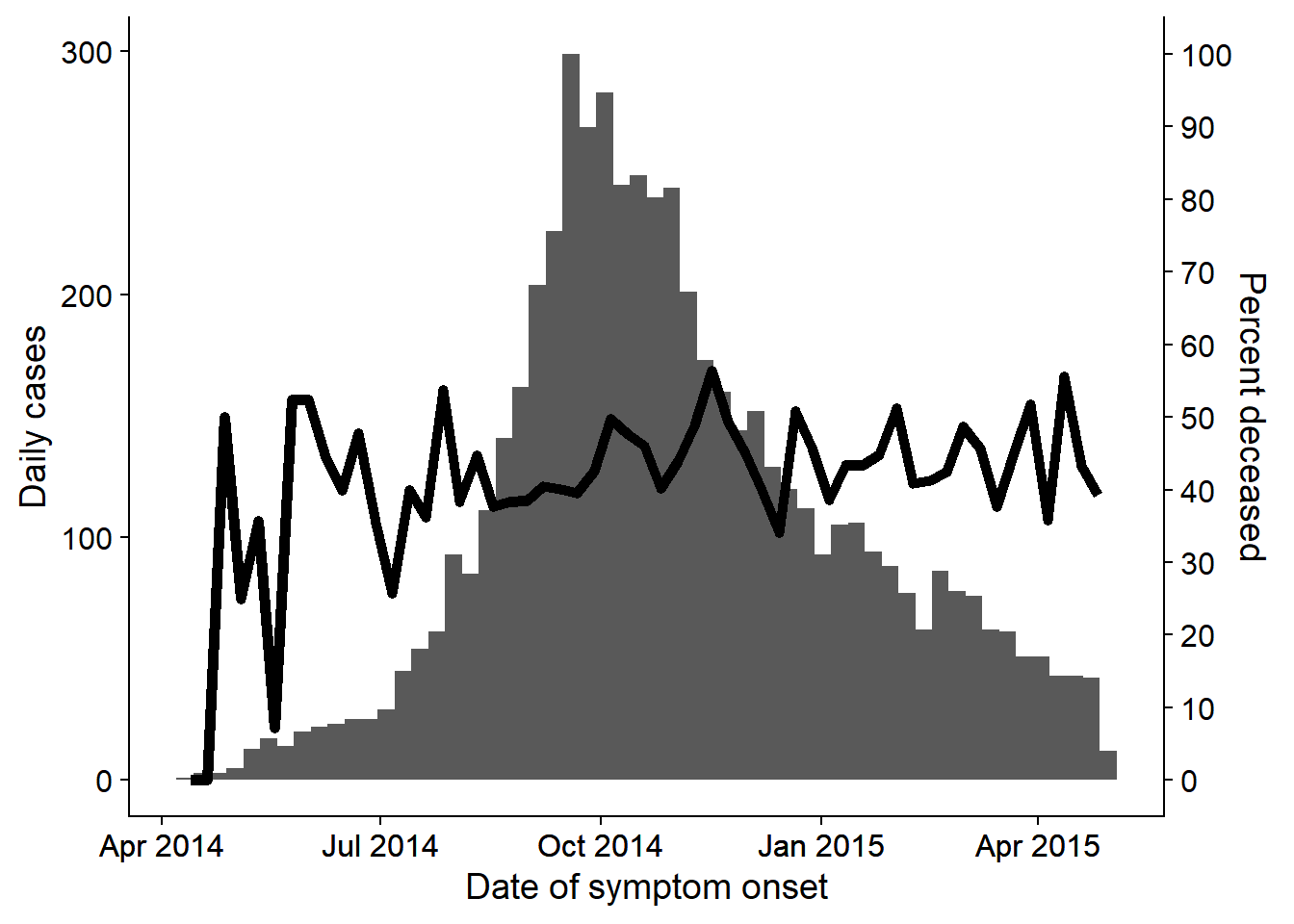
32.5 Двойные оси
Несмотря на то, что в сообществе визуализации данных ведутся ожесточенные дискуссии о правомерности использования двойных осей, многие руководители эпидемиологических исследований по-прежнему хотят использовать эпидкривую или подобный график с процентами, наложенными на вторую ось. Более подробно этот вопрос рассматривается на странице [Советы по использованию ggplot], но ниже приведен один пример с использованием метода cowplot:
- Строятся два разных графика, которые затем объединяются с помощью пакета cowplot.
- Графики должны иметь совершенно одинаковую ось x (заданные границы), иначе данные и метки не будут совпадать.
- В каждом из них используется
theme_cowplot(), причем в одном из них ось y перенесена в правую часть графика
#загрузка пакета
pacman::p_load(cowplot)
# Построить первый график гистограммы эпидкривой
#######################################
plot_cases <- linelist %>%
# Количество случаев в неделю
ggplot()+
# построить гистограмму
geom_histogram(
mapping = aes(x = date_onset),
# Разрывы корзины каждую неделю, начиная с понедельника перед первым случаем и заканчивая понедельником после последнего случая
breaks = weekly_breaks_all)+ # предварительно заданный вектор недельных дат (см. начало раздела ggplot)
# указать начало и конец оси дат для выравнивания с другими графиками
scale_x_date(
limits = c(min(weekly_breaks_all), max(weekly_breaks_all)))+ # минимум/максимум заданных недельных разрывов гистограммы
# метки
labs(
y = "Daily cases",
x = "Date of symptom onset"
)+
theme_cowplot()
# построить второй график процента умерших за неделю
###########################################
plot_deaths <- linelist %>% # начать с построчного списка
group_by(week = floor_date(date_onset, "week")) %>% # create week column
# Суммировать, чтобы получить еженедельный процент умерших
summarise(n_cases = n(),
died = sum(outcome == "Death", na.rm=T),
pct_died = 100*died/n_cases) %>%
# начать построение графика
ggplot()+
# строка еженедельного процента погибших
geom_line( # создать линию процента смертей
mapping = aes(x = week, y = pct_died), # указать высоту y в качестве столбца pct_died
stat = "identity", # установить высоту строки равной значению в столбце pct_death, а не количеству строк (которое задано по умолчанию)
size = 2,
color = "black")+
# Те же границы оси дат, что и на другом графике - идеальное выравнивание
scale_x_date(
limits = c(min(weekly_breaks_all), max(weekly_breaks_all)))+ # минимум/максимум заданных недельных разрывов гистограммы
# регулировки по оси y
scale_y_continuous( # отрегулировать ось y
breaks = seq(0,100, 10), # установка интервалов разрывов по оси процентов
limits = c(0, 100), # задать протяженность оси процентов
position = "right")+ # сдвинуть ось процентов вправо
# Метка по оси Y, метка по оси X отсутствует
labs(x = "",
y = "Percent deceased")+ # метка оси процентов
theme_cowplot() # добавьте это, чтобы два графика хорошо объединилисьТеперь используйте cowplot для наложения двух графиков. Внимание было уделено выравниванию по оси x, стороне оси y и использованию theme_cowplot().
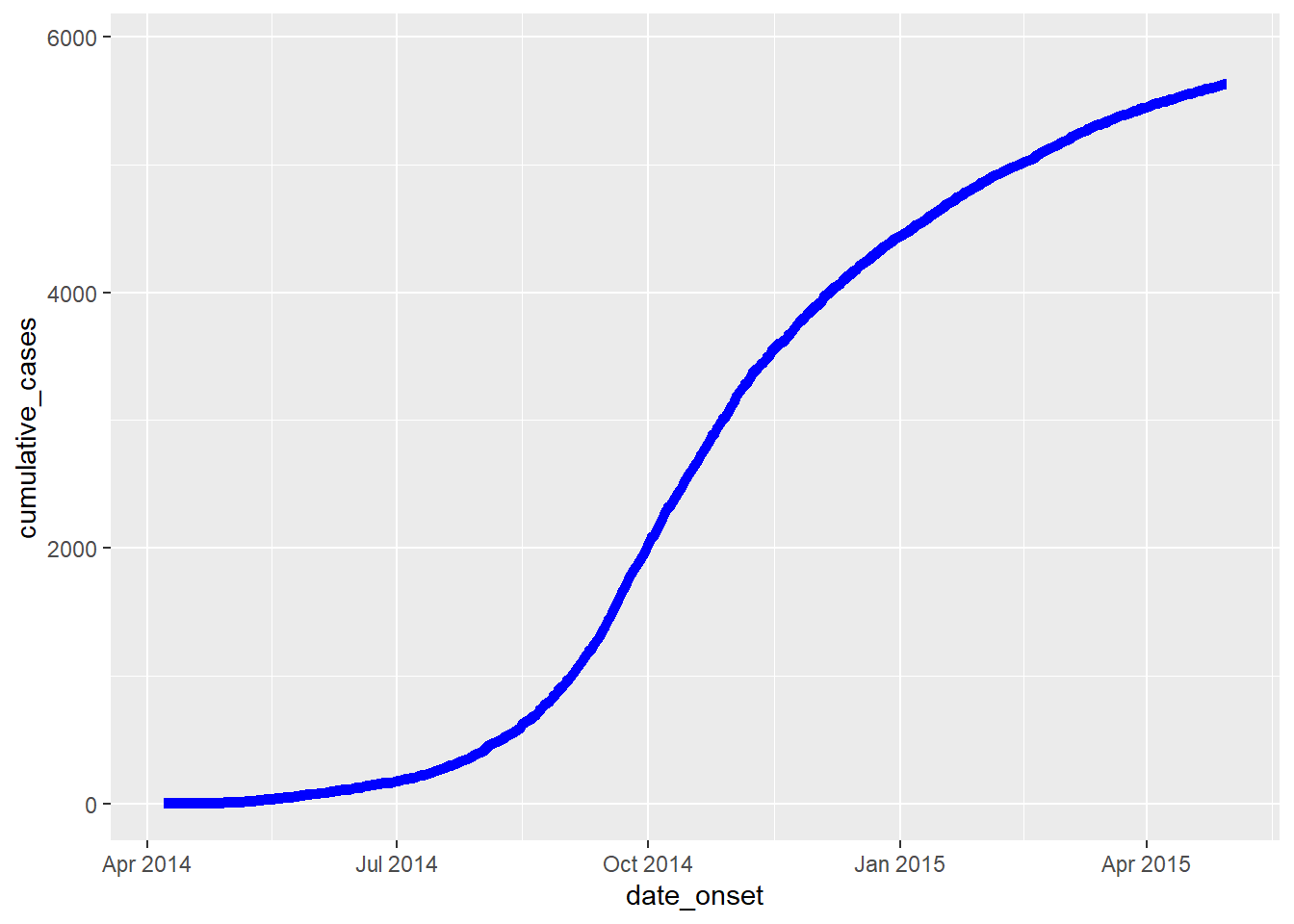
32.6 Кумулятивная заболеваемость
Примечание: При использовании incidence2 см. раздел о том, как можно получить кумулятивную заболеваемость с помощью простой функции. На этой странице мы рассмотрим, как рассчитать кумулятивную заболеваемость и построить ее с помощью функции ggplot().
Если вы начинаете с построчного списка случаев, создайте новый столбец, содержащий суммарное количество случаев за день во вспышке, используя функцию cumsum() из базового R:
cumulative_case_counts <- linelist %>%
count(date_onset) %>% # количество строк за день (возвращается в столбец "n")
mutate(
cumulative_cases = cumsum(n) # новый столбец суммарного количества строк на каждую дату
)Первые 10 строк показаны ниже:
Затем этот кумулятивный столбец может быть построен по отношению к date_onset с помощью функции geom_line():
plot_cumulative <- ggplot()+
geom_line(
data = cumulative_case_counts,
aes(x = date_onset, y = cumulative_cases),
size = 2,
color = "blue")
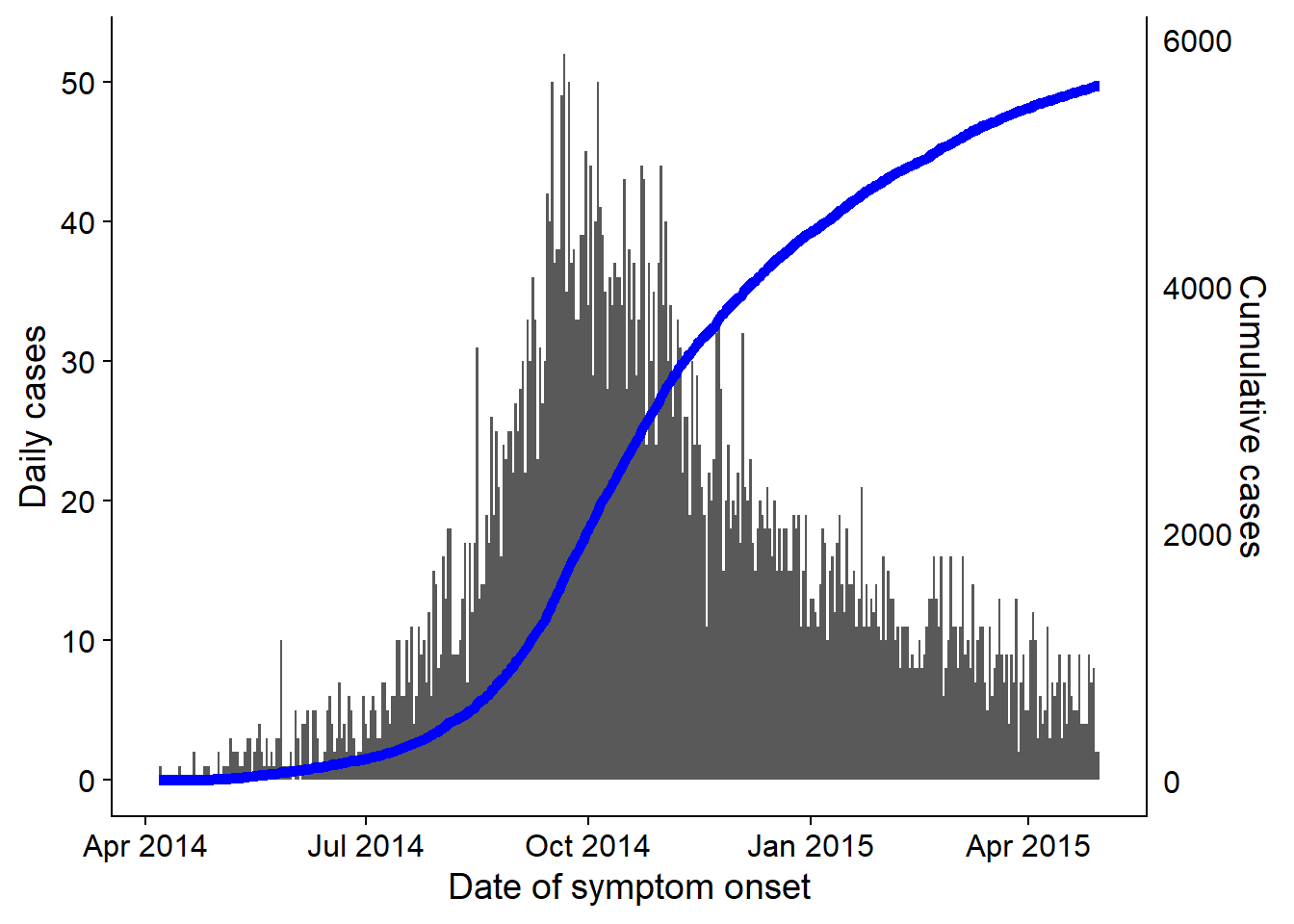
plot_cumulativeЕго также можно наложить на эпидкривую с двумя осями с помощью метода cowplot, описанного выше и на странице [Советы по использованию с ggplot]:
#загрузка пакета
pacman::p_load(cowplot)
# Построить первый график гистограммы эпидкривой
plot_cases <- ggplot()+
geom_histogram(
data = linelist,
aes(x = date_onset),
binwidth = 1)+
labs(
y = "Daily cases",
x = "Date of symptom onset"
)+
theme_cowplot()
# построить второй график линии кумулятивных случаев
plot_cumulative <- ggplot()+
geom_line(
data = cumulative_case_counts,
aes(x = date_onset, y = cumulative_cases),
size = 2,
color = "blue")+
scale_y_continuous(
position = "right")+
labs(x = "",
y = "Cumulative cases")+
theme_cowplot()+
theme(
axis.line.x = element_blank(),
axis.text.x = element_blank(),
axis.title.x = element_blank(),
axis.ticks = element_blank())Теперь используйте cowplot для наложения двух графиков. Внимание было уделено выравниванию по оси x, стороне оси y и использованию theme_cowplot().