8 Вычистка данных и ключевые функции
На данной странице представлены общие шаги процесса “вычистки” набора данных, а также объяснены многие важные функции управления данными в R.
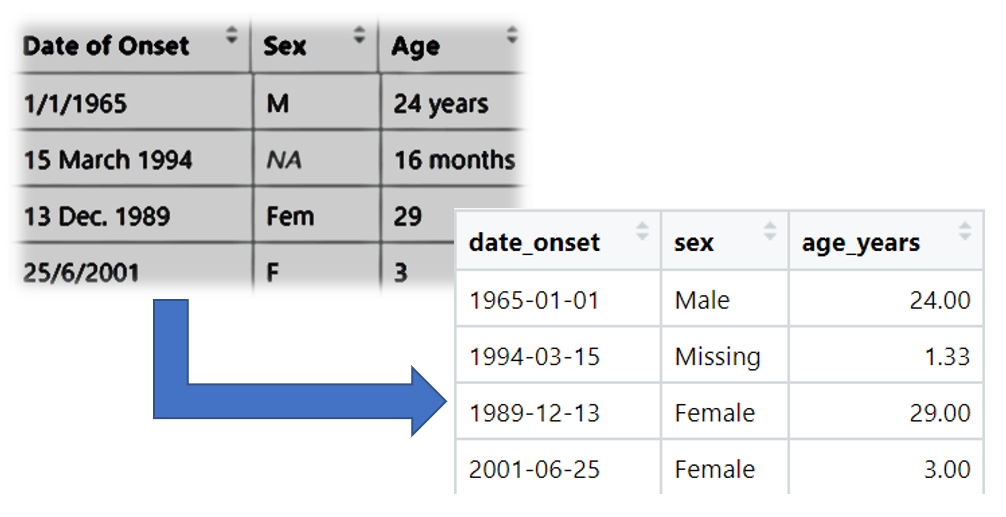
Чтобы продемонстрировать вычистку данных, для начала мы импортируем сырой набор данных с построчным списком, а затем пошагово разберем процесс вычистки. В коде R это будет выражено в “цепочке канала”, которая использует “оператор канала” %>%, который передает набор данных из одной операции в другую.
Ключевые функции
Это руководство подчеркивает использование функций из семьи пакетов R tidyverse. Основные функции R, представленные на данной странице, перечислены ниже.
Многие из этих функций относятся к пакету R dplyr, который предоставляет “глагольные” функции для решения проблем манипуляций с данными (название является отсылкой на “датафрейм-Plier - щипцы. dplyr является частью семьи пакетов R tidyverse (она также включает в себя ggplot2, tidyr, stringr, tibble, purrr, magrittr, forcats и другие).
| Функция | Использование | Пакет |
|---|---|---|
%>% |
“оператор канала” (передает) данные из одной функции в другую | magrittr |
mutate() |
создает, трансформирует и переопределяет столбцы | dplyr |
select() |
сохраняет, удаляет, выбирает или переименовывает столбцы | dplyr |
rename() |
переименовывает столбцы | dplyr |
clean_names() |
стандартизирует синтаксис названия столбцов | janitor |
as.character(), as.numeric(), as.Date(), и т.п. |
конвертирует класс столбца | базовый R |
across() |
трансформирует несколько столбцов одновременно | dplyr |
| tidyselect функции | используют логику для выбора столбцов | tidyselect |
filter() |
сохраняет определенные строки | dplyr |
distinct() |
дедупликация строк | dplyr |
rowwise() |
операции по строкам/внутри строки | dplyr |
add_row() |
добавление строк вручную | tibble |
arrange() |
сортировка строк | dplyr |
recode() |
перекодировка зеачений в столбце | dplyr |
case_when() |
перекодирует значения в столбце, используя более сложные логические критерии | dplyr |
replace_na(), na_if(), coalesce()
|
специальные функции для перекодирования | tidyr |
age_categories() и cut()
|
создает категорийные группы из числового столбца | epikit и базовый R |
match_df() |
перекодирует/вычищает значения, используя словарь данных | matchmaker |
which() |
применяет логические критерии; выдает индексы | базовый R |
Если вы хотите увидеть сравнение этих функций с командами в Stata или SAS, см. страницу [Переход к R]((transition_to_R.ru.qmd).
Вы можете столкнуться с альтернативной системой управления данными из пакета R data.table с такими операторами, как := и частым использованием квадратных скобок [ ]. Такой подход и синтаксис кратко объясняются на странице Таблицы данных.
Номенклатура
В данном руководстве мы, как правило, используем понятия “столбцы” и “строки” вместо “переменных” и “наблюдений”. Как объясняется в данном пособии по “аккуратным данным”, большинство эпидемиологических статистических наборов данных структурно состоят из строк, столбцов и значений.
Переменные содержат значения, которые измеряют одинаковую базовую характеристику (например, возрастную группу, исход или дату появления симптомов). Наблюдения содержат все значения, измеренные по той же единице (например, по человеку, месту или лабораторному образцу). Так что эти аспекты может быть сложно определить очень конкретно.
В “аккуратных” наборах данных каждый столбец представляет переменную, каждая строка - наблюдения, а каждая ячейка - одно значение. Однако в некоторых наборах данных, с которыми вы столкнетесь, эта схема не будет соблюдена. Например, в “широком/горизонтальном” наборе данных переменная может быть разделена на несколько столбцов (см. пример на странице Поворот данных). Аналогично, наблюдения могут быть разделены на несколько строк.
Большая часть этого руководства посвящена управлению и преобразованию данных, поэтому ссылка на более конкретные структура строк и столбцов более актуальна, чем на более абстрактные понятия, такие как наблюдения и переменные. Есть исключения на страницах, посвященных анализу данных, где вы будете чаще встречать такие понятия, как переменные и наблюдения.
8.1 Канал вычистки
На этой странице представлены типичные шаги по вычистке данных, и они последовательно добавляются в канал вычистки.
В эпидемиологическом анализе и обработке данных шаги по вычистке часто проводятся последовательно и связываются вместе. В R это часто выглядит как “цепочка канала”, где сырой набор данных передается или “ставится в канал” из одного шага вычистки в другой.
Такие цепочки используют “глагольные функции” dplyr и оператор канала из magrittr %>%. Этот канал начинается с “сырых” данных (“linelist_raw.xlsx”) и оканчивается “чистым” датафреймом в R (linelist), который можно использовать, сохранять, экспортировать и т.п.
В цепочке канала вычистки очень важен порядок шагов. Шаги по вычистке могут включать:
- Импорт данных
- Вычистка или изменение названия столбцов
- Дедупликация
- Создание столбцов и трансформация (например, перекодирование или стандартизация значений)
- Фильтрация или добавления строк
8.2 Загрузка пакетов
Данный фрагмент кода показывает загрузку пакетов, требуемых для анализа. В данном руководстве мы полагаемся на p_load() из pacman, который устанавливает пакеты, если необходимо, и загружает их для использования. Вы также можете загружать установленные пакеты с помощью library() из базового R. См. страницу Основы R для получения более подробной информации о пакетах R.
pacman::p_load(
rio, # импорт данных
here, # относительные пути к файлу
janitor, # вычистка данных и таблицы
lubridate, # работа с датами
matchmaker, # вычистка на основе словаря
epikit, # функция age_categories() (возрастные категории)
tidyverse # управление данными и визуализация
)8.3 Импорт данных
Импорт
Здесь мы импортируем “сырой” Excel файл с построчным списком, используя функцию import() из пакета rio. Пакет rio гибко работает с многими типами файлов (например, .xlsx, .csv, .tsv, .rds. См. страницу Импорт и экспорт для получения более подробной информации и советов по необычным ситуациям (например, пропуск строк, установка отсутствующих значений, импорт Google таблиц и т.п.).
Если вы хотите параллельно повторять эти действия, кликните для скачивания “сырого” построчного списка (as .xlsx file).
Если ваш набор данных большой и требует много времени для импортирования, может быть полезно сделать импорт командой, отдельной от цепочки канала и сохранить “сырой” набор данных как отдельный файл. Это позволит легко проводить сопоставление между оригиналом и вычищенными версиями.
Ниже мы импортируем сырой Excel файл и сохраняем его как датафрейм linelist_raw. Мы предполагаем, что файл расположен в вашей рабочей директории или корневой папке проекта R, поэтому в пути к файлу не уточняются подпапки.
linelist_raw <- import("linelist_raw.xlsx")Вы можете видеть первые 50 строк датафрейма ниже. Примечание: базовая функция R head(n) позволяет вам просматривать только первое n количество строк в консоли R.
Обзор
Вы можете использовать функцию skim() из пакета skimr, чтобы сделать обзор всего датафрейма (см. страницу Описательные таблицы для получения более детальной информации). Столбцы резюмируются по классу/типу, например, текстовые, числовые. Примечание: “POSIXct” - это тип класса сырых данных (см. Работа с датами.
skimr::skim(linelist_raw)| Name | linelist_raw |
| Number of rows | 6611 |
| Number of columns | 28 |
| _______________________ | |
| Column type frequency: | |
| character | 17 |
| numeric | 8 |
| POSIXct | 3 |
| ________________________ | |
| Group variables | None |
Variable type: character
| skim_variable | n_missing | complete_rate | min | max | empty | n_unique | whitespace |
|---|---|---|---|---|---|---|---|
| case_id | 137 | 0.98 | 6 | 6 | 0 | 5888 | 0 |
| date onset | 293 | 0.96 | 10 | 10 | 0 | 580 | 0 |
| outcome | 1500 | 0.77 | 5 | 7 | 0 | 2 | 0 |
| gender | 324 | 0.95 | 1 | 1 | 0 | 2 | 0 |
| hospital | 1512 | 0.77 | 5 | 36 | 0 | 13 | 0 |
| infector | 2323 | 0.65 | 6 | 6 | 0 | 2697 | 0 |
| source | 2323 | 0.65 | 5 | 7 | 0 | 2 | 0 |
| age | 107 | 0.98 | 1 | 2 | 0 | 75 | 0 |
| age_unit | 7 | 1.00 | 5 | 6 | 0 | 2 | 0 |
| fever | 258 | 0.96 | 2 | 3 | 0 | 2 | 0 |
| chills | 258 | 0.96 | 2 | 3 | 0 | 2 | 0 |
| cough | 258 | 0.96 | 2 | 3 | 0 | 2 | 0 |
| aches | 258 | 0.96 | 2 | 3 | 0 | 2 | 0 |
| vomit | 258 | 0.96 | 2 | 3 | 0 | 2 | 0 |
| time_admission | 844 | 0.87 | 5 | 5 | 0 | 1091 | 0 |
| merged_header | 0 | 1.00 | 1 | 1 | 0 | 1 | 0 |
| …28 | 0 | 1.00 | 1 | 1 | 0 | 1 | 0 |
Variable type: numeric
| skim_variable | n_missing | complete_rate | mean | sd | p0 | p25 | p50 | p75 | p100 |
|---|---|---|---|---|---|---|---|---|---|
| generation | 7 | 1.00 | 16.60 | 5.71 | 0.00 | 13.00 | 16.00 | 20.00 | 37.00 |
| lon | 7 | 1.00 | -13.23 | 0.02 | -13.27 | -13.25 | -13.23 | -13.22 | -13.21 |
| lat | 7 | 1.00 | 8.47 | 0.01 | 8.45 | 8.46 | 8.47 | 8.48 | 8.49 |
| row_num | 0 | 1.00 | 3240.91 | 1857.83 | 1.00 | 1647.50 | 3241.00 | 4836.50 | 6481.00 |
| wt_kg | 7 | 1.00 | 52.69 | 18.59 | -11.00 | 41.00 | 54.00 | 66.00 | 111.00 |
| ht_cm | 7 | 1.00 | 125.25 | 49.57 | 4.00 | 91.00 | 130.00 | 159.00 | 295.00 |
| ct_blood | 7 | 1.00 | 21.26 | 1.67 | 16.00 | 20.00 | 22.00 | 22.00 | 26.00 |
| temp | 158 | 0.98 | 38.60 | 0.95 | 35.20 | 38.30 | 38.80 | 39.20 | 40.80 |
Variable type: POSIXct
| skim_variable | n_missing | complete_rate | min | max | median | n_unique |
|---|---|---|---|---|---|---|
| infection date | 2322 | 0.65 | 2012-04-09 | 2015-04-27 | 2014-10-04 | 538 |
| hosp date | 7 | 1.00 | 2012-04-20 | 2015-04-30 | 2014-10-15 | 570 |
| date_of_outcome | 1068 | 0.84 | 2012-05-14 | 2015-06-04 | 2014-10-26 | 575 |
8.4 Имена столбцов
В R имена столбцов - это “заголовок” или “верхнее” значение в столбце. Они используются, чтобы ссылаться на столбцы в коде, а также являются значеними по умолчанию в подписях к диаграммам.
Другие статистические программы, такие как SAS и STATA используют “подписи”, которые существуют параллельно как более длинные печатные версии коротких имен столбцов. Хотя в R и есть возможность добавлять подписи столбцов к данным, на практике это не часто применяется. Чтобы сделать имена столбцов более удобными для печати графиков, как правило, их отображение корректируется в рамках команд для построения диаграммы, которые создают выходные данные (например, подписи осей или легенду для графика или заголовки столбцов в печатной таблице - см. раздел по шкалам на странице с советами по использованию ggplot и страницу Таблицы для презентации). Если вы хотите присвоить подписи столбцов в данных, более детально прочитайте об этом тут и тут.
Так как имена столбцов очень часто используются в R, они должны иметь “чистый” синтаксис. Мы рекомендуем следующее:
- Короткие имена
- Без пробелов (замените пробелы на нижние подчеркивания _ )
- Без специальных знаков (&, #, <, >, …)
- Номенклатура в едином стиле (например, все столбцы с датами названы по принципу дата_заболевания, дата_регистрации, дата_смерти)
Названия столбцов набора данных linelist_raw напечатаны ниже с помощью names() из базового R. Мы можем увидеть, что изначально:
- Некоторые имена содержат пробелы (например,
infection date)
- Для дат используются разные принципы именования (
date onsetилиinfection date)
- Наверное, был объединенный заголовок в двух последних столбцах в .xlsx. Мы это знаем, поскольку имя двух объединенных столбцов (“merged_header”) было просто присвоено R первому столбцу, а второму столбцу было присвоено временное имя “…28” (поскольку он был пустым и это 28й столбец).
names(linelist_raw) [1] "case_id" "generation" "infection date" "date onset"
[5] "hosp date" "date_of_outcome" "outcome" "gender"
[9] "hospital" "lon" "lat" "infector"
[13] "source" "age" "age_unit" "row_num"
[17] "wt_kg" "ht_cm" "ct_blood" "fever"
[21] "chills" "cough" "aches" "vomit"
[25] "temp" "time_admission" "merged_header" "...28" ПРИМЕЧАНИЕ: Чтобы ссылаться на имя столбца, которое содержит пробелы, заключите это имя в обратные одинарные кавычки, например: linelist$` '\x60infection date\x60'`. Обратите внимание, что на клавиатуре обратная кавычка (`) отличается от простой одинарной кавычки (’).
Автоматическая вычистка
Функция clean_names() из пакета janitor стандартизирует имена столбцов и делает их уникальными следующим образом:
- Конвертирует все имена так, чтобы они содержали только нижние подчеркивания, цифры и буквы
- Символы с диакритическими знаками транслитерируются в ASCII (например, немецкая о умлаут становится “o”, испанская “энье” становится “n”)
- Предпочтения по капитализации для новых имен столбцов можно указать, используя аргумент
case =(“snake” это значение по умолчанию, альтернативами могут быть “sentence”, “title”, “small_camel”…)
- Вы можете уточнить конкретную замену имен, указав вектор в аргументе
replace =(например,replace = c(onset = "date_of_onset"))
- Здесь есть онлайн виньетка
Ниже начинается цепочка канала вычистки, мы используем clean_names() для сырого построчного списка.
# подставляем сырой набор данных через функцию clean_names(), присваиваем результату имя "linelist"
linelist <- linelist_raw %>%
janitor::clean_names()
# смотрим новые имена столбцов
names(linelist) [1] "case_id" "generation" "infection_date" "date_onset"
[5] "hosp_date" "date_of_outcome" "outcome" "gender"
[9] "hospital" "lon" "lat" "infector"
[13] "source" "age" "age_unit" "row_num"
[17] "wt_kg" "ht_cm" "ct_blood" "fever"
[21] "chills" "cough" "aches" "vomit"
[25] "temp" "time_admission" "merged_header" "x28" ПРИМЕЧАНИЕ: Имя последнего столбца “…28” было изменено на “x28”.
Вычистка имен вручную
Часто даже после шага стандартизации, описанного выше, может потребоваться переименовать имена столбцов вручную. Ниже производится переименование с помощью функции rename() из пакета dplyr в рамках цепочки канала. rename() использует стиль NEW = OLD - новое имя столбца приводится до старого имени столбца.
Ниже к цепочке канала вычистки добавляется команда переименования. Пробелы добавляются стратегическим образом, чтобы выровнять код для облегчения его чтения.
# ЦЕПОЧКА КАНАЛА ВЫЧИСТКИ (начинается с сырых данных, которые подставляются в шаги по вычистке)
##################################################################################
linelist <- linelist_raw %>%
# стандартизируем синтаксис имен столбцов
janitor::clean_names() %>%
# вручную переименовываем столбцы
# НОВОЕ имя # СТАРОЕ имя
rename(date_infection = infection_date,
date_hospitalisation = hosp_date,
date_outcome = date_of_outcome)Вы можете увидеть, что изменились имена столбцов:
[1] "case_id" "generation" "date_infection"
[4] "date_onset" "date_hospitalisation" "date_outcome"
[7] "outcome" "gender" "hospital"
[10] "lon" "lat" "infector"
[13] "source" "age" "age_unit"
[16] "row_num" "wt_kg" "ht_cm"
[19] "ct_blood" "fever" "chills"
[22] "cough" "aches" "vomit"
[25] "temp" "time_admission" "merged_header"
[28] "x28" Переименование по положению столбца
Вы можете также делать переименование по положению столбца вместо имени столбца, например:
rename(newNameForFirstColumn = 1,
newNameForSecondColumn = 2)Переименование с помощью select() и summarise()
В качестве быстрого способа вы также можете переименовывать столбцы с помощью функций select() и summarise() из dplyr. select() используется, чтобы сохранить только некоторые столбцы (она описана ниже на этой странице). Функция summarise() разбирается на страницах Группирование данных и Описательные таблицы. Эти функции также используют формат new_name = old_name. Пример:
linelist_raw %>%
select(# НОВОЕ имя # СТАРОЕ имя
date_infection = `infection date`, # переименовать и СОХРАНИТЬ ТОЛЬКО эти столбцы
date_hospitalisation = `hosp date`)Прочие сложности
Пустые имена столбцов в Excel
В R не может быть столбцов в наборе данных без имени столбца (заголовка). Поэтому если вы импортируете набор данных Excel с данными, но без заголовков столбцов, R заполнит эти заголовки названиями как “…1” или “…2”. Цифра представляет собой номер столбца (например, 4-й столбец в наборе данных не имеет заголовка, тогда R назовет его “…4”).
Вы можете вычистить эти имена вручную, указав номер их положения (см. пример выше), либо присвоенное им имя (linelist_raw$...1).
Объединенные имена столбцов и ячейки в Excel
Объединенные ячейки в Excel файле встречаются довольно часто в полученных данных. Как объясняется на странице Переход к R, объединенные ячейки могут быть более удобны для чтения данных человеком, но не являются “аккуратными данными” и могут вызввать много проблем для машинного чтения данных. R не может справиться с объединенными ячейками.
Напоминайте людям, осуществляющим ввод данных, что данные, читаемые человеком - не то же самое, что машиночитаемые данные. Стремитесь обучать пользователей принципам аккуратных данных. Если возможно, попробуйте изменить процедуры, чтобы данные поступали в аккуратном формате без объединенных ячеек.
- У каждой переменной должен быть свой столбец.
- У каждого наблюдения должна быть своя строка.
- У каждого значения должна быть своя ячейка.
При использовании функции import() из rio, значение в объединенной ячейке будет присвоено первой ячейке, а последующие ячейки останутся пустыми.
Одно из решений для проблемы объединенных ячеек - импортировать данные с помощью функции readWorkbook() из пакета openxlsx. Задайте аргумент fillMergedCells = TRUE. Это задает значение из объединенной ячейки для всех ячеек в объединенном диапазоне.
linelist_raw <- openxlsx::readWorkbook("linelist_raw.xlsx", fillMergedCells = TRUE)ВНИМАНИЕ: Если имена столбцов объединены с помощью readWorkbook(), у вас получатся дублирующиеся имена столбцов, которые вам нужно будет исправить вручную - R не очень хорошо справляется с дублирующимися именами столбцов! Вы можете переименовать их, ссылаясь на их положение (например, столбец 5), как говорится в разделе по вычистке имен столбцов вручную.
8.5 Выбор или смена порядка столбцов
Используйте select() из пакета dplyr для выбора столбцов, которые вы хотите сохранить, а также для уточнения их порядка в датафрейме.
ВНИМАНИЕ: В примерах ниже датафрейм linelist модифицируется с помощью select() и отображается, но не сохраняется. Это сделано в целях демонстрации. Модифицированные имена столбцов печатаются с помощью передачи датафрейм в канале в функцию names().
Здесь представлены ВСЕ имена столбцов в построчном списке на этом этапе цепочки канала вычистки:
names(linelist) [1] "case_id" "generation" "date_infection"
[4] "date_onset" "date_hospitalisation" "date_outcome"
[7] "outcome" "gender" "hospital"
[10] "lon" "lat" "infector"
[13] "source" "age" "age_unit"
[16] "row_num" "wt_kg" "ht_cm"
[19] "ct_blood" "fever" "chills"
[22] "cough" "aches" "vomit"
[25] "temp" "time_admission" "merged_header"
[28] "x28" Сохранение столбцов
Выберите только те столбцы, которые вы хотите сохранить
Вставьте их имена в команду select() без кавычек. Они появятся в датафрейме в том порядке, который вы указали. Обратите внимание, что если вы включите имя несуществующего столбца, R выдаст ошибку (см. использование any_of() ниже, если вы хотите избежать ошибок в этой ситуации).
# набор данных linelist передается в канал в команду select(), а names() выводит на печать только имена столбцов
linelist %>%
select(case_id, date_onset, date_hospitalisation, fever) %>%
names() # отображает имена столбцов[1] "case_id" "date_onset" "date_hospitalisation"
[4] "fever" Функции-помощники “tidyselect”
Эти функции-помощники существуют, чтобы упростить уточнение того, какие столбцы сохранить, убрать или преобразовать. Это функции из пакета tidyselect, который включен в tidyverse и лежит в основе того, как столбцы выбираются в функциях dplyr.
Например, если вы хотите изменить порядок столбцов, полезной функцией является everything(), которая означает “все другие еще не упомянутые столбцы”. Команда ниже перемещает столбцы date_onset и date_hospitalisation в начало (влево) набора данных, но сохраняет все другие столбцы после. Обратите внимание, что everything() записана с пустыми скобками:
# перемещает date_onset и date_hospitalisation в начало
linelist %>%
select(date_onset, date_hospitalisation, everything()) %>%
names() [1] "date_onset" "date_hospitalisation" "case_id"
[4] "generation" "date_infection" "date_outcome"
[7] "outcome" "gender" "hospital"
[10] "lon" "lat" "infector"
[13] "source" "age" "age_unit"
[16] "row_num" "wt_kg" "ht_cm"
[19] "ct_blood" "fever" "chills"
[22] "cough" "aches" "vomit"
[25] "temp" "time_admission" "merged_header"
[28] "x28" Ниже представлены другие функции-помощники “tidyselect”, которые также работают внутри функций dplyr, такие как select(), across() и summarise():
-
everything()- все другие не упомянутые столбцы
-
last_col()- последний столбец
-
where()- применяет функцию ко всем столбцам и выбирает, какие из них являются ИСТИНОЙ
-
contains()- столбцы, содержащие последовательность знаков- пример:
select(contains("time"))
- пример:
-
starts_with()- соответствует упомянутому префиксу- пример:
select(starts_with("date_"))
- пример:
-
ends_with()- соответствует упомянутому суффиксу- пример:
select(ends_with("_post"))
- пример:
-
matches()- для применения регулярного выражения (regex)- пример:
select(matches("[pt]al"))
- пример:
-
num_range()- числовой диапазон, например, x01, x02, x03
-
any_of()- соответствует ЕСЛИ столбец существует, но не выдает ошибку, если он не найден- пример:
select(any_of(date_onset, date_death, cardiac_arrest))
- пример:
Кроме того, используйте обычные операторы, например, c(), чтобы перечислить несколько столбцов, : для последовательно расположенных столбцов, ! для противоположных, & для И, и | для ИЛИ.
Используйте where(), чтобы уточнить логические критерии для столбцов. Если вы представляете функцию внутри where(), не включайте пустые скобки функции. Команда ниже выбирает столбцы с числовым классом.
# выбирает столбцы в числовом классе
linelist %>%
select(where(is.numeric)) %>%
names()[1] "generation" "lon" "lat" "row_num" "wt_kg"
[6] "ht_cm" "ct_blood" "temp" Используйте contains(), чтобы выбрать только те столбцы, в которых название столбцы содержит конкретную последовательность знаков. ends_with() и starts_with() дадут вам большую детализацию.
# выбирает столбцы, содержащие определенные знаки
linelist %>%
select(contains("date")) %>%
names()[1] "date_infection" "date_onset" "date_hospitalisation"
[4] "date_outcome" Функция matches() работает аналогично функции contains(), но ей может быть задано регулярное выражение (см. страницу Текст и последовательности), например, несколько последовательностей, разделенных чертами со значением ИЛИ внутри скобок:
# поиск нескольких соответствий знаков
linelist %>%
select(matches("onset|hosp|fev")) %>% # обратите внимание на символ ИЛИ "|"
names()[1] "date_onset" "date_hospitalisation" "hospital"
[4] "fever" ВНИМАНИЕ: Если имя столбца, которое вы задали, не существует в данных, вам будет выдана ошибка, а выполнение кода будет остановлено. Рассмотрите возможность использования any_of(), чтобы перечислить столбцы, которые, возможно, не существуют, что особенно полезно при отрицательном выборе (удалении).
Существует только один из этих столбцов, но программа не выдает ошибку, и выполнение кода продолжается без остановки канала вычистки.
linelist %>%
select(any_of(c("date_onset", "village_origin", "village_detection", "village_residence", "village_travel"))) %>%
names()[1] "date_onset"Удаление столбцов
Укажите, какие столбцы удалить, поставив знак минус “-” перед именем столбца (например, select(-outcome)), или вектором имен столбцов (как показано ниже). Все другие столбцы будут сохранены.
linelist %>%
select(-c(date_onset, fever:vomit)) %>% # удаляет date_onset и все столбцы от fever до vomit
names() [1] "case_id" "generation" "date_infection"
[4] "date_hospitalisation" "date_outcome" "outcome"
[7] "gender" "hospital" "lon"
[10] "lat" "infector" "source"
[13] "age" "age_unit" "row_num"
[16] "wt_kg" "ht_cm" "ct_blood"
[19] "temp" "time_admission" "merged_header"
[22] "x28" Вы можете также удалить столбец, используя базовый синтаксис R, определив его как NULL. Например:
linelist$date_onset <- NULL # удаление столбца с помощью базового синтаксиса R Отдельная команда
select() также может использоваться как независимая команда (не как часть канала). В таком случае первым аргументом будет изначальный датафрейм, с которым проводится операция.
# Создание нового построчного списка со столбцами с идентификатором и возрастом
linelist_age <- select(linelist, case_id, contains("age"))
# отображение имен столбцов
names(linelist_age)[1] "case_id" "age" "age_unit"Добавление в цепочку канала
В linelist_raw, существует несколько столбцов, которые нам не нужны: row_num, merged_header и x28. Мы удаляем их с помощью команды select() в цепочке канала вычистки:
# ЦЕПОЧКА КАНАЛА ВЫЧИСТКИ (начинается с сырых данных, которые передаются по шагам вычистки)
##################################################################################
# начало цепочки канала вычистки
###########################
linelist <- linelist_raw %>%
# стандартизируем синтаксис имени столбца
janitor::clean_names() %>%
# переименовываем столбцы вручную
# НОВОЕ имя # СТАРОЕ имя
rename(date_infection = infection_date,
date_hospitalisation = hosp_date,
date_outcome = date_of_outcome) %>%
# ВЫШЕ ПОКАЗАНЫ РАНЕЕ ПРОДЕЛАННЫЕ И ПРЕДСТАВЛЕННЫЕ ШАГИ ВЫЧИСТКИ
#####################################################
# удаляем столбцы
select(-c(row_num, merged_header, x28))8.6 Дедупликация
См. страницу руководства Дедупликация, где показан широкий спектр вариантов дедупликации данных. Здесь представлен только простой пример дедупликации строки.
Пакет dplyr предлагает функцию distinct(). Эта функция рассматривает каждую строку и сокращает датафрейм только до уникальных строк. То есть, она удаляет те строки, которые являются 100% дубликатами.
При оценке дублирующихся строк она принимает во внимание диапазон столбцов - по умолчанию рассматриваются все столбцы. Как показано на странице по дедупликации, вы можете скорректировать диапазон столбцов, чтобы уникальность строк оценивуалась только по определенным столбцам.
В этом простом примере мы просто добавляем пустую команду distinct() в цепочку канала. Она сделает так, что не останется строк, которые являются 100% дубликатами других строк (оценивается по всем столбцам).
Мы начинаем с nrow(linelist) строк в linelist.
linelist <- linelist %>%
distinct()После дедупликации у нас осталось nrow(linelist) строк. Любые удаленные строки были 100% дубликатами других строк.
Ниже в цепочку канала добавляется команда distinct():
# ЦЕПОЧКА КАНАЛА ВЫЧИСТКИ (начинается с сырых данных, которые передаются по шагам вычистки)
##################################################################################
# начало цепочки канала вычистки
###########################
linelist <- linelist_raw %>%
# стандартизируем синтаксис имен столбцов
janitor::clean_names() %>%
# переименовываем столбцы вручную
# НОВОЕ имя # СТАРОЕ имя
rename(date_infection = infection_date,
date_hospitalisation = hosp_date,
date_outcome = date_of_outcome) %>%
# удаляем столбцы
select(-c(row_num, merged_header, x28)) %>%
# ВЫШЕ ПОКАЗАНЫ РАНЕЕ ПРОДЕЛАННЫЕ И ПРЕДСТАВЛЕННЫЕ ШАГИ ВЫЧИСТКИ
#####################################################
# дедупликация
distinct()8.7 Создание и преобразование столбцов
Мы рекомендуем использовать функцию mutate() из dplyr, чтобы добавить новый столбец или преобразовать существующий.
Ниже представлен пример создания нового столбца с помощью mutate(). Синтаксис: mutate(новое_имя_столбца = значение или преобразование)
В Stata есть похожая команда generate, но функцию R mutate() можно также использовать для модификации существующего столбца.
Новые столбцы
Самая простая команда mutate() для создания нового столбца может выглядеть следующим образом. Она создает новый столбец new_col, где значения в каждой строке - 10.
linelist <- linelist %>%
mutate(new_col = 10)Вы также можете ссылаться на значения в других столбцах для проведения расчетов. Ниже создается столбец bmi, который должен содержать индекс массы тела (ИМТ) для каждого случая, он рассчитывается по формуле ИМТ = кг/м^2, используя столбец ht_cm и столбец wt_kg.
linelist <- linelist %>%
mutate(bmi = wt_kg / (ht_cm/100)^2)Если вы создаете несколько новых столбцов, отделите каждый запятой и новой строкой. Ниже представлены некоторые примеры новых столбцов, включая те, которые состоят из значений из других столбцов, объединенных с помощью str_glue() из пакета stringr (см. страницу Текст и последовательности.
new_col_demo <- linelist %>%
mutate(
new_var_dup = case_id, # новый столбец = дубликат/копия другого существующего столбца
new_var_static = 7, # новый столбец = все значения такие же
new_var_static = new_var_static + 5, # вы можете переписать столбец, это может быть расчет с использованием других переменных
new_var_paste = stringr::str_glue("{hospital} on ({date_hospitalisation})") # новый столбец = склейка значений других столбцов
) %>%
select(case_id, hospital, date_hospitalisation, contains("new")) # показывает только новые столбцы в демонстрационных целяхРассмотрите новые столбцы. В демонстрационных целях показаны только новые столбцы и столбцы, использованные для их создания:
СОВЕТ: Вариацией mutate() является функция transmute(). Эта функция добавляет новый столбец, как и mutate(), но также выкидывает/удаляет все другие столбцы, которые вы не упомянули внутри скобок.
# СПРЯТАНО ОТ ЧИТАТЕЛЯ
# удаляем новые демонстрационные столбцы, созданные выше
# linelist <- linelist %>%
# select(-contains("new_var"))Конвертация класса столбцов
Столбцы, содержащие значения, которые являются датами, числами или логическими значениями (ИСТИНА/ЛОЖЬ) будут вести себя в соответствии с ожиданиями, только если они правильно классифицированы. Есть разница между “2” в текстовом классе и 2 в числовом классе!
Существуют способы, чтобы задать класс столбцов в командах импорта, но это часто сложно сделать. См. раздел Основы R по классам объектов, чтобы получить дополнительную информацию о конвертации класса объектов и столбцов.
Сначала давайте проведем некоторые проверки важных столбцов, чтобы посмотреть, к правильному классу ли они относятся. Мы это также видели в начале, когда выполняли команду skim().
В настоящее время столбец age относится к текстовому классу. Чтобы провести количественный анализ, нам нужно, чтобы цифры распознавались как числовой класс!
class(linelist$age)[1] "character"Класс столбца date_onset также является текстовым! Для проведения анализа даты должны распознаваться как даты!
class(linelist$date_onset)[1] "character"Чтобы устранить эту проблему, используйте возможность функции mutate() по переопределению столбца с трансформацией. Мы определяем столбец как самого себя, но конвертируем его в другой класс. Здесь приведен базовый пример конвертации, чтобы класс столбца age был числовым:
linelist <- linelist %>%
mutate(age = as.numeric(age))Аналогичным образом вы можете использовать as.character() и as.logical(). Чтобы конвертировать класс в Фактор, вы можете использовать factor() из базового R или as_factor() из forcats. Более подробно вы можете об этом узнать на странице Факторы.
Вы должны быть осторожны при конвертации в класс Дата. На странице Работа с датами объясняются несколько методов. Как правило, сырые значения даты должны быть в одинаковом формате, чтобы конвертация прошла правильно (например, “ММ/ДД/ГГГГ”, или “ДД ММ ГГГГ”). После конвертации в класс Дата, проверьте ваши данные, чтобы подтвердить, что каждое значение было сконвертировано верно.
Сгруппированные данные
Если ваш датафрейм уже сгруппирован (см. страницу Группирование данных), mutate() может повести себя иначе, чем в наборе данных без группировки. Любые резюмирующие функции, такие как mean() (среднее значение), median() (медиана), max() (максимум) и т.п. будут рассчитаны по группе, а не по всем строкам.
# возраст нормализован по среднему значению для ВСЕХ строк
linelist %>%
mutate(age_norm = age / mean(age, na.rm=T))
# возраст нормализован по среднему значению по группе больниц
linelist %>%
group_by(hospital) %>%
mutate(age_norm = age / mean(age, na.rm=T))Более подробно об использовании mutate () в сгруппированных датафреймах можно почитать в этой документации по tidyverse mutate.
Трансформация нескольких столбцов
Часто чтобы написать краткий код, вы можете захотеть применить одинаковую трансформацию сразу к нескольким столбцам. Трансформацию можно применить одновременно к нескольким столбцам, используя функцию across() из пакета dplyr (также содержится внутри пакета tidyverse). across() можно использовать с любой функцией dplyr, но она часто используется внутри select(), mutate(), filter() или summarise(). См. применение этой функции к summarise() на странице Описательные таблицы.
Укажите столбцы в аргументе .cols = и функцию(и) к применению в .fns =. Любые дополнительные аргументы, которые должны быть заданы для функции .fns могут быть включены после запятой, все еще внутри across().
Выбор столбцов в across()
Укажите столбцы в аргументе .cols =. Вы можете назвать их по отдельности или использовать функцию-помощника “tidyselect”. Уточните функцию в .fns =. Обратите внимание, что в использовании режима функции ниже, функция пишется без скобок ( ).
Здесь трансформация as.character() применяется к конкретным столбцам, названным внутри across().
linelist <- linelist %>%
mutate(across(.cols = c(temp, ht_cm, wt_kg), .fns = as.character))Доступны функции-помощники “tidyselect”, которые помогут вам в указании столбцов. Они детально представлены выше в разделе по Выбору и изменению порядка столбцов, и включают в себя: everything(), last_col(), where(), starts_with(), ends_with(), contains(), matches(), num_range() и any_of().
Здесь приведен пример того, как можно изменить все столбцы в текстовый класс:
#чтобы изменить все столбцы на текстовый класс
linelist <- linelist %>%
mutate(across(.cols = everything(), .fns = as.character))Конвертация в текстовый класс всех столбцов, где в названии есть последовательность символов “date” (обратите внимание на размещение запятых и скобок):
#чтобы изменить все столбцы на текстовый класс
linelist <- linelist %>%
mutate(across(.cols = contains("date"), .fns = as.character))Ниже представлен пример мутации столбцов, которые в настоящее время относятся к классу POSIXct (сырой класс датывремени, который показывает метки времени) - иными словами, где функция is.POSIXct() оценивается как TRUE (истина). Затем мы хотим применить функцию as.Date() к этим столбцам, чтобы конвертировать их в обычный класс Дата.
linelist <- linelist %>%
mutate(across(.cols = where(is.POSIXct), .fns = as.Date))- Обратите внимание, что внутри
across()мы также используем функциюwhere()какis.POSIXctдля оценки ИСТИНА или ЛОЖЬ.
- Обратите внимание, что
is.POSIXct()относится к пакету lubridate. Другие похожие “is” функции, такие какis.character(),is.numeric()иis.logical()относятся к базовому R
Функции across()
Вы можете прочитать документацию по ?across для получения дополнительной информации о том, как задавать функции для across(). Несколько общих моментов: существует несколько способов, чтобы уточнить, какую(ие) функцию(и) выполнить для столбца, и вы даже можете задать собственные функции:
- Вы можете задать только имя функции (например,
meanилиas.character)
- Вы можете задать функцию в стиле purrr (например,
~ mean(.x, na.rm = TRUE)) (см. [эту страницу][Итерации, циклы и списки])
- Вы можете задать несколько функций, представив список (например,
list(mean = mean, n_miss = ~ sum(is.na(.x))).- Если вы задаете несколько функций, несколько трансформированных столбцов будут выданы вам на столбец входных данных, с уникальными именами в формате
col_fn. Вы можете скорректировать, как будут названы новые столбцы с помощью аргумента.names =, используя синтаксис glue (см. страницу Текст и последовательности), где{.col}и{.fn}являются сокращениями для столбца входных данных и функции.
- Если вы задаете несколько функций, несколько трансформированных столбцов будут выданы вам на столбец входных данных, с уникальными именами в формате
Вот несколько онлайн ресурсов по использованию across(): мысли/обоснование создателя Хэдли Уикема
coalesce()
Эта функция dplyr находит первое неотсутствующее значение в каждом положении. Она “заполняет” отсутствующие значения первым доступным значением в указанном вами порядке.
Вот пример вне контекста датафрейма: Представим, что у вас есть два вектора, один содержит деревню, в которой выявлен пациент, а другой - деревню проживания пациента. Вы можете использовать coalesce для выбора первого неотсутствующего значения для каждого индекса:
village_detection <- c("a", "b", NA, NA)
village_residence <- c("a", "c", "a", "d")
village <- coalesce(village_detection, village_residence)
village # печать[1] "a" "b" "a" "d"Это работает таким же образом, если вы зададите столбцы датафрейма: для каждой строки функция присвоит новое значение столбца с помощью первого неотсутствующего значения в указанных вами столбцах (в указанном порядке).
linelist <- linelist %>%
mutate(village = coalesce(village_detection, village_residence))Это пример “построчной” операции. Чтобы посмотреть более сложные построчные расчеты, см. раздел ниже по построчным расчетам.
Кумулятивная математика
Если вы хотите, чтобы столбец отражал кумулятивную сумму/среднее значение/минимум/максимум и т.п., как оценено по строкам датафрейма на этот момент, используйте следующие функции:
cumsum() выдает кумулятивную сумму, как показано ниже:
sum(c(2,4,15,10)) # выдает только одно число[1] 31cumsum(c(2,4,15,10)) # выдает кумулятивную сумму на каждом шаге[1] 2 6 21 31Это можно использовать в датафрейме при создании нового столбца. Например, чтобы рассчитать кумулятивное количество случаев в день при вспышке, можно использовать следующий код:
cumulative_case_counts <- linelist %>% # начинаем с построчного списка случаев
count(date_onset) %>% # подсчитываем строки в день как столбец 'n'
mutate(cumulative_cases = cumsum(n)) # новый столбец кумулятивной суммы на каждой строкеНиже представлены первые 10 строк:
head(cumulative_case_counts, 10) date_onset n cumulative_cases
1 2012-04-15 1 1
2 2012-05-05 1 2
3 2012-05-08 1 3
4 2012-05-31 1 4
5 2012-06-02 1 5
6 2012-06-07 1 6
7 2012-06-14 1 7
8 2012-06-21 1 8
9 2012-06-24 1 9
10 2012-06-25 1 10См. также страницу Эпидемические кривые, чтобы узнать, как построить график кумулятивной заболеваемости с помощью эпидкривой.
См. также:cumsum(), cummean(), cummin(), cummax(), cumany(), cumall()
Использование базового R
Чтобы определить новый столбец (или переопределить столбец), используя базовый R, напишите имя датафрейма, соединенного с помощью $, для нового столбца (или столбца, который должен быть модифицирован). Используйте оператор присваивания <-, чтобы определить новое(ые) значение(я). Помните, что при использовании базового R вы должны уточнять имя датафрейма каждый раз перед именем столбца (например, dataframe$column). Здесь приведен пример создания столбца bmi с использованием базового R:
linelist$bmi = linelist$wt_kg / (linelist$ht_cm / 100) ^ 2)Добавление к цепочке канала
Ниже добавляется новый столбец к цепочке канала, а также конвертируются некоторые классы.
# ЦЕПОЧКА КАНАЛА ВЫЧИСТКИ (начинается с сырых данных, которые передаются по шагам вычистки)
##################################################################################
# начало цепочки канала вычистки
###########################
linelist <- linelist_raw %>%
# стандартизируем синтаксис имен столбцов
janitor::clean_names() %>%
# переименовываем столбцы вручную
# НОВОЕ имя # СТАРОЕ имя
rename(date_infection = infection_date,
date_hospitalisation = hosp_date,
date_outcome = date_of_outcome) %>%
# удаляем столбцы
select(-c(row_num, merged_header, x28)) %>%
# дедупликация
distinct() %>%
# ВЫШЕ ПОКАЗАНЫ РАНЕЕ ПРОДЕЛАННЫЕ И ПРЕДСТАВЛЕННЫЕ ШАГИ ВЫЧИСТКИ
###################################################
# добавляем новый столбец
mutate(bmi = wt_kg / (ht_cm/100)^2) %>%
# конвертируем класс столбцов
mutate(across(contains("date"), as.Date),
generation = as.numeric(generation),
age = as.numeric(age)) 8.8 Перекодирование значений
Здесь представлено несколько сценариев, при которых вам нужно перекодировать (изменить) значения:
- чтобы редактировать одно конкретное значение (например, одна дата с неправильным годом или форматом)
- чтобы сделать единообразными значения, которые имеют разное правописание
- чтобы создать новый столбец категориальных значений
- чтобы создать новый столбец числовых категорий (например, возрастные категории)
Конкретные значения
Чтобы изменить значения вручную, вы можете использовать функцию recode() внутри функции mutate().
Представьте,что в дате стоит какая-то бессмысленная дата (например, “2014-14-15”): вы можете исправить дату вручную в сырых исходных данных, либо вы можете записать изменение в рамках цепочки канала вычистки с помощью mutate() и recode(). Последний вариант будет более прозрачным и воспроизводимям, если кто-то захочет понять или повторить ваш анализ.
# исправление неправильных значений # старое значение # новое значение
linelist <- linelist %>%
mutate(date_onset = recode(date_onset, "2014-14-15" = "2014-04-15"))Строку mutate() выше можно прочитать как: “изменить столбец date_onset, чтобы он был равен столбцу date_onset с перекодированием, так чтобы СТАРОЕ ЗНАЧЕНИЕ было изменено на НОВОЕ ЗНАЧЕНИЕ”. Обратите внимание, что эта последовательность (СТАРОЕ = НОВОЕ) в recode() противоположна большинству последовательностей в R (новое = старое). Сообщество разработчиков R уже работает над этим изменением.
Вот еще один пример перекодирования нескольких значений в одном столбце.
В linelist необходимо вычистить значения в столбце “hospital”. Есть несколько вариантов написания и много отсутствующих значений.
table(linelist$hospital, useNA = "always") # печать таблицы всех уникальных значений, включая отсутствующие
Central Hopital Central Hospital
11 457
Hospital A Hospital B
290 289
Military Hopital Military Hospital
32 798
Mitylira Hopital Mitylira Hospital
1 79
Other Port Hopital
907 48
Port Hospital St. Mark's Maternity Hospital (SMMH)
1756 417
St. Marks Maternity Hopital (SMMH) <NA>
11 1512 Команда recode() ниже переопределяет столбец “hospital” (больницы) как текущий столбец “hospital”, но с указанными изменениями по перекодированию. Не забывайте запятые после каждого!
linelist <- linelist %>%
mutate(hospital = recode(hospital,
# для справки: СТАРОЕ = НОВОЕ
"Mitylira Hopital" = "Military Hospital",
"Mitylira Hospital" = "Military Hospital",
"Military Hopital" = "Military Hospital",
"Port Hopital" = "Port Hospital",
"Central Hopital" = "Central Hospital",
"other" = "Other",
"St. Marks Maternity Hopital (SMMH)" = "St. Mark's Maternity Hospital (SMMH)"
))Теперь мы видим, что написание в столбце hospital было исправлено и унифицировано:
table(linelist$hospital, useNA = "always")
Central Hospital Hospital A
468 290
Hospital B Military Hospital
289 910
Other Port Hospital
907 1804
St. Mark's Maternity Hospital (SMMH) <NA>
428 1512 СОВЕТ: Количество пробелов до и после знака равно не имеет значения. Упростите чтение вашего кода, выровнив знаки = для всех или большинства строк. Кроме того, рассмотрите возможность добавления комментария со знаком решетка, чтобы пояснить будущим читателям, какая сторона является СТАРОЙ, а какая - НОВОЙ.
СОВЕТ: Иногда в наборе данных может иметься пустое текстовое значение (не стандартное значение в R для отсутствующих значений - NA). Вы можете ссылаться на это значения с помощью двух кавычек без пробела между ними (““).
По логике
Ниже мы показываем, как перекодировать значения в столбце, используя логику и условия:
- Используя
replace(),ifelse()иif_else()для простой логики - Используя
case_when()для более сложной логики
Простая логика
replace()
Чтобы перекодировать с простыми логическими критериями, вы можете использовать replace() внутри mutate(). replace() - это функция из базового R. Используйте логическое условие, чтобы уточнить, какие строки менять. Общий синтаксис выглядит следующим образом:
mutate(col_to_change = replace(col_to_change, критерии для строк, новое значение)).
Одна из частых ситуаций для использования replace() - это изменение только одного значения в одной строке, используя уникальный идентификатор строки. Ниже пол меняется на “Female” в строке, где столбец case_id равен “2195”.
# Пример: изменение пола в одном конкретном наблюдении на "Female"
linelist <- linelist %>%
mutate(gender = replace(gender, case_id == "2195", "Female"))Эквивалент команды с использованием синтаксиса базового R и индексных квадратных скобок [ ] приведен ниже. Его следует читать как: “Изменить значение столбца gender в датаврейме linelist (для строк, где столбец case_id в linelist имеет значение ‘2195’) на ‘Female’”.
linelist$gender[linelist$case_id == "2195"] <- "Female"
ifelse() и if_else()
Еще один инструмент простой логики - это ifelse() и функция-партнер if_else(). Однако в большинстве случаев перекодирования понятнее будет использовать case_when() (описан ниже). Эти команды “if else” (если не) являются упрощенными версиями программного утверждения if (если) и else (кроме). Общий синтаксис выглядит следующим образом:ifelse(условие, значение, которое выдается, если условие оценено как ИСТИНА, значение, которое выдается, если условие оценено как ЛОЖЬ)
Ниже определяется столбецsource_known. Его значение в соответствующей строке установлено на “known” (известен), если в столбце source не отсутствует значение строки. Если в столбце source отсутствует значение, тогда значение source_known устанавливается как “unknown” (неизвестен).
linelist <- linelist %>%
mutate(source_known = ifelse(!is.na(source), "known", "unknown"))if_else() - это специальная версия из dplyr, которая работает с датами. Обратите внимание, что если ‘true’ (истиным) значением является дата, то ‘false’ (ложное) значение должно также квалифицироваться как дата, поэтому вместо просто NA используется специальное значение NA_real_ .
# создаем столбец дата смерти, в котором значение будет NA, если пациент не умер.
linelist <- linelist %>%
mutate(date_death = if_else(outcome == "Death", date_outcome, NA_real_))Избегайте использования чрезмерно большого количества команд ifelse одновременно… вместо этого используйте case_when()! case_when() гораздо легче читать, и вы допустите меньше ошибок.
Вне контекста датафрейма, если вы хотите, чтобы объект, используемые в вашем коде, поменял значение, рассмотрите использование switch() из базового R.
Сложная логика
Используйте case_when() из dplyr, если вы перекодируете в большое количество новых групп, либо если вам нужно использовать сложные логические утверждения для перекодирования значений. Эта функция оценивает каждую строку в датафрейме, оценивает, соответствуют ли строки указанным критериям и присваивает правильное новое значение.
Команды case_when() состоят из утверждений, которые имеют правую сторону и левую сторону, разделенную “тильдой” ~. Логические критерии указаны слева, а согласующиеся значения - справа в каждом утверждении. Утверждения разделяются запятыми.
Например, здесь мы используем столбцы age и age_unit, чтобы создать столбец age_years:
linelist <- linelist %>%
mutate(age_years = case_when(
age_unit == "years" ~ age, # если единица возраста годы
age_unit == "months" ~ age/12, # если единица возраста месяцы, разделить возраст на 12
is.na(age_unit) ~ age)) # если единица возраста отсутствует, предположить, что это годы
# в любых других обстоятельствах - присвоить NA (отсутствует)По мере того как оценивается каждая строка данных, применяются/оцениваются критерии в порядке написания утверждений case_when() - сверху вниз. Если верхний критерий оценивается как TRUE (истина) для определенной строки, присваивается значение из правой стороны, а остальные критерии даже не тестируются для этой строки данных. Таким образом, лучше сначала записать наиболее специфичный критерий, а последним записать наиболее общий критерий. Строке данных, которая не соответствует ни одному критерию, будет присвоено NA.
Иногда вы можете записать финальное утверждение, которое присваивает значение для всех прочих сценариев, не описанных в одной из предыдущих строк. Чтобы это сделать, разместите TRUE в левой стороне, что охватит любую строку, которая не соответствовала ни одному из предыдущих критериев. Правой стороне этого утверждения можно присвоить значение, например “проверь меня!” или отсутствует.
Ниже приведен еще один пример использования case_when() для создания нового столбца с классификацией пациента в соответствии с определением случая подтвержденного и подозрительного случая:
linelist <- linelist %>%
mutate(case_status = case_when(
# если у пациента есть лабораторный тест и он положительный,
# тогда он отмечается как подтвержденный случай
ct_blood < 20 ~ "Confirmed",
# при условии, что у пациента нет положительного лабораторного теста,
# если у пациента есть "источник" (эпидемиологическая связь) И есть жар,
# тогда он маркируется как подозрительный случай
!is.na(source) & fever == "yes" ~ "Suspect",
# любой другой пациент, не рассмотренный выше
# отмечается для дальнейшего наблюдения
TRUE ~ "To investigate"))ВНИМАНИЕ: Значения справа должны быть все в одном классе - либо числовые, текстовые, дата, логические и т.п. Чтобы присвоить отсутствующее значение (NA), вам может потребоваться использовать особые варианты NA, такие как NA_character_, NA_real_ (для числовых или POSIX), и as.Date(NA). Более детально см. страницу Работа с датами.
Отсутствующие значения
Ниже приведены специальные функции для работы с отсутствующими значениями в контексте вычистки данных.
См. страницу Отсутствующие данные для получения более детальных советов о том, как определять и работать с отсутствующими значениями. Например, функция is.na(), которая логически тестирует на предмет отсутствия.
replace_na()
Чтобы изменить отсутствующие значения (NA) на конкретное значение, например “Missing” (“Отсутствует”), используйте функцию replace_na() из dplyr внутри mutate(). Обратите внимание, что она используется таким же образом, как и recode выше - имя переменной необходимо повторить в replace_na().
linelist <- linelist %>%
mutate(hospital = replace_na(hospital, "Missing"))fct_explicit_na()
Это функция из пакета forcats. Пакет forcats работает со столбцами с классом Фактор. Факторы - то, как R работает с порядковыми значениями, такими как c("First" (первый), "Second" (второй), "Third"(третий)) или задает порядок, в котором появляются значения (например, больницы) в таблицах и графиках. См. страницу Факторы.
Если ваши данные относятся к классу Фактор и вы попытаетесь конвертировать NA в “Missing” (“отсутствует”) с помощью replace_na(), вы увидите следующее сообщение об ошибке: invalid factor level, NA generated (недействительный уровень фактора, сгенерирован NA). Вы попытались добавить “Missing” как значение, а оно не было определено как возможный уровень фактора и было отклонено.
Самый простой способ решить эту проблему - использовать функцию из пакета forcats fct_explicit_na(), которая конвертирует столбец в класс фактор и конвертирует значения NA на текстовые “(Missing - отсутствует)”.
linelist %>%
mutate(hospital = fct_explicit_na(hospital))Более медленная альтернатива - добавить уровень фактора, используя fct_expand() и затем конвертируя отсутствующие значения.
na_if()
Чтобы конвертировать конкретное значение в NA, используйте функцию na_if() из dplyr. Команда ниже проводит противоположную replace_na() операцию. В примере ниже любые значения “Missing” в столбце hospital конвертируются в NA.
linelist <- linelist %>%
mutate(hospital = na_if(hospital, "Missing"))Примечание: na_if() не может использоваться для логических критериев (например, “all values > 99” (“все значения > 99”)) - используйте replace() или case_when() для этих целей:
# Конвертация температуры выше 40 в NA
linelist <- linelist %>%
mutate(temp = replace(temp, temp > 40, NA))
# Конвертация даты заболевания раньше, чем 1 января 2000 в отсутствующую
linelist <- linelist %>%
mutate(date_onset = replace(date_onset, date_onset > as.Date("2000-01-01"), NA))Словарь вычистки
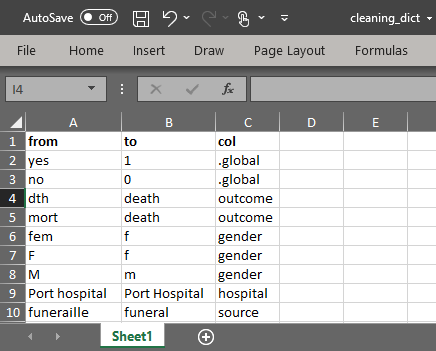
Используйте пакет R matchmaker и его функцию match_df() для вычистки датафрейма с помощью словаря вычистки.
- Создайте словарь вычистки с 3 столбцами:
- столбец “из” (неправильное значение)
- столбец “в” (правильное значение)
- столбец, указывающий столбец, к которому применяются изменения (или “.global”, чтобы применить ко всем столбцам)
- столбец “из” (неправильное значение)
Примечание: записи в словаре для конкретного столбца будут превалировать над записями в словаре .global.
- Импортируйте файл со словарем в R. Этот пример можно скачать, следуя инструкциям на странице Скачивание руководства и данных.
cleaning_dict <- import("cleaning_dict.csv")- Поставьте сырой постройчный список в функцию
match_df(), уточнивdictionary =датафрейм словаря вычистки. Аргументfrom =должен указывать на имя столбца словаря, в котором содержатся “старые значения”, аргументby =должен быть столбцом словаря, в котором содержатся соответствующие “новые” значения, а третий столбец указывает столбец, в котором необходимо проводить изменение. Используйте.globalв столбцеby =, чтобы применить изменение ко всем столбцам. Четвертый столбец словаряorderможет быть использован для уточнения порядка факторов новых значений.
Дополнительные детали можно прочитать в документации по пакету, выполнив ?match_df. Обратите внимание, что эта функция может потребовать длительного времени выполнения для большого набора данных.
linelist <- linelist %>% # укажите или поставьте в канал ваш набор данных
matchmaker::match_df(
dictionary = cleaning_dict, # имя вашего словаря
from = "from", # столбец со значениям, которые нужно заменить (по умолчанию столбец 1)
to = "to", # столбец с итоговыми значениями (по умолчанию столбец 2)
by = "col" # столбец с названиями столбцов (по умолчанию столбец 3)
)Теперь пролистайте вправо, чтобы увидеть, как изменились значения - особенно gender (со строчных на заглавные), а все столбцы с симптомами были трансформированы из yes/no в 1/0.
Обратите внимание, что ваши имена столбцов в словаре вычистки должны соответствовать именам столбцов в вашем скрипте вычистки на данный момент. См. онлайн ссылку для пакета по построчным спискам для получения дополнительной информации.
Добавление в цепочку канала
Ниже в цепочку канала добавляются некоторые новые столбцы и проводятся преобразования столбцов.
# ЦЕПОЧКА КАНАЛА ВЫЧИСТКИ (начинается с сырых данных, которые передаются по шагам вычистки)
##################################################################################
# начало цепочки канала вычистки
###########################
linelist <- linelist_raw %>%
# стандартизируем синтаксис имен столбцов
janitor::clean_names() %>%
# переименовываем столбцы вручную
# НОВОЕ имя # СТАРОЕ имя
rename(date_infection = infection_date,
date_hospitalisation = hosp_date,
date_outcome = date_of_outcome) %>%
# удаляем столбцы
select(-c(row_num, merged_header, x28)) %>%
# дедупликация
distinct() %>%
# добавляем столбец
mutate(bmi = wt_kg / (ht_cm/100)^2) %>%
# конвертируем класс столбцов
mutate(across(contains("date"), as.Date),
generation = as.numeric(generation),
age = as.numeric(age)) %>%
# добавляем столбец: задержка госпитализации
mutate(days_onset_hosp = as.numeric(date_hospitalisation - date_onset)) %>%
# ВЫШЕ ПОКАЗАНЫ РАНЕЕ ПРОДЕЛАННЫЕ И ПРЕДСТАВЛЕННЫЕ ШАГИ ВЫЧИСТКИ
###################################################
# чистые значения столбца hospital
mutate(hospital = recode(hospital,
# OLD = NEW
"Mitylira Hopital" = "Military Hospital",
"Mitylira Hospital" = "Military Hospital",
"Military Hopital" = "Military Hospital",
"Port Hopital" = "Port Hospital",
"Central Hopital" = "Central Hospital",
"other" = "Other",
"St. Marks Maternity Hopital (SMMH)" = "St. Mark's Maternity Hospital (SMMH)"
)) %>%
mutate(hospital = replace_na(hospital, "Missing")) %>%
# создаем столбец age_years (из age и age_unit)
mutate(age_years = case_when(
age_unit == "years" ~ age,
age_unit == "months" ~ age/12,
is.na(age_unit) ~ age,
TRUE ~ NA_real_))8.9 Числовые категории
Здесь мы опишем некоторые особые подходы к созданию категорий из числовых столбцов. Частые примеры включают возрастные категории, группы лабораторных результатов и т.п. Здесь мы обсудим:
-
age_categories(), из пакета epikit
-
cut(), из базового R
-
case_when()
- квантильные разбивки с помощью
quantile()иntile()
Обзор распределения
Для данного примера мы создадим столбец age_cat, используя столбец age_years.
# проверьте класс переменной возраста в linelist
class(linelist$age_years)[1] "numeric"Сначала рассмотрим распределение ваших данных, чтобы определить соответствующие пороговые точки. См. страницу основы ggplot.
summary(linelist$age_years, na.rm=T) Min. 1st Qu. Median Mean 3rd Qu. Max. NA's
0.00 6.00 13.00 16.04 23.00 84.00 107 ВНИМАНИЕ: Иногда числовые переменные импортируются как “текстовый” класс. Это происходит, если в каких-то значениях имеются нечисловые символы, например, запись возраста как “2 месяца” или (в зависимости от настроек вашего региона в R), если вместо точки используется запятая в десятичных числах (например, “4,5” как четыре с половиной года)..
age_categories()
С пакетом epikit вы можете использовать функцию age_categories(), чтобы легким образом разделить на категории и промаркировать числовые столбцы (примечание: эту функцию можно применить и к невозрастным числовым переменным). В качестве бонуса, получившийся столбец автоматически будет являться упорядоченным фактором.
Вот несколько требуемых входных данных:
- Числовой вектор (столбец)
- Аргумент
breakers =- предоставляет числовой вектор точек разрыва на новые группы
Для начала самый простой пример:
# Простой пример
################
pacman::p_load(epikit) # загружаем пакеты
linelist <- linelist %>%
mutate(
age_cat = age_categories( # создаем новый столбец
age_years, # числовой столбец, из которого будут сделаны группы
breakers = c(0, 5, 10, 15, 20, # точки разрыва
30, 40, 50, 60, 70)))
# показать таблицу
table(linelist$age_cat, useNA = "always")
0-4 5-9 10-14 15-19 20-29 30-39 40-49 50-59 60-69 70+ <NA>
1227 1223 1048 827 1216 597 251 78 27 7 107 Значения точек разрыва, которые вы указываете, по умолчанию будут нижними порогами - т.е. они включаются в “более высокую” группу / группы “открыты” с нижней/левой стороны. Как показано ниже, вы можете добавить 1 к каждому значению разрыва, чтобы создать группы, которые будут открыты сверху/справа.
# Включаем верхние границы для тех же категорий
############################################
linelist <- linelist %>%
mutate(
age_cat = age_categories(
age_years,
breakers = c(0, 6, 11, 16, 21, 31, 41, 51, 61, 71)))
# показать таблицу
table(linelist$age_cat, useNA = "always")
0-5 6-10 11-15 16-20 21-30 31-40 41-50 51-60 61-70 71+ <NA>
1469 1195 1040 770 1149 547 231 70 24 6 107 Вы можете скорректировать то, как будут отображаться подписи с помощью separator =. По умолчанию ставится “-”
Вы можете скорректировать обращение с верхними цифрами с помощью аргумента ceiling =. Чтобы задать верхний разрыв установите ceiling = TRUE. В таком случае будет предоставлено самое высокое значение разрыва, которое будет “потолком” и не будет создаваться категория “XX+”. Любые значения выше наивысшего разрыва (или upper =, если он определен) будут категоризироваьтся как NA. Ниже представлен пример, где ceiling = TRUE, поэтому нет категории XX+ и значениям выше 70 (самое высокое значение разрыва) присваивается значение NA.
# устанавливаем потолок на TRUE (ИСТИНА)
##########################
linelist <- linelist %>%
mutate(
age_cat = age_categories(
age_years,
breakers = c(0, 5, 10, 15, 20, 30, 40, 50, 60, 70),
ceiling = TRUE)) # 70 - это потолок, все, что выше, становится NA
# показать таблицу
table(linelist$age_cat, useNA = "always")
0-4 5-9 10-14 15-19 20-29 30-39 40-49 50-59 60-70 <NA>
1227 1223 1048 827 1216 597 251 78 28 113 Альтернативно вместо breakers = вы можете предоставить все значения для lower =, upper =, и by =:
-
lower =самое нижнее число, которое вы хотите рассмотреть - по умолчанию 0
-
upper =самое верхнее число, которое вы хотите рассмотреть
-
by =количество лет между группами
linelist <- linelist %>%
mutate(
age_cat = age_categories(
age_years,
lower = 0,
upper = 100,
by = 10))
# показать таблицу
table(linelist$age_cat, useNA = "always")
0-9 10-19 20-29 30-39 40-49 50-59 60-69 70-79 80-89 90-99 100+ <NA>
2450 1875 1216 597 251 78 27 6 1 0 0 107 См. справку по функции для получения дополнительной информации (введите ?age_categories в консоли R).
cut()
cut() - это альтернатива для age_categories() из базового R, но, я думаю, вы увидите, почему была разработана функция age_categories() для упрощения процесса. Некоторые заметные различия от age_categories() включают:
- Вам не нужно устанавливать/загружать другой пакет
- Вы можете уточнить, будут ли группы открыты/закрыты справа/слева
- Вы можете сами задать точные подписи
- Если вы хотите, чтобы 0 был включен в самую нижнюю группу, вы должны это указать
Базовый синтаксис внутри cut() - сначала задать числовой столбец, который нужно вырезать (age_years), и затем аргумент с разрывами, который является числовым вектором c() разрывов. При использовании cut(), получившийся в результате столбец будет упорядоченным фактором.
По умолчанию разделение на категории происходит таким образом, что правая/верхняя сторона является “открытой” и инклюзивной (а левая/нижняя сторона является “закрытой” или исключающей). Это поведение, противоположное функции age_categories(). По умолчанию подписи используют запись “(A, B]”, что означает, что A не включается, а B включается. Вы можете задать обратное поведение, указав аргумент right = TRUE.
Таким образом, по умолчанию, значения “0” исключаются из самой нижней группы и категоризируются как NA! Значения “0” могут включать младенцев, закодированных как возраст 0, так что будьте осторожны! Чтобы это изменить, добавьте аргумент include.lowest = TRUE, чтобы любые значения с “0” включались в самую нижнюю группу. Автоматически сгенерированная подпись для самой нижней категории тогда будет “[A],B]”. Обратите внимание, что если вы включите аргумент include.lowest = TRUE и right = TRUE, экстремальное включение теперь будет применяться к наивысшему значению разрыва и категории, а не к самой нижней.
Вы можете задать вектор пользовательских подписей, используя аргумент labels =. Так как они записываются вручную, убедитесь, что они записаны правильно” Проверьте свою работу, используя кросс-табуляцию, как описано ниже.
В качестве примера применим cut() к age_years, чтобы создать новую переменную age_cat:
# Создаем новую переменную, разделив числовую переменную возраста
# нижний разрыв исключается, а верхний разрыв включен в каждую категорию
linelist <- linelist %>%
mutate(
age_cat = cut(
age_years,
breaks = c(0, 5, 10, 15, 20,
30, 50, 70, 100),
include.lowest = TRUE # включаем 0 в самую нижнюю группу
))
# подсчитываем количество наблюдений в каждой группе
table(linelist$age_cat, useNA = "always")
[0,5] (5,10] (10,15] (15,20] (20,30] (30,50] (50,70] (70,100]
1469 1195 1040 770 1149 778 94 6
<NA>
107 Проверьте свою работу!!! Проверьте, что каждое значение возраста было распределено в правильную категорию, проведя кросс-табуляцию числовых и категориальных столбцов. Рассмотрите присвоение пограничных значений (например, 15, если соседние категории включают 10-15 и 16-20).
# Кросс-табуляция числовыъ и категориальных столбцов.
table("Numeric Values" = linelist$age_years, # имена, указанные в таблице для ясности.
"Categories" = linelist$age_cat,
useNA = "always") # не забудьте проверить значения NA Categories
Numeric Values [0,5] (5,10] (10,15] (15,20] (20,30] (30,50] (50,70]
0 136 0 0 0 0 0 0
0.0833333333333333 1 0 0 0 0 0 0
0.25 2 0 0 0 0 0 0
0.333333333333333 6 0 0 0 0 0 0
0.416666666666667 1 0 0 0 0 0 0
0.5 6 0 0 0 0 0 0
0.583333333333333 3 0 0 0 0 0 0
0.666666666666667 3 0 0 0 0 0 0
0.75 3 0 0 0 0 0 0
0.833333333333333 1 0 0 0 0 0 0
0.916666666666667 1 0 0 0 0 0 0
1 275 0 0 0 0 0 0
1.5 2 0 0 0 0 0 0
2 308 0 0 0 0 0 0
3 246 0 0 0 0 0 0
4 233 0 0 0 0 0 0
5 242 0 0 0 0 0 0
6 0 241 0 0 0 0 0
7 0 256 0 0 0 0 0
8 0 239 0 0 0 0 0
9 0 245 0 0 0 0 0
10 0 214 0 0 0 0 0
11 0 0 220 0 0 0 0
12 0 0 224 0 0 0 0
13 0 0 191 0 0 0 0
14 0 0 199 0 0 0 0
15 0 0 206 0 0 0 0
16 0 0 0 186 0 0 0
17 0 0 0 164 0 0 0
18 0 0 0 141 0 0 0
19 0 0 0 130 0 0 0
20 0 0 0 149 0 0 0
21 0 0 0 0 158 0 0
22 0 0 0 0 149 0 0
23 0 0 0 0 125 0 0
24 0 0 0 0 144 0 0
25 0 0 0 0 107 0 0
26 0 0 0 0 100 0 0
27 0 0 0 0 117 0 0
28 0 0 0 0 85 0 0
29 0 0 0 0 82 0 0
30 0 0 0 0 82 0 0
31 0 0 0 0 0 68 0
32 0 0 0 0 0 84 0
33 0 0 0 0 0 78 0
34 0 0 0 0 0 58 0
35 0 0 0 0 0 58 0
36 0 0 0 0 0 33 0
37 0 0 0 0 0 46 0
38 0 0 0 0 0 45 0
39 0 0 0 0 0 45 0
40 0 0 0 0 0 32 0
41 0 0 0 0 0 34 0
42 0 0 0 0 0 26 0
43 0 0 0 0 0 31 0
44 0 0 0 0 0 24 0
45 0 0 0 0 0 27 0
46 0 0 0 0 0 25 0
47 0 0 0 0 0 16 0
48 0 0 0 0 0 21 0
49 0 0 0 0 0 15 0
50 0 0 0 0 0 12 0
51 0 0 0 0 0 0 13
52 0 0 0 0 0 0 7
53 0 0 0 0 0 0 4
54 0 0 0 0 0 0 6
55 0 0 0 0 0 0 9
56 0 0 0 0 0 0 7
57 0 0 0 0 0 0 9
58 0 0 0 0 0 0 6
59 0 0 0 0 0 0 5
60 0 0 0 0 0 0 4
61 0 0 0 0 0 0 2
62 0 0 0 0 0 0 1
63 0 0 0 0 0 0 5
64 0 0 0 0 0 0 1
65 0 0 0 0 0 0 5
66 0 0 0 0 0 0 3
67 0 0 0 0 0 0 2
68 0 0 0 0 0 0 1
69 0 0 0 0 0 0 3
70 0 0 0 0 0 0 1
72 0 0 0 0 0 0 0
73 0 0 0 0 0 0 0
76 0 0 0 0 0 0 0
84 0 0 0 0 0 0 0
<NA> 0 0 0 0 0 0 0
Categories
Numeric Values (70,100] <NA>
0 0 0
0.0833333333333333 0 0
0.25 0 0
0.333333333333333 0 0
0.416666666666667 0 0
0.5 0 0
0.583333333333333 0 0
0.666666666666667 0 0
0.75 0 0
0.833333333333333 0 0
0.916666666666667 0 0
1 0 0
1.5 0 0
2 0 0
3 0 0
4 0 0
5 0 0
6 0 0
7 0 0
8 0 0
9 0 0
10 0 0
11 0 0
12 0 0
13 0 0
14 0 0
15 0 0
16 0 0
17 0 0
18 0 0
19 0 0
20 0 0
21 0 0
22 0 0
23 0 0
24 0 0
25 0 0
26 0 0
27 0 0
28 0 0
29 0 0
30 0 0
31 0 0
32 0 0
33 0 0
34 0 0
35 0 0
36 0 0
37 0 0
38 0 0
39 0 0
40 0 0
41 0 0
42 0 0
43 0 0
44 0 0
45 0 0
46 0 0
47 0 0
48 0 0
49 0 0
50 0 0
51 0 0
52 0 0
53 0 0
54 0 0
55 0 0
56 0 0
57 0 0
58 0 0
59 0 0
60 0 0
61 0 0
62 0 0
63 0 0
64 0 0
65 0 0
66 0 0
67 0 0
68 0 0
69 0 0
70 0 0
72 1 0
73 3 0
76 1 0
84 1 0
<NA> 0 107Смена подписи значений NA
Вам может потребоваться присвоить значениям NA подпись, например, “Missing” (отсутствует). Поскольку новый столбец относится к классу Фактор (ограниченные значения), вы не можете просто использовать replace_na(), поскольку это значение будет отклонено. Вместо этого используйте fct_explicit_na() из пакета forcats, как объясняется на странице Факторы.
linelist <- linelist %>%
# cut() создает age_cat, автоматически в классе Фактор
mutate(age_cat = cut(
age_years,
breaks = c(0, 5, 10, 15, 20, 30, 50, 70, 100),
right = FALSE,
include.lowest = TRUE,
labels = c("0-4", "5-9", "10-14", "15-19", "20-29", "30-49", "50-69", "70-100")),
# делает отсутствующие значения явными
age_cat = fct_explicit_na(
age_cat,
na_level = "Missing age") # вы можете задать подпись
) Warning: There was 1 warning in `mutate()`.
ℹ In argument: `age_cat = fct_explicit_na(age_cat, na_level = "Missing age")`.
Caused by warning:
! `fct_explicit_na()` was deprecated in forcats 1.0.0.
ℹ Please use `fct_na_value_to_level()` instead.# таблица для просмотра подсчета
table(linelist$age_cat, useNA = "always")
0-4 5-9 10-14 15-19 20-29 30-49
1227 1223 1048 827 1216 848
50-69 70-100 Missing age <NA>
105 7 107 0 Быстрое создание разрывов и подписей
Для быстрого создания разрывов и векторов подписей можно использовать код ниже. См. страницу Основы R для получения информации о seq() и rep().
# Создание разрывов от 0 до 90 с шагом 5
age_seq = seq(from = 0, to = 90, by = 5)
age_seq
# Создание подписей для созданных выше категорий, предполагая настройки cut() по умолчанию
age_labels = paste0(age_seq + 1, "-", age_seq + 5)
age_labels
# Проверьте, что оба вектора имеют одинаковую длину
length(age_seq) == length(age_labels)Более подробно о cut() можно прочитать на странице Справки, введя ?cut в консоли R.
Разрывы по квантилям
Как правило, “квантили” или “процентили” означают значение, ниже которого находится определенная доля значений. Например, 95-й процентиль возрастов в linelist будет возраст, ниже которого находится 95% всех возрастов.
Однако в простой речи “квартили” и “децили” могут также означать группы данных, равномерно разделенных на 4 или 10 групп (обратите внимание, будет на один разрыв больше, чем групп).
Чтобы получить разрывы по квантилям, можете использовать quantile() из пакета stats из базового R. Вы задаете числовой вектор (например, столбец в наборе данных) и вектор значений числовой вероятности, варьирующихся от 0 до 1.0. Разрывы будут выданы как числовой вектор. Детали статистической методологии можно изучить, введя ?quantile.
- Если у вашего входного числового вектора есть какие-то отсутствующие значения, лучше установить
na.rm = TRUE
- Задайте
names = FALSE, чтобы получить неименованный числовой вектор
quantile(linelist$age_years, # укажите числовой вектор, с которым надо работать
probs = c(0, .25, .50, .75, .90, .95), # укажите, какие процентили вам нужны
na.rm = TRUE) # игнорировать отсутствующие значения 0% 25% 50% 75% 90% 95%
0 6 13 23 33 41 Вы можете использовать результаты quantile() в качестве разрывов в age_categories() или cut(). Ниже мы создаем новый столбец deciles, используя cut(), где разрывы задаются с использованием quantiles() для age_years. Ниже мы отображаем результаты, используя tabyl() из пакета janitor, чтобы вы могли увидеть проценты (см. страницу Описательные таблицы). Обратите внимание, что они не составляют точно 10% в каждой группе.
linelist %>% # начинаем с построчного списка
mutate(deciles = cut(age_years, # создаем новый столбец decile через cut() по столбцу age_years
breaks = quantile( # определяем разрывы, используя quantile()
age_years, # операция для age_years
probs = seq(0, 1, by = 0.1), # от 0.0 до 1.0 с шагом 0.1
na.rm = TRUE), # игнорировать отсутствующие значения
include.lowest = TRUE)) %>% # для cut() включите возраст 0
janitor::tabyl(deciles) # передайте по каналу для создания таблицы для отображения deciles n percent valid_percent
[0,2] 748 0.11319613 0.11505922
(2,5] 721 0.10911017 0.11090601
(5,7] 497 0.07521186 0.07644978
(7,10] 698 0.10562954 0.10736810
(10,13] 635 0.09609564 0.09767728
(13,17] 755 0.11425545 0.11613598
(17,21] 578 0.08746973 0.08890940
(21,26] 625 0.09458232 0.09613906
(26,33] 596 0.09019370 0.09167820
(33,84] 648 0.09806295 0.09967697
<NA> 107 0.01619249 NAГруппы равного размера
Еще один инструмент для создания числовых групп - функция ntile() из пакета dplyr, которая попробует разбить ваши данные на n групп равного размера - но помните, что в отличие от quantile(), одно и то же значение может появиться в нескольких группах. Задайте числовой вектор и затем количество групп. Значения в новом созданном столбце - просто “номера” групп (например, от 1 до 10), а не диапазон самих значений, как при использовании cut().
# создаем группы с помощью ntile()
ntile_data <- linelist %>%
mutate(even_groups = ntile(age_years, 10))
# создаем таблицу с подсчетом количества и пропорциями по группам
ntile_table <- ntile_data %>%
janitor::tabyl(even_groups)
# присваиваем мин/макс значения, чтобы продемонстрировать диапазоны
ntile_ranges <- ntile_data %>%
group_by(even_groups) %>%
summarise(
min = min(age_years, na.rm=T),
max = max(age_years, na.rm=T)
)Warning: There were 2 warnings in `summarise()`.
The first warning was:
ℹ In argument: `min = min(age_years, na.rm = T)`.
ℹ In group 11: `even_groups = NA`.
Caused by warning in `min()`:
! no non-missing arguments to min; returning Inf
ℹ Run `dplyr::last_dplyr_warnings()` to see the 1 remaining warning.# объединяем и печатаем - обратите внимание, что значения присутствуют в нескольких группах
left_join(ntile_table, ntile_ranges, by = "even_groups") even_groups n percent valid_percent min max
1 651 0.09851695 0.10013844 0 2
2 650 0.09836562 0.09998462 2 5
3 650 0.09836562 0.09998462 5 7
4 650 0.09836562 0.09998462 7 10
5 650 0.09836562 0.09998462 10 13
6 650 0.09836562 0.09998462 13 17
7 650 0.09836562 0.09998462 17 21
8 650 0.09836562 0.09998462 21 26
9 650 0.09836562 0.09998462 26 33
10 650 0.09836562 0.09998462 33 84
NA 107 0.01619249 NA Inf -Infcase_when()
Можно использовать функцию case_when() из dplyr для создания категорий из числового столбца, но легче использовать age_categories() из epikit или cut(), поскольку они автоматически создадут упорядоченный фактор.
При использовании case_when(), пожалуйста, проверьте правильность использования, как описывалось выше в разделе по перекодированию данной страницы. Также помните, что значения в правой стороне должны быть одного класса. Так что, если вы хотите видеть NA справа, вам следует написать “Missing” или использовать специальное значение NA в виде NA_character_.
Добавление в цепочку канала
Ниже код для создания двух категориальных столбцов возраста добавляется в цепочку канала вычистки:
# ЦЕПОЧКА КАНАЛА ВЫЧИСТКИ (начинается с сырых данных, которые передаются по шагам вычистки)
##################################################################################
# начало цепочки канала вычистки
###########################
linelist <- linelist_raw %>%
# стандартизируем синтаксис имен столбцов
janitor::clean_names() %>%
# переименовываем столбцы вручную
# НОВОЕ имя # СТАРОЕ имя
rename(date_infection = infection_date,
date_hospitalisation = hosp_date,
date_outcome = date_of_outcome) %>%
# удаляем столбцы
select(-c(row_num, merged_header, x28)) %>%
# дедупликация
distinct() %>%
# добавляем столбец
mutate(bmi = wt_kg / (ht_cm/100)^2) %>%
# конвертируем класс столбцов
mutate(across(contains("date"), as.Date),
generation = as.numeric(generation),
age = as.numeric(age)) %>%
# добавляем столбец: задержка госпитализации
mutate(days_onset_hosp = as.numeric(date_hospitalisation - date_onset)) %>%
# чистые значения столбца hospital
mutate(hospital = recode(hospital,
# СТАРОЕ = НОВОЕ
"Mitylira Hopital" = "Military Hospital",
"Mitylira Hospital" = "Military Hospital",
"Military Hopital" = "Military Hospital",
"Port Hopital" = "Port Hospital",
"Central Hopital" = "Central Hospital",
"other" = "Other",
"St. Marks Maternity Hopital (SMMH)" = "St. Mark's Maternity Hospital (SMMH)"
)) %>%
mutate(hospital = replace_na(hospital, "Missing")) %>%
# создаем столбец age_years (из age и age_unit)
mutate(age_years = case_when(
age_unit == "years" ~ age,
age_unit == "months" ~ age/12,
is.na(age_unit) ~ age)) %>%
# ВЫШЕ ПОКАЗАНЫ РАНЕЕ ПРОДЕЛАННЫЕ И ПРЕДСТАВЛЕННЫЕ ШАГИ ВЫЧИСТКИ
###################################################
mutate(
# возрастные категории: пользовательские
age_cat = epikit::age_categories(age_years, breakers = c(0, 5, 10, 15, 20, 30, 50, 70)),
# возрастные категории: от 0 до 85 по 5 лет
age_cat5 = epikit::age_categories(age_years, breakers = seq(0, 85, 5)))8.10 Добавление строк
По одной
Добавление строк вручную по одной является трудоемким процессом, но это можно сделать с помощью add_row() из пакета dplyr. Помните, что каждый столбец должен содержать значения только из одного класса (либо текстовые, либо числовые, либо логические и т.п.). Поэтому добавление строки требует определенных нюансов для сохранения этой структуры.
linelist <- linelist %>%
add_row(row_num = 666,
case_id = "abc",
generation = 4,
`infection date` = as.Date("2020-10-10"),
.before = 2)Используйте .before и .after. для уточнения места размещения добавляемой строки. .before = 3 разместит новую строку перед текущей 3-й строкой. По умолчанию строка добавляется в конце. Не уточненные столбцы останутся пустыми (NA).
Новый номер строки может выглядеть странно (“…23”) но номера строк ранее существовавших строк изменились. Поэтому если вы дважды используете команду, внимательно рассмотрите/протестируйте вставку.
Если класс, который вы задаете, не сходится, возникнет такое сообщение об ошибке:
Error: Can't combine ..1$infection date <date> and ..2$infection date <character>(При вставке строки со значением даты, помните, что нужно дату нужно указать в функции as.Date(), например, as.Date("2020-10-10")).
Связывание строк
Чтобы объединить наборы данных путем связывания строк одного датафрейма с нижней частью другого датафрейма, вы можете использовать bind_rows() из пакета dplyr. Это более подробно объясняется на странице Соединение данных.
8.11 Фильтрация строк
Типичный шаг вычистки после того, как вы провели вычистку столбцов и перекодировали значения, включает фильтрацию датафрейма до конкретных строк, используя глагольную функцию filter() из пакета dplyr.
Внутри функции filter(), увкажите логику, которая должна быть истинной TRUE, чтобы сохранить строку в наборе данных. Ниже мы покажем, как отфильтровать строки на основе простых и сложных логических условий.
Простой фильтр
Этот простой пример переопределяет датафрейм linelist как самого себя после фильтрации строк, чтобы они соответствовали логическому условию. Сохраняются только те строки, где логическое утверждение внутри скобок оценивается как истинное TRUE.
В данном примере логическим утверждением является gender == "f", которое проверяет, является ли значение в столбце gender равным “f” (чувствительно к регистру).
До применения фильтра количество строк в linelist равно nrow(linelist).
linelist <- linelist %>%
filter(gender == "f") # сохраняются только те строки, где пол равен "f"После применения фильтра количество строк в linelist равно linelist %>% filter(gender == "f") %>% nrow().
Отфильтровывание отсутствующих значений
Достаточно часто нужно отфильтровывать строки, в которых имеются отсутствующие значения. Не поддавайтесь желанию просто записать filter(!is.na(column) & !is.na(column)), а вместо этого используйте функцию tidyr, которая специально создана для этой цели: drop_na(). При выполнении с пустыми скобками она удаляет строки с любыми отсутствующими значениями. Альтернативно, вы можете указать имена конкретных столбцов, которые надо оценить на предмет отсутствия значений, либо использовать функции-помощники “tidyselect”, описанные выше.
linelist %>%
drop_na(case_id, age_years) # удаляет строки с отсутствующими значениями в case_id или age_yearsСм. страницу Отсутствующие данные, где показаны многие приемы для анализа и управления отсутствующими значениями в ваших данных.
Фильтр по номеру строки
В датафрейме или таблице tibble у каждой строки, как правило, будет “номер строки”, который (при просмотре в окне просмотра R Viewer) появляется слева от первого столбца. Этот номер сам по себе не является истинным столбцом данных, но он может быть использован в утверждении для filter().
Чтобы отфильтровать по “номеру строки”, вы можете использовать функцию row_number() из dplyr с открытыми скобками, как часть логического утверждения для фильтрации. Часто нужно использовать оператор %in% и диапазон чисел как часть логического утверждения, как показано ниже. Чтобы увидеть первое N количество строк, вы можете также использовать специальную функцию из dplyr head().
# Просмотр первых 100 строк
linelist %>% head(100) # или используйте tail(), чтобы просмотреть последнее n количество строк
# Показать только 5 строку
linelist %>% filter(row_number() == 5)
# Просмотреть строки с 2 по 20, а также три конкретных столбца
linelist %>% filter(row_number() %in% 2:20) %>% select(date_onset, outcome, age)Вы также можете конвертировать номера строк в истинный столбец, передав ваш датафрейм в функцию tibble rownames_to_column() (ничего не пишите в скобках).
Сложный фильтр
Можно выстроить более сложные логические утверждения с использованием скобок ( ), операторов ИЛИ |, отрицание !, %in%, а также И &. Пример приведен ниже:
Примечание: Вы можете использовать оператор ! перед логическим критериями, чтобы их отрицать. Например, !is.na(column) оценивает как истину, если значения столбца не отсутствует. Аналогично, !column %in% c("a", "b", "c") оценивает как истину, если значение столбца не является вектором.
Рассмотрение данных
Ниже представлена простая команда из одной строки для создания гистограммы даты заболевания. Обратите внимание, что вторая более мелкая вспышка 2012-2013 гг. также включена в этот сырой набор данных. Для нашего анализа нам нужно удалить записи по этой более ранней вспышке.
Как фильтры работают с отсутствующими числовыми значениями и датами
Можем ли мы просто отфильтровать по date_onset до строк только после июня 2013? Внимание! Применение кода filter(date_onset > as.Date("2013-06-01"))) удалит строки в более поздней эпидемии с отсутствующей датой заболевания!
ВНИМАНИЕ: Фильтра по больше чем (>) или меньше чем (<) дата или число может удалить любые строки с отсутствующими значениями (NA)! Это происходит потому, что NA рассматривается как бесконечно большое или малое число.
(См. страницу Работа с датами для получения более детальной информации о работе с датами и пакете lubridate)
Дизайн фильтра
Рассмотрите кросс-табуляцию, чтобы убедиться, что мы исключили только нужные строки:
table(Hospital = linelist$hospital, # название больницы
YearOnset = lubridate::year(linelist$date_onset), # год в date_onset
useNA = "always") # показать отсутствующие значения YearOnset
Hospital 2012 2013 2014 2015 <NA>
Central Hospital 0 0 351 99 18
Hospital A 229 46 0 0 15
Hospital B 227 47 0 0 15
Military Hospital 0 0 676 200 34
Missing 0 0 1117 318 77
Other 0 0 684 177 46
Port Hospital 9 1 1372 347 75
St. Mark's Maternity Hospital (SMMH) 0 0 322 93 13
<NA> 0 0 0 0 0По каким еще критериям мы можем отфильтровать для удаления первой вспышки (в 2012 и2013) из набора данных? Мы видим, что:
- Первая эпидемия в 2012 и 2013 годах произошла в больницах Hospital A, Hospital B, а также было 10 случаев в Port Hospital.
- Больницы Hospitals A и B не имели случаев во второй эпидемии, а в Port Hospital они были.
Мы хотим исключить:
-
nrow(linelist %>% filter(hospital %in% c("Hospital A", "Hospital B") | date_onset < as.Date("2013-06-01")))строк и началом в 2012 и 2013 в больницах hospital A, B, или Port:- Исключить
nrow(linelist %>% filter(date_onset < as.Date("2013-06-01")))строк с началом в 2012 и 2013 - Исключить
nrow(linelist %>% filter(hospital %in% c('Hospital A', 'Hospital B') & is.na(date_onset)))строк из Hospitals A и B с отсутствующими датами заболевания
-
Не исключать
nrow(linelist %>% filter(!hospital %in% c('Hospital A', 'Hospital B') & is.na(date_onset)))других строк с отсутствующими датами заболевания.
- Исключить
Мы начинаем с пострточного списка из nrow(linelist)` строк. Ниже представлено наше утверждение для фильтра:
linelist <- linelist %>%
# сохраняет строки, где дата заболевания после 1 июня 2013 ИЛИ где отсутствует дата заболевания и это была больница КРОМЕ Hospital A или B
filter(date_onset > as.Date("2013-06-01") | (is.na(date_onset) & !hospital %in% c("Hospital A", "Hospital B")))
nrow(linelist)[1] 6019Когда мы повторно делаем кросс-табуляцию, мы видим, что Hospitals A и B были полностью удалены, также удалены 10 случаев из больницы Port Hospital от 2012 и 2013 года, а другие значения остались - как мы и хотели.
table(Hospital = linelist$hospital, # название больницы
YearOnset = lubridate::year(linelist$date_onset), # год date_onset
useNA = "always") # показать отсутствующие значения YearOnset
Hospital 2014 2015 <NA>
Central Hospital 351 99 18
Military Hospital 676 200 34
Missing 1117 318 77
Other 684 177 46
Port Hospital 1372 347 75
St. Mark's Maternity Hospital (SMMH) 322 93 13
<NA> 0 0 0В одну команду фильтра можно включить несколько утверждений (отделенных запятыми), либо вы всегда можете поставить в канал отдельную команду filter() для ясности.
Примечание: некоторые читатели могут заметить, что было бы проще просто отфильтровать по date_hospitalisation, поскольку этот столбец заполнен на 100% без отсутствующих значений. Это верно. Но date_onset использовался для демонстрации сложного фильтра.
Отдельная команда
Фильтрацию можно также выполнять как отдельную команду (не как часть цепочки канала). Как и другие глагольные функции dplyr, в данном случае первым аргументом должен быть сам набор данных.
# датафрейм <- фильтр(датафрейм, условие(я) для сохранения строк)
linelist <- filter(linelist, !is.na(case_id))Вы можете также использовать базовый R, для выделения подмножества, используя квадратные скобки, которые отражают [строки, столбцы], которые вы хотите сохранить.
# датафрейм <- датафрейм[условия строки, условия столбца] (пустое пространство означает сохранить все)
linelist <- linelist[!is.na(case_id), ]Быстрое рассмотрение записей
Часто вам может быть необходимо быстро просмотреть несколько записей только по некоторым столбцам. Базовая функция R View() напечатает датафрейм для просмотра в RStudio.
Просмотр построчного списка в RStudio:
View(linelist)Здесь приведены два примера просмотра конкретных ячеек (конкретные строки и конкретные столбцы):
С функциями dplyr filter() и select():
В функции View() передайте набор данных в функцию filter(), чтобы сохранить определенные строки, затем select() для сохранения определенных столбцов. Например, чтобы рассмотреть даты заболевания и госпитализации по 3 конкретным случаям:
View(linelist %>%
filter(case_id %in% c("11f8ea", "76b97a", "47a5f5")) %>%
select(date_onset, date_hospitalisation))Вы можете сделать то же самое с синтаксисом базового R, используя квадратные скобки [ ], чтобы определить подмножество, которое вы хотите видеть.
View(linelist[linelist$case_id %in% c("11f8ea", "76b97a", "47a5f5"), c("date_onset", "date_hospitalisation")])Добавление в цепочку канала
# ЦЕПОЧКА КАНАЛА ВЫЧИСТКИ (начинается с сырых данных, которые передаются по шагам вычистки)
##################################################################################
# начало цепочки канала вычистки
###########################
linelist <- linelist_raw %>%
# стандартизируем синтаксис имен столбцов
janitor::clean_names() %>%
# переименовываем столбцы вручную
# НОВОЕ имя # СТАРОЕ имя
rename(date_infection = infection_date,
date_hospitalisation = hosp_date,
date_outcome = date_of_outcome) %>%
# удаляем столбцы
select(-c(row_num, merged_header, x28)) %>%
# дедупликация
distinct() %>%
# добавляем столбец
mutate(bmi = wt_kg / (ht_cm/100)^2) %>%
# конвертируем класс столбцов
mutate(across(contains("date"), as.Date),
generation = as.numeric(generation),
age = as.numeric(age)) %>%
# добавляем столбец: задержка госпитализации
mutate(days_onset_hosp = as.numeric(date_hospitalisation - date_onset)) %>%
# чистые значения столбца hospital
mutate(hospital = recode(hospital,
# СТАРОЕ = НОВОЕ
"Mitylira Hopital" = "Military Hospital",
"Mitylira Hospital" = "Military Hospital",
"Military Hopital" = "Military Hospital",
"Port Hopital" = "Port Hospital",
"Central Hopital" = "Central Hospital",
"other" = "Other",
"St. Marks Maternity Hopital (SMMH)" = "St. Mark's Maternity Hospital (SMMH)"
)) %>%
mutate(hospital = replace_na(hospital, "Missing")) %>%
# создаем столбец age_years (из age и age_unit)
mutate(age_years = case_when(
age_unit == "years" ~ age,
age_unit == "months" ~ age/12,
is.na(age_unit) ~ age)) %>%
mutate(
# возрастные категории: пользовательские
age_cat = epikit::age_categories(age_years, breakers = c(0, 5, 10, 15, 20, 30, 50, 70)),
# возрастные категории: от 0 до 85 по 5 лет
age_cat5 = epikit::age_categories(age_years, breakers = seq(0, 85, 5))) %>%
# ВЫШЕ ПОКАЗАНЫ РАНЕЕ ПРОДЕЛАННЫЕ И ПРЕДСТАВЛЕННЫЕ ШАГИ ВЫЧИСТКИ
###################################################
filter(
# сохраняем только строки, где case_id не отсутствует
!is.na(case_id),
# также фильтруем, чтобы сохранить только вторую вспышку
date_onset > as.Date("2013-06-01") | (is.na(date_onset) & !hospital %in% c("Hospital A", "Hospital B")))8.12 Построчные расчеты
Если вы хотите провести расчеты внутри строки, вы можете использовать функцию rowwise() из dplyr. См. эту онлайн виньетку по построчным расчетам.
Например, этот код применяет rowwise() и затем создает новый столбец, который складывает количество указанных столбцов симптомов, которые имеют значение “yes” (да), для каждой строки в построчном списке. Столбцы указываются внутри функции sum() по имени внутри вектора c(). rowwise(), по сути, является особым типом group_by(), так что лучше использовать ungroup(), когда вы закончите (см. страницу Группирование данных).
linelist %>%
rowwise() %>%
mutate(num_symptoms = sum(c(fever, chills, cough, aches, vomit) == "yes")) %>%
ungroup() %>%
select(fever, chills, cough, aches, vomit, num_symptoms) # for display# A tibble: 5,888 × 6
fever chills cough aches vomit num_symptoms
<chr> <chr> <chr> <chr> <chr> <int>
1 no no yes no yes 2
2 <NA> <NA> <NA> <NA> <NA> NA
3 <NA> <NA> <NA> <NA> <NA> NA
4 no no no no no 0
5 no no yes no yes 2
6 no no yes no yes 2
7 <NA> <NA> <NA> <NA> <NA> NA
8 no no yes no yes 2
9 no no yes no yes 2
10 no no yes no no 1
# ℹ 5,878 more rowsПри указании столбцов для оценки вы можете использовать функции-помощники “tidyselect”, описанные в разделе select() данной страницы. Вам может потребоваться лишь одна корректировка (поскольку вы не используете их внутри функции dplyr, такой как select() или summarise()).
Поместите критерии, уточняющие столбец, внутрь функции c_across() из dplyr. Это необходимо сделать, поскольку c_across (документация) разработан для работы конкретно с rowwise(). Например, следующий код:
- Применяет
rowwise(), так что следующая операция (sum()) применяется к каждой строке (не сумма по всем столбцам)
- Создает новый столбец
num_NA_dates, определенный для каждой строки как число столбцов (с именем, содержащим “date”), для которогоis.na()оценивается как TRUE (истина) (в них отсутствуют данные).
-
ungroup()для удаления эффектов от примененияrowwise()для последующих шагов
linelist %>%
rowwise() %>%
mutate(num_NA_dates = sum(is.na(c_across(contains("date"))))) %>%
ungroup() %>%
select(num_NA_dates, contains("date")) # для отображения# A tibble: 5,888 × 5
num_NA_dates date_infection date_onset date_hospitalisation date_outcome
<int> <date> <date> <date> <date>
1 1 2014-05-08 2014-05-13 2014-05-15 NA
2 1 NA 2014-05-13 2014-05-14 2014-05-18
3 1 NA 2014-05-16 2014-05-18 2014-05-30
4 1 2014-05-04 2014-05-18 2014-05-20 NA
5 0 2014-05-18 2014-05-21 2014-05-22 2014-05-29
6 0 2014-05-03 2014-05-22 2014-05-23 2014-05-24
7 0 2014-05-22 2014-05-27 2014-05-29 2014-06-01
8 0 2014-05-28 2014-06-02 2014-06-03 2014-06-07
9 1 NA 2014-06-05 2014-06-06 2014-06-18
10 1 NA 2014-06-05 2014-06-07 2014-06-09
# ℹ 5,878 more rowsВы также можете задать другие функции, такие как max(), чтобы получить последнюю или наиболее недавнюю дату для каждой строки:
linelist %>%
rowwise() %>%
mutate(latest_date = max(c_across(contains("date")), na.rm=T)) %>%
ungroup() %>%
select(latest_date, contains("date")) # для отображения# A tibble: 5,888 × 5
latest_date date_infection date_onset date_hospitalisation date_outcome
<date> <date> <date> <date> <date>
1 2014-05-15 2014-05-08 2014-05-13 2014-05-15 NA
2 2014-05-18 NA 2014-05-13 2014-05-14 2014-05-18
3 2014-05-30 NA 2014-05-16 2014-05-18 2014-05-30
4 2014-05-20 2014-05-04 2014-05-18 2014-05-20 NA
5 2014-05-29 2014-05-18 2014-05-21 2014-05-22 2014-05-29
6 2014-05-24 2014-05-03 2014-05-22 2014-05-23 2014-05-24
7 2014-06-01 2014-05-22 2014-05-27 2014-05-29 2014-06-01
8 2014-06-07 2014-05-28 2014-06-02 2014-06-03 2014-06-07
9 2014-06-18 NA 2014-06-05 2014-06-06 2014-06-18
10 2014-06-09 NA 2014-06-05 2014-06-07 2014-06-09
# ℹ 5,878 more rows8.13 Упорядочивание и сортировка
Используйте функцию arrange() из dplyr для сортировки или упорядочивания строк по значениям столбцов.
Просто перечислите столбцы в порядке, в котором они должны быть отсортированы. Укажите .by_group = TRUE, если вы хотите, чтобы сортировка сначала произошла по любым группировкам, применяемым к данным (см. страницу Группирование данных).
По умолчанию столбец будет отсортирован в “возрастающем” порядке (который применяется как к числовым, так и текстовым столбцам). Вы можете отсортировать переменную в “убывающем” порядке, обернув ее в desc().
Сортировка данных с помощью arrange() особенно полезна, когда вы готовите Таблицы для презентации, используя slice(), чтобы взять “верхние” строки в каждой группе, либо чтобы задать порядок уровня факторов по порядку появления.
Например, чтобы отсортировать строки нашего построчного списка по больницам hospital, затем по дате заболевания date_onset в убывающем порядке, мы используем:
linelist %>%
arrange(hospital, desc(date_onset))