24 Моделирование эпидемий
24.1 Обзор
Существует растущий набор инструментов моделирования эпидемий, который позволяет нам проводить достаточно сложные виды анализа при минимальных усилиях. В данном разделе представлен обзор того, как использовать эти инструменты для следующих целей:
- оценка эффективного репродуктивного числа Rt и связанной с ним статистических показателей, таких как время удваивания
- подготовка краткосрочных прогнозов будущей заболеваемости
Этот раздел не является обзором методологий и статистических методов, лежащих в основе этих инструментов, для работ по этой теме см. вкладку Ресурсы. Убедитесь, что вы понимаете эти методы, прежде чем начнете использовать эти инструменты; это позволит вам правильно интерпретировать полученные результаты.
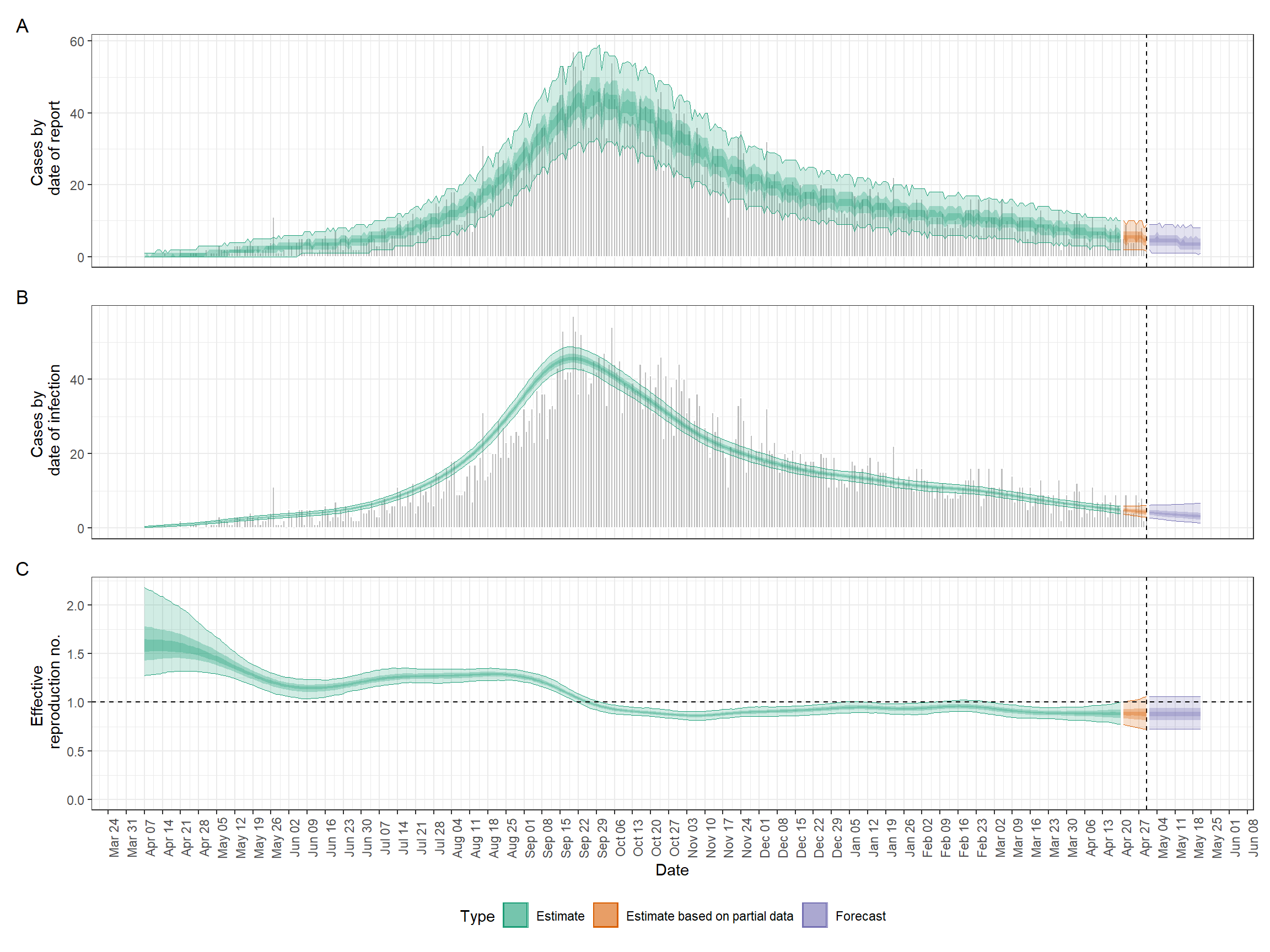
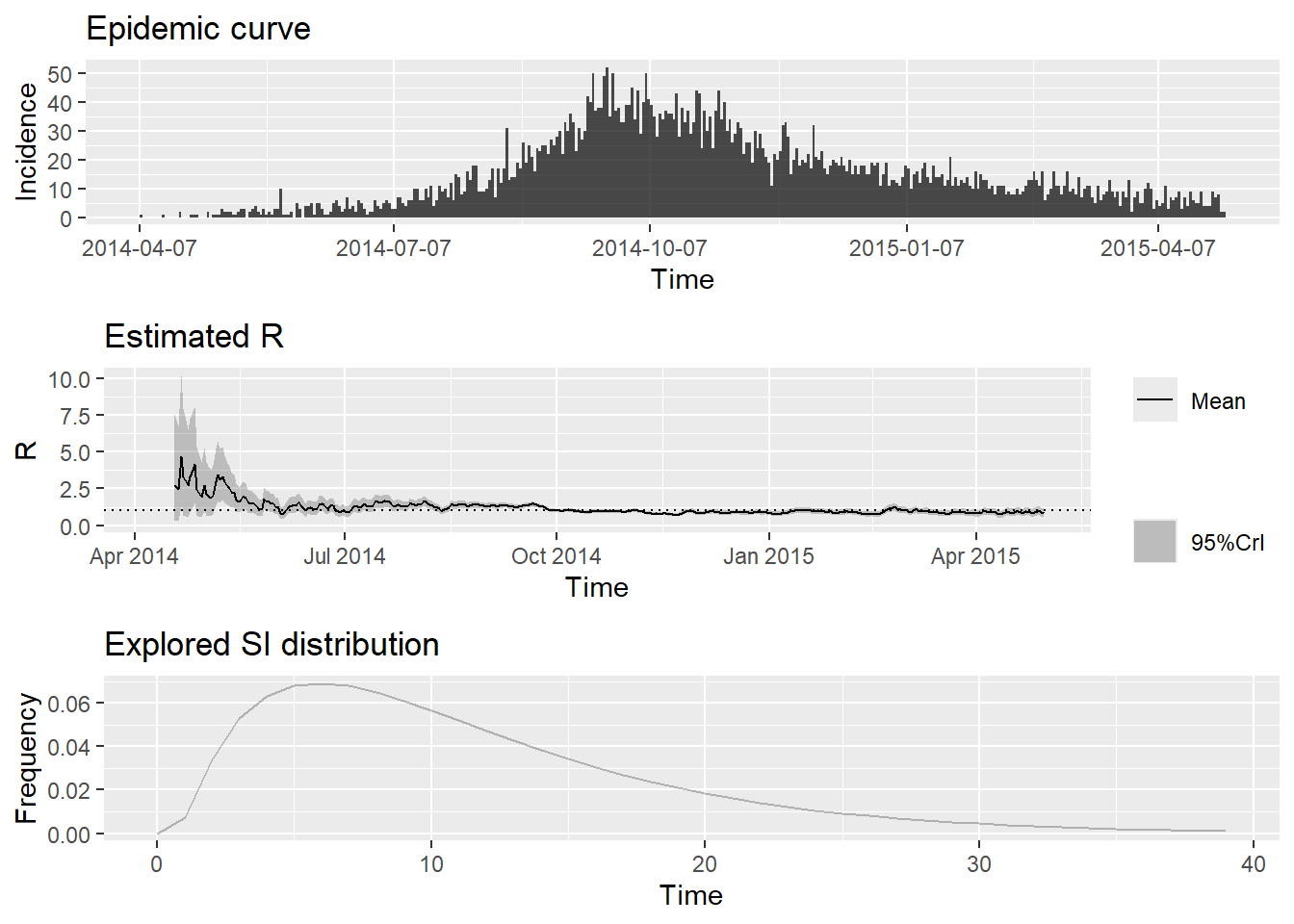
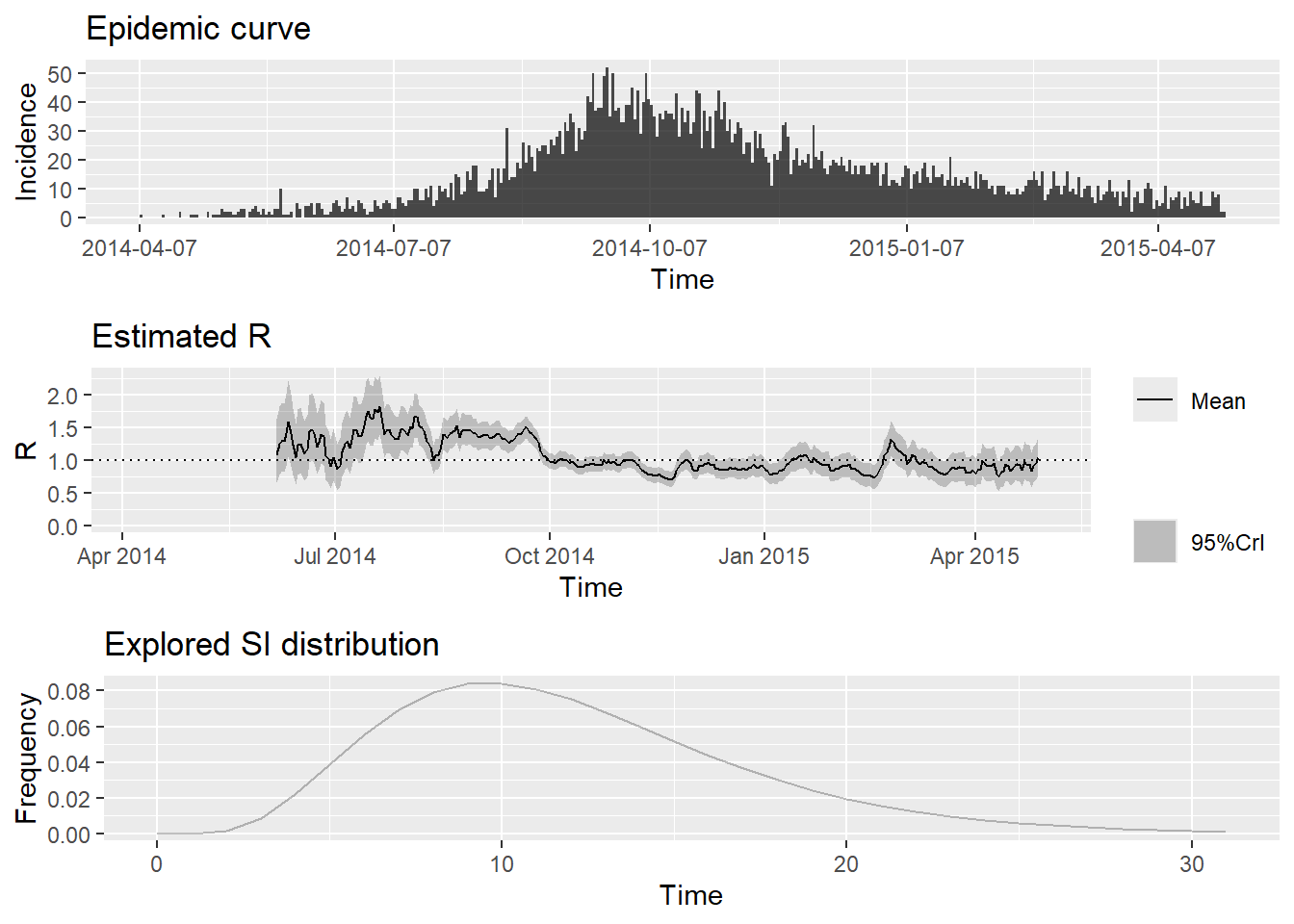
Ниже приведен пример выходных данных, которые мы подготовим в этом разделе.
24.2 Подготовка
Мы будем использовать два разных методов и пакетов для оценки Rt, в частности, EpiNow и EpiEstim, а также пакет projections для прогнозирования заболеваемости.
Данный фрагмент кода показывает загрузку пакетов, необходимых для анализа. В настоящем руководстве мы фокусируемся на использовании p_load() из pacman, которая устанавливает пакет, если необходимо, и загружает его для использования. Вы можете также установить пакеты с помощью library() из базового R. См. страницу Основы R для получения дополнительной информации о пакетах R.
pacman::p_load(
rio, # импорт файлов
here, # расположение файла
tidyverse, # управление данными + графика ggplot2
epicontacts, # анализ сетей передачи
EpiNow2, # оценка Rt
EpiEstim, # оценка Rt
projections, # прогнозирование заболеваемости
incidence2, # работа с данными по заболеваемости
epitrix, # полезные эпидемиологические функции
distcrete # дискретные распределения задержек
)Мы будем использовать вычищенный построчный список для всего анализа в данном разделе. Если вы хотите выполнять шаги параллельно, кликните, чтобы скачать “вычищенный” построчный список (как файл .rds). См. страницу Скачивание руководства и данных, чтобы скачать все примеры данных, используемые в данном руководстве.
# импорт вычищенного построчного списка
linelist <- import("linelist_cleaned.rds")24.3 Оценка Rt
EpiNow2 или EpiEstim
Репродуктивное число R - мера трансмиссивности заболевания и определяется как ожидаемое число вторичных случаев на один инфицированный случай. В полностью восприимчивой популяции, данное число представляет собой базовое репродуктивное число R0. Однако поскольку число восприимчивых лиц в популяции меняется в ходе вспышки или пандемии, а также по мере реализации различных мер реагирования, наиболее часто используемой мерой трансмиссивности является эффективное репродуктивное число Rt; оно определяется как ожидаемое число вторичных случаев на один инфицированный случай в определенное время t.
Пакет EpiNow2 дает наиболее продвинутые рамки для оценки Rt. У него имеются два ключевых преимущества по сравнению с другим часто используемым пакетом, EpiEstim:
- Он учитывает задержку в регистрации, следовательно может оценить Rt, даже если последние данные неполные.
- Он оценивает Rt в даты заражения, а не даты возникновения симпмтомов или регистрации, что означает, чтор эффект вмешательства будет немедленно отображаться в изменении в Rt, а не с задержкой.
Однако у него имеются два ключевых недостатка:
- Он требует знания распределения времени генерации (т.е. распределения задержки между заражением первичного и вторичных случаев), распределения инкубационного периода (т.е. распределения задержек между заражением и появлением симптомов), а также распределения других задержек, актуальных для ваших данных (например, если у вас есть даты отчетности, вам нужно распределение задержек от момента возникновения симптомов до даты отчетности). Хотя это обеспечивает более точную оценку Rt, EpiEstim требует только распределения серийного интервала (т.е. распределения задержек между появлением симптомов у первичного и вторичного случая), что может быть единственным доступным вам распределением.
- EpiNow2 - значительно более медленный, чем EpiEstim, в некоторых случаях в 100-1000 раз! Например, оценка Rt для примера вспышки, рассматриваемого в данном разделе, занимает около 4 часов (выполнено для большого количества итераций, чтобы обеспечить высокую точность, количество которых можно сократить, при необходимости, однако это не отменяет того, что алгоритм в целом работает медленно. Это может быть проблематичным, если вы регулярно обновляете свои оценки Rt.
То, какой пакет вы выберете, зависит от ваших данных, времени, а также доступных вычислительных мощностей.
EpiNow2
Оценка распределений задержек
Распределения задержек, требуемые для выполнения EpiNow2, зависят от имеющихся данных. По сути, вам нужно описать задержку от даты заражения до даты события, которое вы хотите использовать для оценки Rt. Если вы используете дату появления симптомов, это будет просто распределение инкубационного периода. Если вы используете дату регистрации, вам нужна задержка между заражением и регистрацией. Поскольку это распределение вряд ли будет известно напрямую, EpiNow2 позволяет вам связать несколько распределений задержек вместе; в данном случае, задержку от заражения до появления симптомов (т.е. инкубационный период, который, скорее всего, известен), а также с момента появления симптомов до регистрации (что вы часто можете оценить, исходя из своих данных).
Поскольку в нашем построчном списке есть даты возникновения симптомов для всех случаев, нам потребуется только распределение инкубационного периода, чтобы связать наши данные (т.е. даты возникновения симптомов) с датой заражения. Мы можем либо оценить это распределение исхода из данных, либо использовать данные из литературы.
В литературе оценочный инкубационный период Эболы (взят из этой работы) имеет среднее значение 9.1, стандартное отклонение 7.3, а максимальное значение 30, что записывается следующим образом:
incubation_period_lit <- list(
mean = log(9.1),
mean_sd = log(0.1),
sd = log(7.3),
sd_sd = log(0.1),
max = 30
)Обратите внимание, что EpiNow2 требует предоставления распределений задержек в логарифмической шкале, поэтому вокруг каждого значения имеется log (кроме параметра max, который, внезапно, должен указываться в натуральных величинах). mean_sd и sd_sd задают оценки стандартного отклонения среднего значения и стандартного отклонения. Поскольку они в данном случае неизвестны, мы выбираем условное значение 0.1.
В данном анализе мы вместо этого оцениваем распределение инкубационного периода из самого построчного списка, используя функцию bootstrapped_dist_fit, которая построит логнормальное распределение наблюдаемых задержек между инфекцией и возникновением симптомов в построчном списке.
## оценка инкубационного периода
incubation_period <- bootstrapped_dist_fit(
linelist$date_onset - linelist$date_infection,
dist = "lognormal",
max_value = 100,
bootstraps = 1
)Еще одно нужное распределение - время генерации. Поскольку у нас есть данные по времени заражения и цепочкам передачи, мы можем оценить это распределение из построчного списка, рассчитав задержку между временем заражения по парам инфицирующий-инфицируемый. Чтобы это сделать, мы используем удобную функцию get_pairwise из пакета epicontacts, который позволяет нам рассчитать попарные разницы по свойствам построчного списка между парами передачи. Сначала мы создадим объект эпидконтактов (epicontacts) (см. страницу [Цепочки передачи] для получения детальной информации):
## генерация контактов
contacts <- linelist %>%
transmute(
from = infector,
to = case_id
) %>%
drop_na()
## генерация объекта epicontacts
epic <- make_epicontacts(
linelist = linelist,
contacts = contacts,
directed = TRUE
)Затем мы строим разницу между временем заражения по парам передачи, рассчитываем используя get_pairwise, по гамма распределению:
## оцениваем гамма время генерации
generation_time <- bootstrapped_dist_fit(
get_pairwise(epic, "date_infection"),
dist = "gamma",
max_value = 20,
bootstraps = 1
)Выполняем EpiNow2
Теперь нам нужно рассчитать ежедневную заболеваемость из построчного списка, что мы легко можем сделать с помощью функций dplyr group_by() и n(). Обратите внимание, что EpiNow2 требует, чтобы названия столбцов были date и confirm.
## получаем заболеваемость из дат появления симптомов
cases <- linelist %>%
group_by(date = date_onset) %>%
summarise(confirm = n())Затем мы можем оценить Rt, используя функцию epinow. Несколько комментариев по вводным данным:
- Мы можем представить любое количество ‘сцепленных’ распределений задержки в аргумент
delays; Мы просто вставляем их рядом с объектомincubation_periodвнутри функцииdelay_opts. -
return_outputобеспечит выведение выходных данных в R, а не просто сохранение в файл. -
verboseуказывает, что мы хотим видеть вывод прогресса. -
horizonуказывает, на сколько дней мы хотим прогнозировать будущую заболеваемость. - Мы указываем дополнительные опции в аргументе
stan, чтобы указать, на какой срок мы хотем делать предположения. Увеличениеsamplesиchainsдаст вам более точную оценку, которая лучше характеризует неопределенность, однако потребует больше времени на вычисления.
## запуск epinow
epinow_res <- epinow(
reported_cases = cases,
generation_time = generation_time,
delays = delay_opts(incubation_period),
return_output = TRUE,
verbose = TRUE,
horizon = 21,
stan = stan_opts(samples = 750, chains = 4)
)Анализ выходных данных
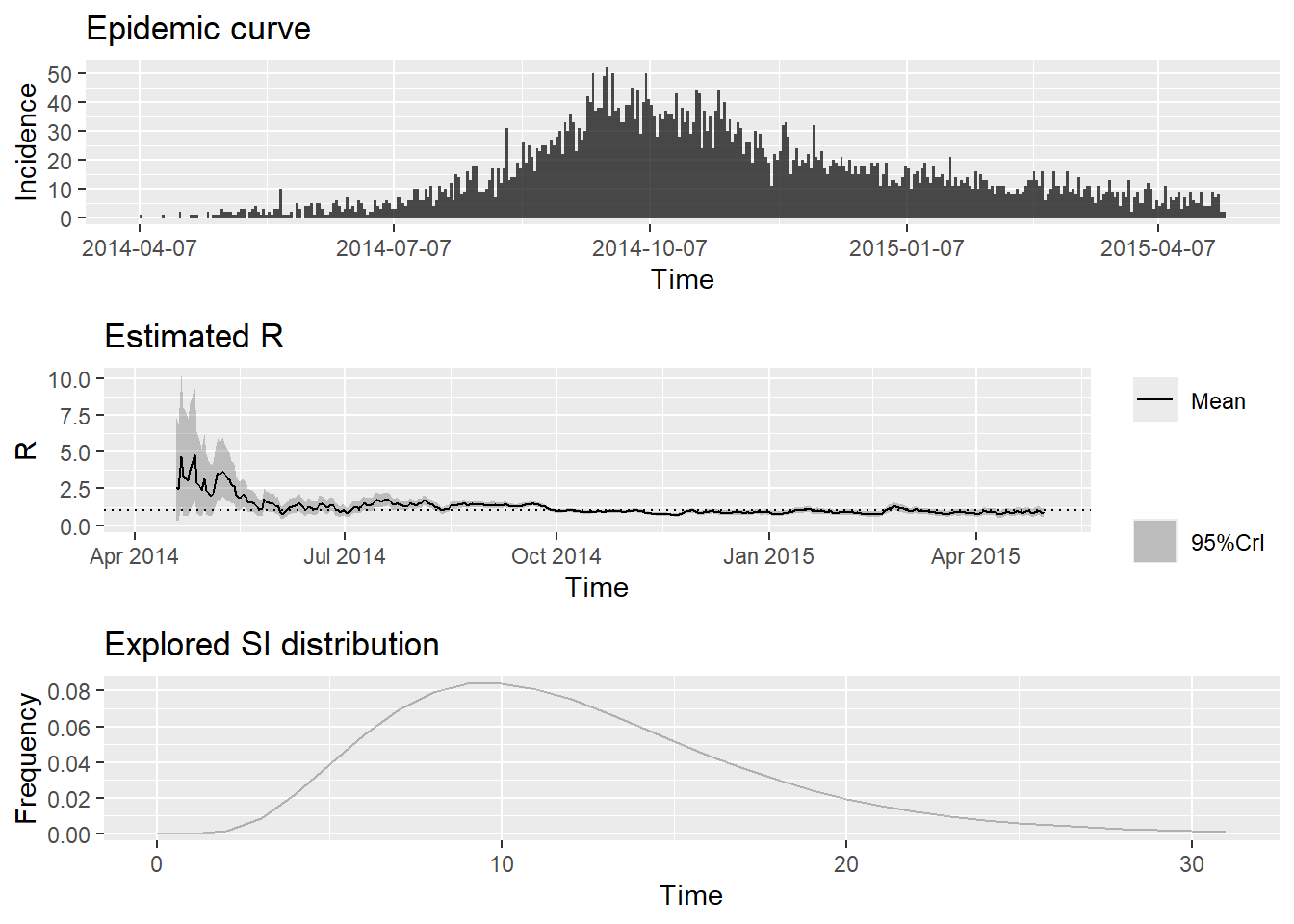
Как только закончится выполнение кода, мы можем легко отобразить сводную информацию. Пролистайте изображение, чтобы увидеть все детали.
Мы можем также рассмотреть сводную статистику:
## сводная таблица
epinow_res$summary measure estimate
<char> <char>
1: New confirmed cases by infection date 4 (2 -- 6)
2: Expected change in daily cases Unsure
3: Effective reproduction no. 0.88 (0.73 -- 1.1)
4: Rate of growth -0.012 (-0.028 -- 0.0052)
5: Doubling/halving time (days) -60 (130 -- -25)
numeric_estimate
<list>
1: <data.table[1x9]>
2: 0.56
3: <data.table[1x9]>
4: <data.table[1x9]>
5: <data.table[1x9]>Для дополнительного анализа и кастомизированного построения графиков, вы можете зайти в обобщенные ежедневные оценки с помощью $estimates$summarised. Мы конвертируем их из формата по умолчанию data.table в таблицу tibble для более удобного использования dplyr.
## извлечение сводной информации и конвертация в tibble
estimates <- as_tibble(epinow_res$estimates$summarised)
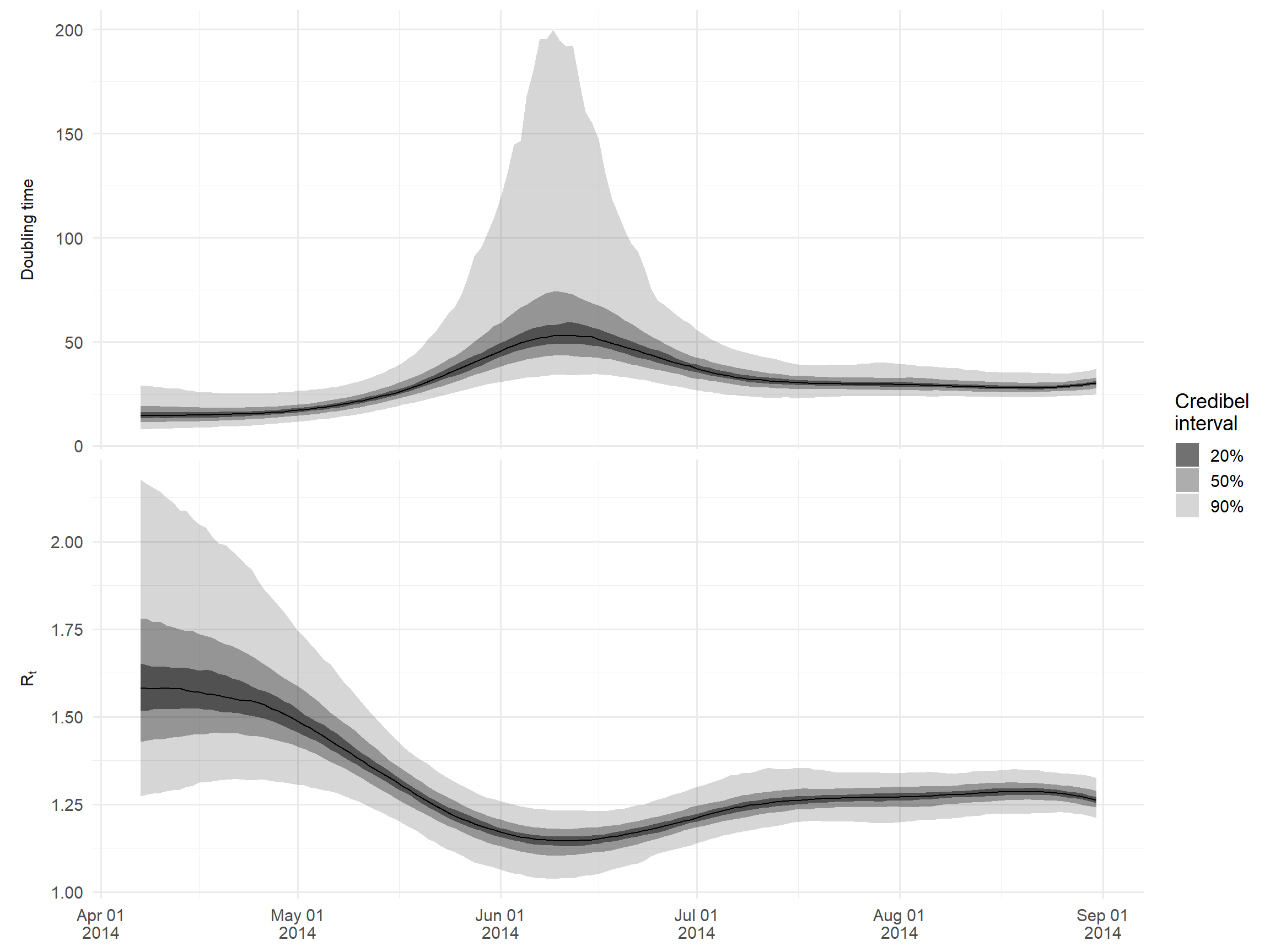
estimatesВ качестве примера давайте построим график времени удваивания и Rt. Мы посмотрим лишь первые несколько месяцев вспышки, когда Rt составляет значительно больше единицы, чтобы избежать построения очень высокого времени удваивания.
Мы используем формулу log(2)/growth_rate для расчета времени удваивания из оценочного темпа роста.
## составим широкий df для построения графика медианы
df_wide <- estimates %>%
filter(
variable %in% c("growth_rate", "R"),
date < as.Date("2014-09-01")
) %>%
## конвертируем темпы роста во время удваивания
mutate(
across(
c(median, lower_90:upper_90),
~ case_when(
variable == "growth_rate" ~ log(2)/.x,
TRUE ~ .x
)
),
## переименовываем переменные, чтобы отразить преобразование
variable = replace(variable, variable == "growth_rate", "doubling_time")
)
## составим длинный df для графика квантилей
df_long <- df_wide %>%
## здесь мы сопоставляем совпадающие квантили (например lower_90 к upper_90)
pivot_longer(
lower_90:upper_90,
names_to = c(".value", "quantile"),
names_pattern = "(.+)_(.+)"
)
## строим график
ggplot() +
geom_ribbon(
data = df_long,
aes(x = date, ymin = lower, ymax = upper, alpha = quantile),
color = NA
) +
geom_line(
data = df_wide,
aes(x = date, y = median)
) +
## используем label_parsed, чтобы разрешить подстрочную подпись
facet_wrap(
~ variable,
ncol = 1,
scales = "free_y",
labeller = as_labeller(c(R = "R[t]", doubling_time = "Doubling~time"), label_parsed),
strip.position = 'left'
) +
## вручную устанавливаем прозрачность квантилей
scale_alpha_manual(
values = c(`20` = 0.7, `50` = 0.4, `90` = 0.2),
labels = function(x) paste0(x, "%")
) +
labs(
x = NULL,
y = NULL,
alpha = "Credibel\ninterval"
) +
scale_x_date(
date_breaks = "1 month",
date_labels = "%b %d\n%Y"
) +
theme_minimal(base_size = 14) +
theme(
strip.background = element_blank(),
strip.placement = 'outside'
)EpiEstim
Чтобы выполнить EpiEstim, нам нужно задать данные по ежедневной заболеваемости и указать серийный интервал (т.е. распределение задержек между появлением симптомов первичных и вторичных случаев).
Данные о заболеваемости могут быть заданы в EpiEstim виде вектора, датафрейма или объекта incidence из оригинального пакета incidence. Вы можете даже различать завозные и местные случаи; см. документацию ?estimate_R для получения детальной информации.
Мы создадим входные данные, используя incidence2. См. дополнительные примеры с пакетом incidence2 на странице [Эпидемические кривые]. Поскольку произошли обновления пакета incidence2, которые не совсем совпадают с ожидаемыми входными данными для estimateR(), требуются некоторые дополнительные шаги. Объект заболеваемости состоит из таблицы tibble с датами и соответствующим им количеством случаев. Мы используем complete() из tidyr, чтобы убедиться, что все даты включены (даже те, когда не было случаев), а затем rename() для переименования столбцов, чтобы они соответствовали тому, чего будет ожидать estimate_R() на последующем шаге.
## получаем заболеваемость из даты появления симптомов
cases <- incidence2::incidence(linelist, date_index = "date_onset") %>% # получаем количество случаев по дням
tidyr::complete(date_index = seq.Date( # проверяем, что все даты представлены
from = min(date_index, na.rm = T),
to = max(date_index, na.rm=T),
by = "day"),
fill = list(count = 0)) %>% # конвертируем NA в 0
rename(I = count, # переименовываем в имена, которые ожидает estimateR
dates = date_index)Пакет дает несколько вариантов для указания серийного интервала, детальная информация о которых представленра в документации ?estimate_R. Мы рассмотрим два из них.
Использование оценок серийного интервала из литературы
Используя опцию method = "parametric_si", мы можем вручную установить среднее значение и стандартное отклонение серийного интервалав объекте config, созданном используя функцию make_config. Мы используем среднее значение и стандартное отклонение 12.0 и 5.2, соответственно, взятые из этой работы:
## создание config
config_lit <- make_config(
mean_si = 12.0,
std_si = 5.2
)Мы можем затем оценить Rt с помощью функции estimate_R:
cases <- cases %>%
filter(!is.na(date))
#создаем датафрейм для функции estimate_R()
cases_incidence <- data.frame(dates = seq.Date(from = min(cases$dates),
to = max(cases$dates),
by = 1))
cases_incidence <- left_join(cases_incidence, cases) %>%
select(dates, I) %>%
mutate(I = ifelse(is.na(I), 0, I))Joining with `by = join_by(dates)`epiestim_res_lit <- estimate_R(
incid = cases_incidence,
method = "parametric_si",
config = config_lit
)Default config will estimate R on weekly sliding windows.
To change this change the t_start and t_end arguments. и выводим сводные выходные данные:
Используем оценки серийного интервала из данных
Поскольку у нас есть данные по датам появления симптомов и цепочкам передачи, мы можем также оценить серийный интервал из построчного списка, рассчитав задержку между датами появления симптомов в парах инфицирующий-инфицируемый. Как мы это делали в разделе EpiNow2, мы используем функцию get_pairwise из пакета epicontacts, которая позволяет нам рассчитать попарные разницы в свойствах построчного списка между парами передачи. Сначала мы создаем объект epicontacts (см. страницу [Цепочки передачи] для получения детальной информации):
## генерируем контакты
contacts <- linelist %>%
transmute(
from = infector,
to = case_id
) %>%
drop_na()
## генерируем объект epicontacts
epic <- make_epicontacts(
linelist = linelist,
contacts = contacts,
directed = TRUE
)Затем мы строим разницу в датах появления симптомов между парами передачи, рассчитанную с помощью get_pairwise, по гамма распределению. Мы используем удобную функцию fit_disc_gamma из пакета epitrix для этой процедуры построения, так как нам нужно дискретезированное распределение.
## оцениваем гамма серийный интервал
serial_interval <- fit_disc_gamma(get_pairwise(epic, "date_onset"))Мы затем передаем эту информацию в объект config, выполняем снова EpiEstim и строим график результатов:
## создаем config
config_emp <- make_config(
mean_si = serial_interval$mu,
std_si = serial_interval$sd
)
## выполняем epiestim
epiestim_res_emp <- estimate_R(
incid = cases_incidence,
method = "parametric_si",
config = config_emp
)Default config will estimate R on weekly sliding windows.
To change this change the t_start and t_end arguments. ## строим график выходных данных
plot(epiestim_res_emp)Определение периода для времени оценки
Эти варианты по умолчанию предоставят недельную скользящую оценку и могут служить предупреждением о том, что оцениваете Rt на слишком раннем этапе вспышки, чтобы получить точную оценку. Вы можете изменить это, установив более позднюю дату начала для оценки, как показано ниже. К сожалению, EpiEstim дает только очень тяжелый способ указания этого времени оценки, там нужно задавать вектор целых чисел, относящийся к дате начала и окончания каждого временного периода.
## определяем вектор дат, начиная с 1 июня
start_dates <- seq.Date(
as.Date("2014-06-01"),
max(cases$dates) - 7,
by = 1
) %>%
## отнимаем дату начала, чтобы конвертировать в числовое значение
`-`(min(cases$dates)) %>%
## конвертируем в целое число
as.integer()
## добавляем 6 дней для скользящего недельного периода
end_dates <- start_dates + 6
## создаем config
config_partial <- make_config(
mean_si = 12.0,
std_si = 5.2,
t_start = start_dates,
t_end = end_dates
)Теперь повторно выполним EpiEstim и мы можем увидеть, что оценки начинаются с июня:
Анализ выходных данных
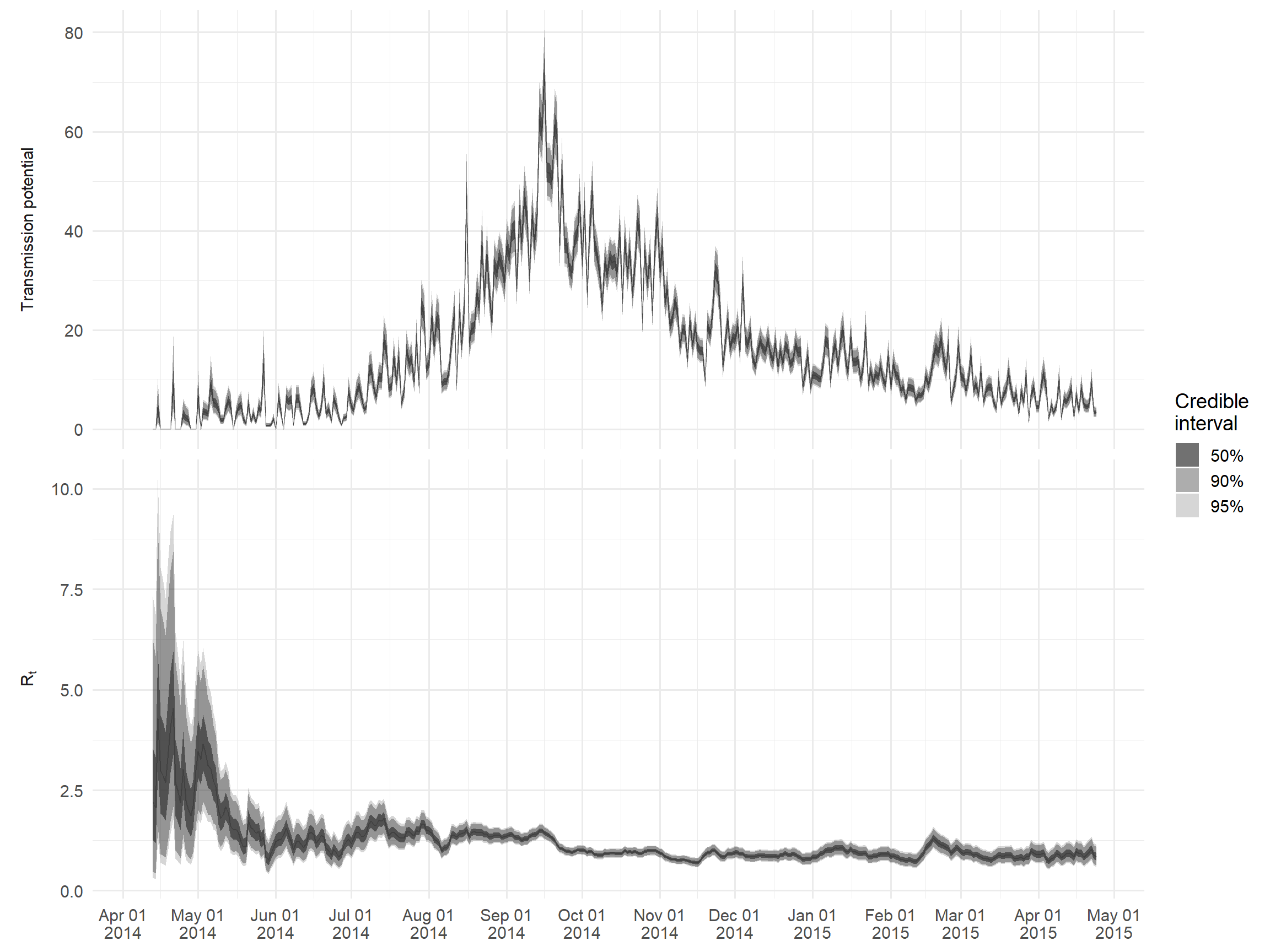
Доступ к основным выходным данным можно получить через $R. В качестве примера создадим график Rt и меру “потенциала распространения”, которую получаем из произведения Rt и числа случаев, зарегистрированных в этот день; это отражает ожидаемое количество случаев в следующем поколении инфекции.
## создаем широкий датафрейм для медианы
df_wide <- epiestim_res_lit$R %>%
rename_all(clean_labels) %>%
rename(
lower_95_r = quantile_0_025_r,
lower_90_r = quantile_0_05_r,
lower_50_r = quantile_0_25_r,
upper_50_r = quantile_0_75_r,
upper_90_r = quantile_0_95_r,
upper_95_r = quantile_0_975_r,
) %>%
mutate(
## извлекаем медианную дату из t_start и t_end
dates = epiestim_res_emp$dates[round(map2_dbl(t_start, t_end, median))],
var = "R[t]"
) %>%
## объединяем с данными по ежедневной заболеваемости
left_join(cases, "dates") %>%
## рассчитываем риск по всем оценкам r
mutate(
across(
lower_95_r:upper_95_r,
~ .x*I,
.names = "{str_replace(.col, '_r', '_risk')}"
)
) %>%
## отделяем оценки r и оценки рисков
pivot_longer(
contains("median"),
names_to = c(".value", "variable"),
names_pattern = "(.+)_(.+)"
) %>%
## присваиваем уровни фактора
mutate(variable = factor(variable, c("risk", "r")))
## создаем длинный датафрейм из квантилей
df_long <- df_wide %>%
select(-variable, -median) %>%
## отделяем оценки r/рисков и уровни квантилей
pivot_longer(
contains(c("lower", "upper")),
names_to = c(".value", "quantile", "variable"),
names_pattern = "(.+)_(.+)_(.+)"
) %>%
mutate(variable = factor(variable, c("risk", "r")))
## создаем график
ggplot() +
geom_ribbon(
data = df_long,
aes(x = dates, ymin = lower, ymax = upper, alpha = quantile),
color = NA
) +
geom_line(
data = df_wide,
aes(x = dates, y = median),
alpha = 0.2
) +
## используем label_parsed, чтобы разрешить подстрочные подписи
facet_wrap(
~ variable,
ncol = 1,
scales = "free_y",
labeller = as_labeller(c(r = "R[t]", risk = "Transmission~potential"), label_parsed),
strip.position = 'left'
) +
## вручную задаем прозрачность квантилей
scale_alpha_manual(
values = c(`50` = 0.7, `90` = 0.4, `95` = 0.2),
labels = function(x) paste0(x, "%")
) +
labs(
x = NULL,
y = NULL,
alpha = "Credible\ninterval"
) +
scale_x_date(
date_breaks = "1 month",
date_labels = "%b %d\n%Y"
) +
theme_minimal(base_size = 14) +
theme(
strip.background = element_blank(),
strip.placement = 'outside'
)24.4 Прогнозирование заболеваемости
EpiNow2
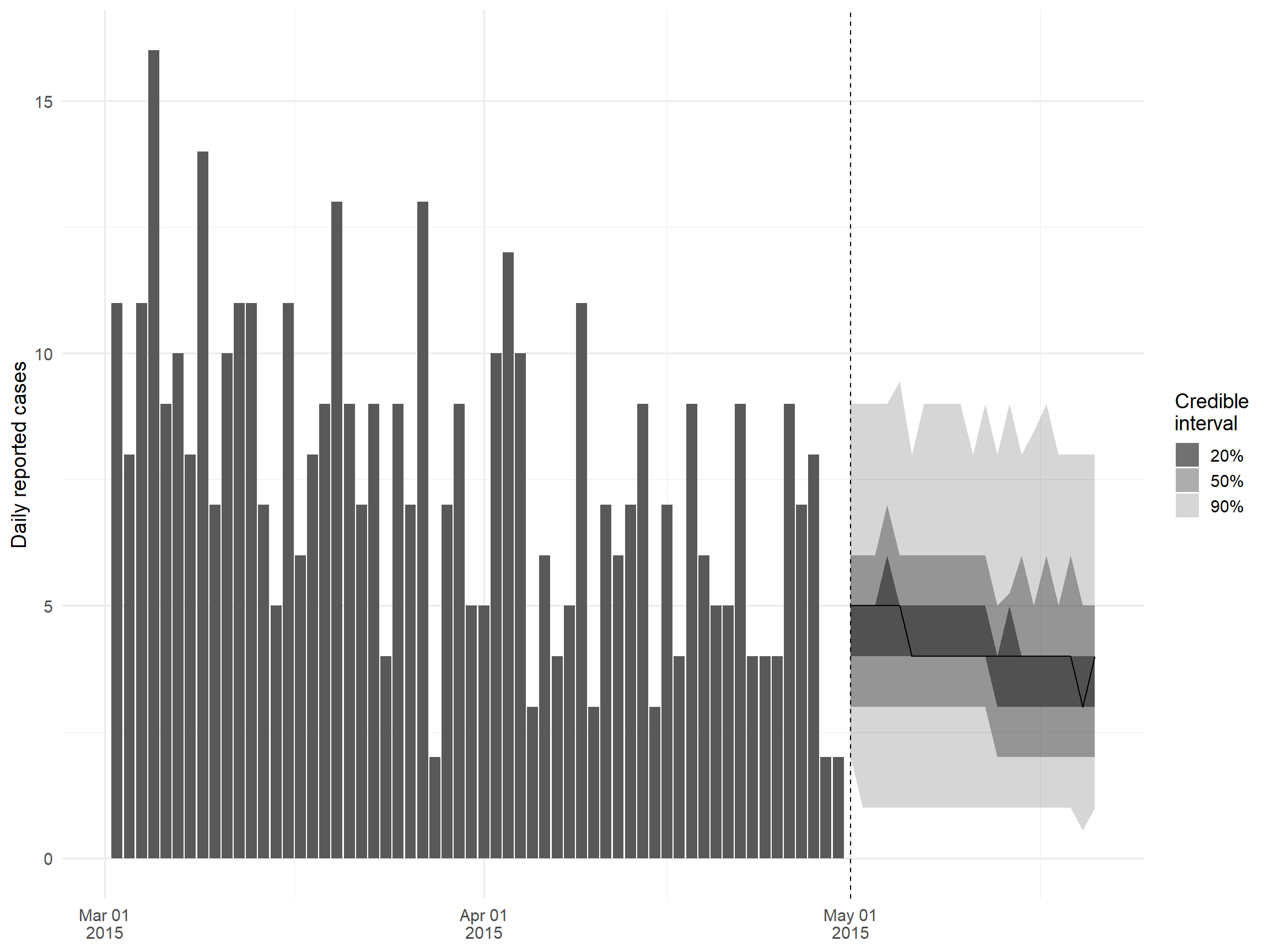
Кроме оценки Rt, EpiNow2 также поддерживает прогнозирование Rt и прогнозирование количества случаев с помощью внутренней интеграфии с пакетом EpiSoon. Вам нужно лишь задать аргумент horizon в функции epinow, указав на сколько дней в будущее вам нужен прогноз; см. раздел EpiNow2 в “Оценка Rt” для получения дополнительной информации о том, как получить и настроить EpiNow2. В данном разделе мы просто построим график выходных данных из анализа, хранящихся в объекте epinow_res.
## определяем минимальную дату для графика
min_date <- as.Date("2015-03-01")
## извлекаем сводные оценки
estimates <- as_tibble(epinow_res$estimates$summarised)
## извлекаем сырые данные по заболеваемости
observations <- as_tibble(epinow_res$estimates$observations) %>%
filter(date > min_date)
## извлекаем прогнозируемые оценки количества случаев
df_wide <- estimates %>%
filter(
variable == "reported_cases",
type == "forecast",
date > min_date
)
## конвертируем в еще более длинный формат для построения графика квантилей
df_long <- df_wide %>%
## здесь мы сопоставляем соответствующие квантили (например, lower_90 с upper_90)
pivot_longer(
lower_90:upper_90,
names_to = c(".value", "quantile"),
names_pattern = "(.+)_(.+)"
)
## создаем график
ggplot() +
geom_histogram(
data = observations,
aes(x = date, y = confirm),
stat = 'identity',
binwidth = 1
) +
geom_ribbon(
data = df_long,
aes(x = date, ymin = lower, ymax = upper, alpha = quantile),
color = NA
) +
geom_line(
data = df_wide,
aes(x = date, y = median)
) +
geom_vline(xintercept = min(df_long$date), linetype = 2) +
## вручную задаем прозрачность квантилей
scale_alpha_manual(
values = c(`20` = 0.7, `50` = 0.4, `90` = 0.2),
labels = function(x) paste0(x, "%")
) +
labs(
x = NULL,
y = "Daily reported cases",
alpha = "Credible\ninterval"
) +
scale_x_date(
date_breaks = "1 month",
date_labels = "%b %d\n%Y"
) +
theme_minimal(base_size = 14)Прогнозы
Пакет projections, разработанный RECON, облегчает создание краткосрочных прогнозов заболеваемости, он требует только знания эффективного репродуктивного числа Rt и серийного интервала. Здесь мы рассмотрим, как использовать оценки серийного интервала из литературы, а также как использовать наши собственные оценки из построчного списка.
Использование оценок серийного интервала из литературы
projections требует дискретизированного распределения серийного интервала класса distcrete из пакета distcrete. Мы будем использовать гамма распределение со средним значением 12.0 и стандартным отклонением 5.2, взятое из этой работы. Чтобы конвертировать эти значения в параметры формы и шкалы, требуемые для гамма распределения, мы будем использовать функцию gamma_mucv2shapescale из пакета epitrix.
## получаем параметры формы и шкалы из среднего значения mu и коэффициента
## вариации (например, коэффициент стандартного отклонения к среднему)
shapescale <- epitrix::gamma_mucv2shapescale(mu = 12.0, cv = 5.2/12)
## создаем объект distcrete
serial_interval_lit <- distcrete::distcrete(
name = "gamma",
interval = 1,
shape = shapescale$shape,
scale = shapescale$scale
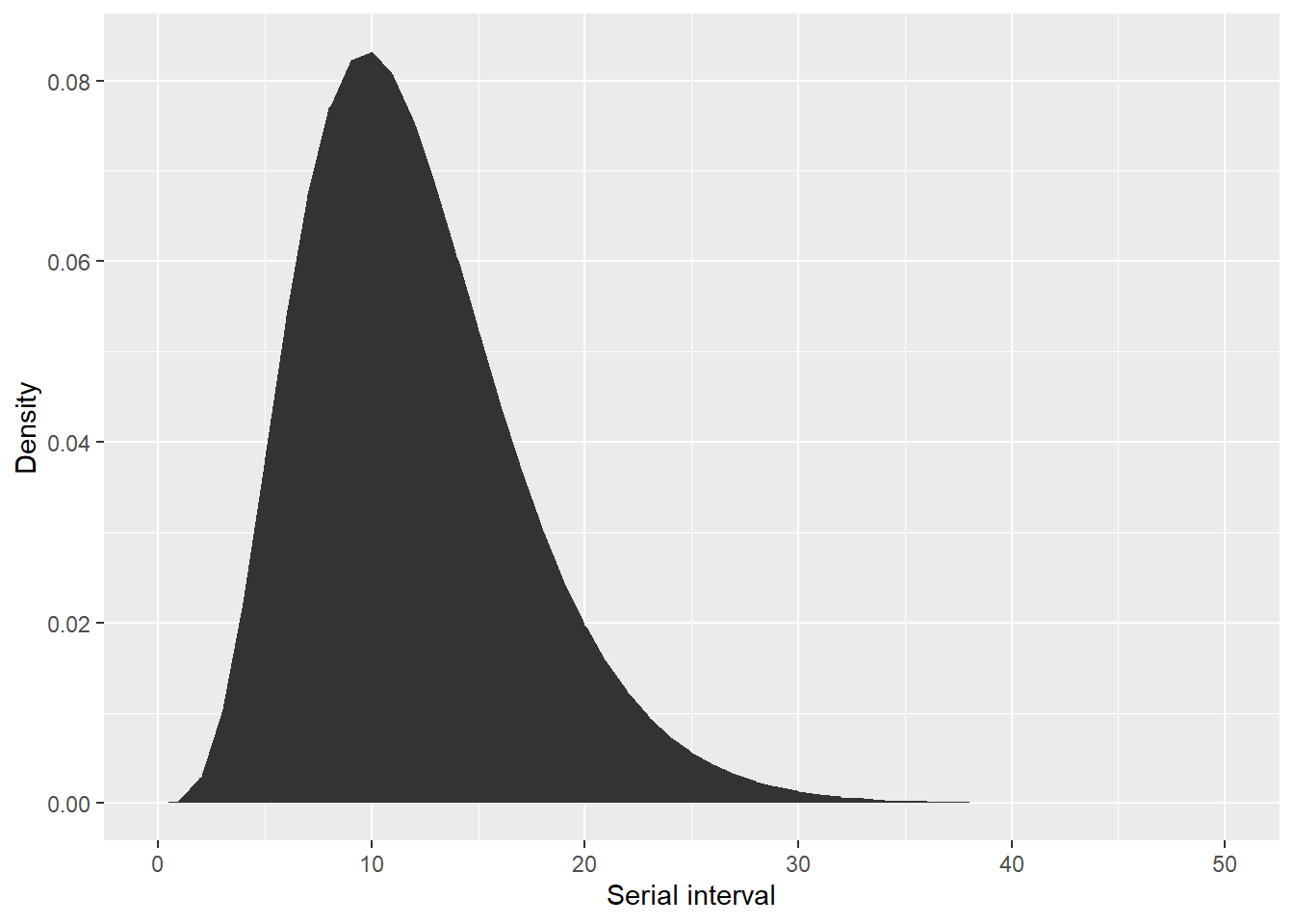
)Вот быстрая проверка, чтобы убедиться, что серийный интервал выглядит верно. Мы смотрим на плотность гамма распределения, которую мы только что задали с помощью $d, что эквивалентно вызову dgamma:
Использование оценок серийного интервала из данных
Поскольку у нас есть данные по датам появления симптомов и цепочкам передачи, мы можем также оценить серийный интервал из построчного списка, рассчитав задержку между датам появления симптомов в парах инфицирующий-инфицируемый. Как мы это делали в разделе EpiNow2, мы вызываем функцию get_pairwise из пакета epicontacts, что позволяет нам рассчитать попарную разницу свойств построчного списка между парами передачи. Сначала мы создаем объект epicontacts (см. страницу [Цепочки передачи] для получения детальной информации):
## генерируем контакты
contacts <- linelist %>%
transmute(
from = infector,
to = case_id
) %>%
drop_na()
## генерируем объект epicontacts
epic <- make_epicontacts(
linelist = linelist,
contacts = contacts,
directed = TRUE
)Затем мы строим разницу в датах появления симптомов между парами передачи, рассчитанную с использованием get_pairwise, по гамма распределению. Мы используем удобную функцию fit_disc_gamma из пакета epitrix для этой процедуры построения, так как нам нужно дискретизированное распределение.
## оцениваем гамма серийный интервал
serial_interval <- fit_disc_gamma(get_pairwise(epic, "date_onset"))
## изучаем оценки
serial_interval[c("mu", "sd")]$mu
[1] 11.51047
$sd
[1] 7.696056Прогнозирование заболеваемости
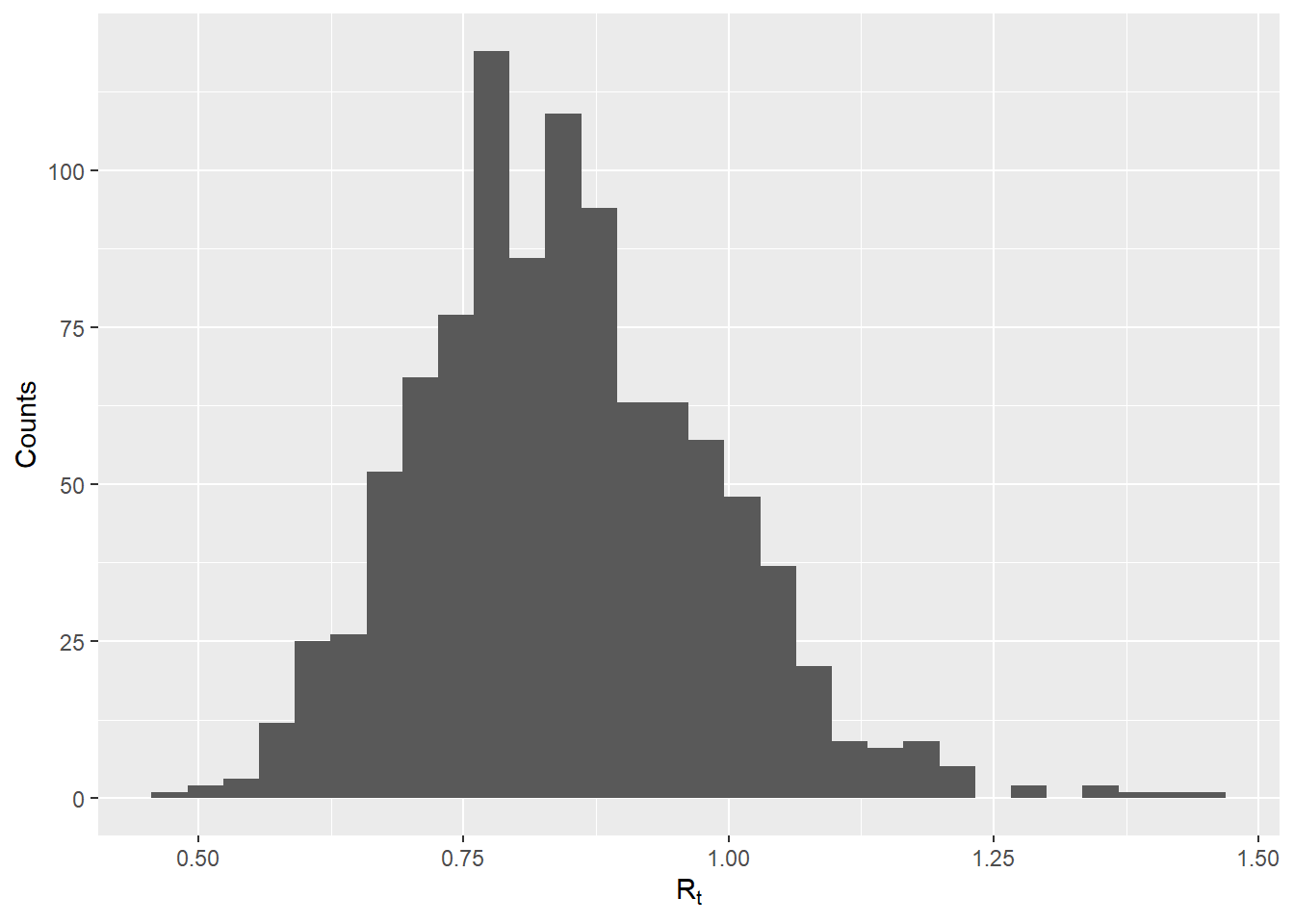
Чтобы прогнозировать будущую заболеваемость, нам нужно задать историческую заболеваемость в форме объекта incidence, а также выборку реалистичных значений Rt. Мы сгенерируем эти значения, используя оценки Rt, сгенерированные EpiEstim в предыдущем разделе (в “Оценке Rt”) и хранящиеся в объекте epiestim_res_emp. В коде ниже мы извлекаем оценки среднего значения и стандартного отклонения для Rt для последнего временного периода вспышки (используя функцию tail для оценки последнего элемента вектора), и делаем моделирование 1000 значений из гамма распределения, используя rgamma. Вы можете также задать собственный вектор значений Rt, которые вы хотите использовать для прогнозов на будущее.
## создаем объект заболеваемости из дат появления симптомов
inc <- incidence::incidence(linelist$date_onset)256 missing observations were removed.## извлекаем реалистичные значения r из наиболее свежей оценки
mean_r <- tail(epiestim_res_emp$R$`Mean(R)`, 1)
sd_r <- tail(epiestim_res_emp$R$`Std(R)`, 1)
shapescale <- gamma_mucv2shapescale(mu = mean_r, cv = sd_r/mean_r)
plausible_r <- rgamma(1000, shape = shapescale$shape, scale = shapescale$scale)
## проверяем распределение
qplot(x = plausible_r, geom = "histogram", xlab = expression(R[t]), ylab = "Counts")`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.Затем используем функцию project(), чтобы сделать сам прогноз. Мы указываем, на сколько дней мы хотим делать прогноз, с помощью аргументов n_days, и задаем число симуляций с помощью аргумента n_sim.
## делаем прогноз
proj <- project(
x = inc,
R = plausible_r,
si = serial_interval$distribution,
n_days = 21,
n_sim = 1000
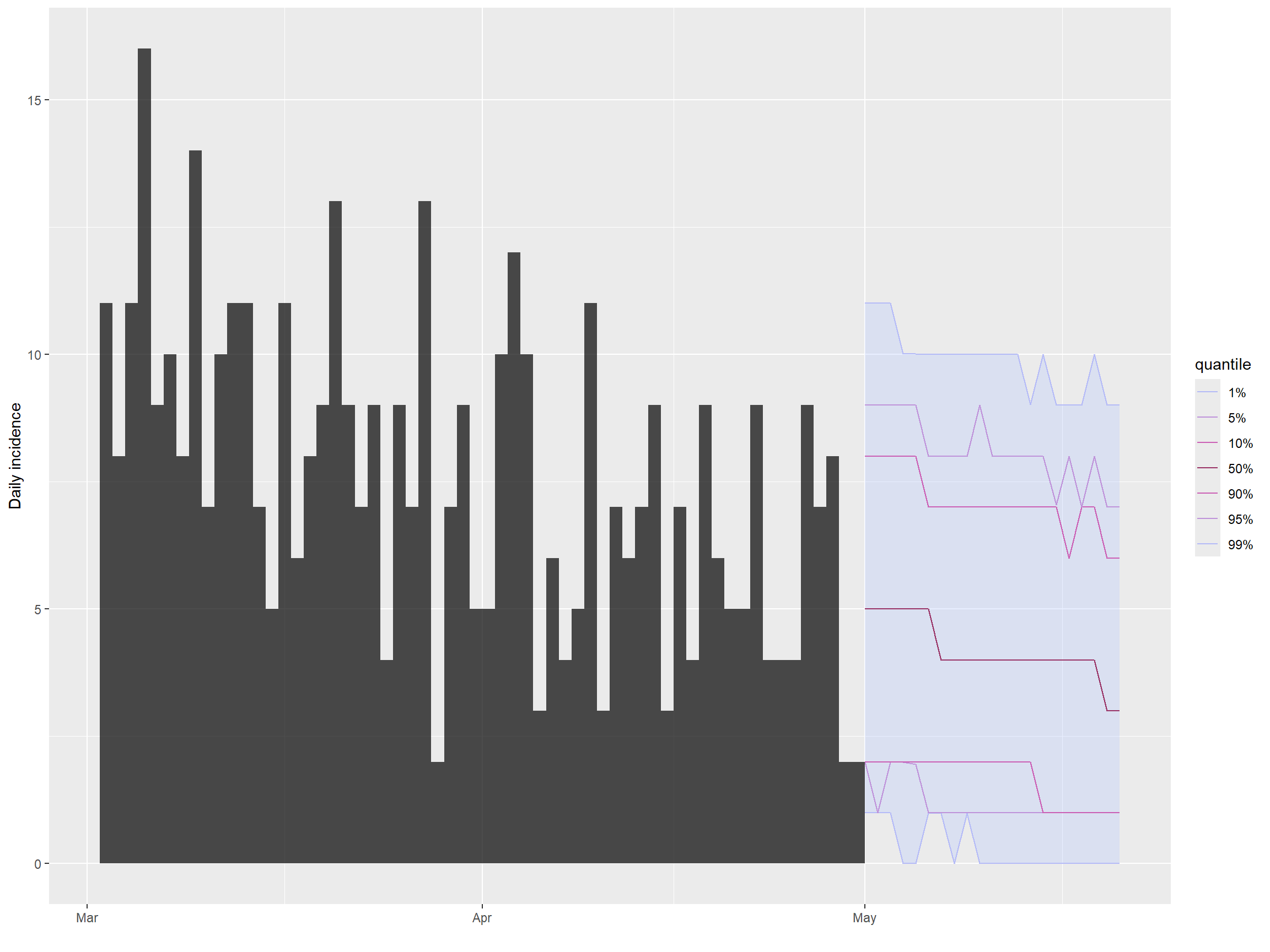
)Затем мы можем построить график заболеваемости и прогнозов, используя функции plot() и add_projections(). Мы можем легко взять подмножество в объекте incidence, чтобы показать только самые последние случаи, с помощью оператора квадратных скобок.
## строим график заболеваемости и прогнозов
plot(inc[inc$dates > as.Date("2015-03-01")]) %>%
add_projections(proj)Можно легко извлечь сырые оценочные данные по ежедневному количеству случаев, конвертировав выходные данные в датафрейм.
## конвертируем в датафрейм для сырых данных
proj_df <- as.data.frame(proj)
proj_df24.5 Ресурсы
- Вот страница, описывающая методологию, реализуемую в EpiEstim.
- Вот страница, описывающая методологию, реализуемую в EpiNow2.
- Вот страница, описывающая различные методологические и практические аспекты для оценок в Rt.